CEFALOSPORINE
1. INTRODUZIONE
Le Cefalosporine rappresentano una classe di antibiotici ampiamente utilizzata in terapia
grazie all’ampio spettro d’azione, alla favorevole azione battericida, al basso indice terapeutico
(in quanto battericide solo sull’organismo infettante) e alla capacità di raggiungere la sede
d’infezione.
Prima fonte della classe delle Cefalosporine fu il Cephalosporium acremonium
(Acremonium chrysogenum) isolato nel 1945 da G.Brotzu a Cagliari. Da questo micete fu
estratta la Cefalosporina C, capostipite naturale della famiglia delle Cefalosporine, che si rivelò
subito interessante per il suo spettro di attività ampio, in quanto attiva sia verso i batteri Gram+
che verso i Gram-; inoltre, rispetto alla penicillina G, presentava il vantaggio di essere resistente
alle β-lattamasi. In realtà, essa non trovò applicazione in sede terapeutica, ma fu di enorme
interesse industriale per produrre l’acido 7-aminocefalosporanico, composto base per
l’ottenimento delle Cefalosporine semisintetiche di uso clinico.[2]
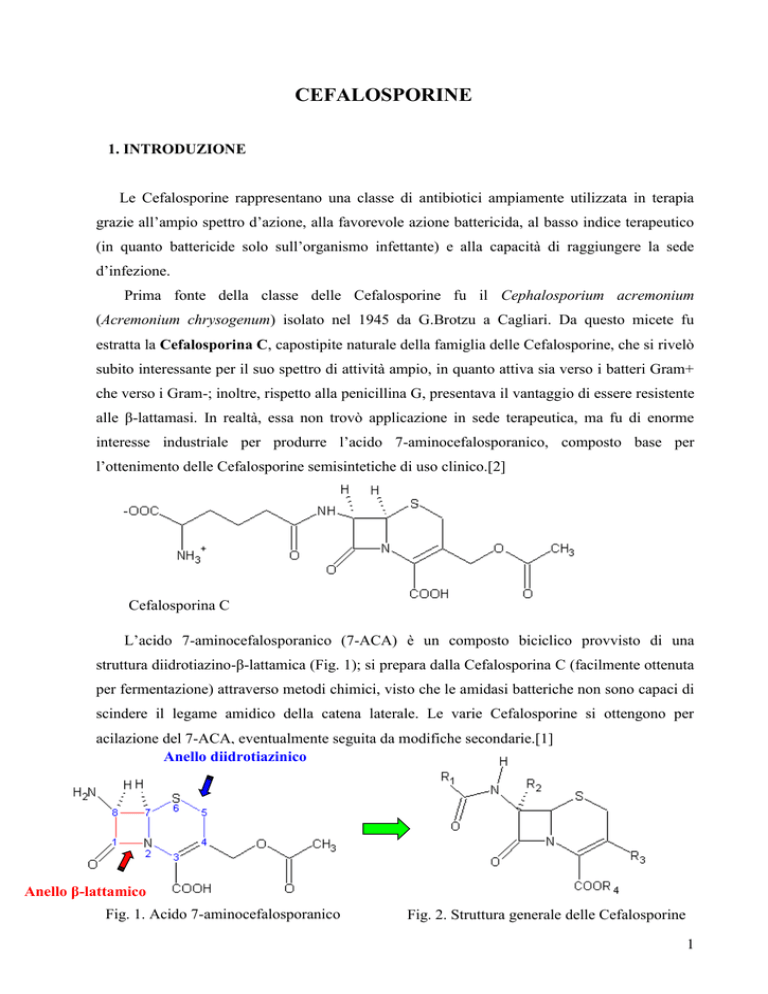
Cefalosporina C
L’acido 7-aminocefalosporanico (7-ACA) è un composto biciclico provvisto di una
struttura diidrotiazino-β-lattamica (Fig. 1); si prepara dalla Cefalosporina C (facilmente ottenuta
per fermentazione) attraverso metodi chimici, visto che le amidasi batteriche non sono capaci di
scindere il legame amidico della catena laterale. Le varie Cefalosporine si ottengono per
acilazione del 7-ACA, eventualmente seguita da modifiche secondarie.[1]
Anello diidrotiazinico
Anello β-lattamico
Fig. 1. Acido 7-aminocefalosporanico
Fig. 2. Struttura generale delle Cefalosporine
1
La realizzazione di questi antibiotici si è susseguita nel tempo, e ciò ha consentito di
classificare le Cefalosporine in farmaci di 1a, 2a, 3a e poi anche di 4a generazione, in base al loro
spettro antibatterico verso i bacilli Gram-.
■ Le Cefalosporine di 1a generazione hanno maggiore affinità per PBP di stafilococchi, sono
le più attive contro Gram+ e hanno uno spettro antibatterico più ristretto per Gram- rispetto a
quelle di 2a generazione.
■ Le cefalosporine di 2a generazione sono più attive contro i Gram- rispetto a quelle di 1a
generazione.
Cefossitina, Cefmetazolo, Cefotetan e Cefurossima sono resistenti alle β-lattamasi di Gram-.
■ Le Cefalosporine di 3a generazione hanno maggiore affinità per PBP di enterobacteri,
maggiore capacità di superare la membrana esterna dei Gram-, sono più attive e hanno uno
spettro antimicrobico ancora più ampio contro Gram-, sono più stabili alle β-lattamasi di Grame sono meno attive di quelle di 1a generazione contro batteri Gram+.
■ Le Cefalosporine di 4a generazione hanno uno spettro d’azione molto ampio, un’alta
affinità per il bersaglio e un’elevata resistenza all’azione idrolitica da parte delle β-lattamasi.
Sono usate per infezioni scarsamente sensibili alle Cefalosporine più vecchie.[1]
La progressiva evoluzione della famiglia delle Cefalosporine ha quindi ampliato lo spettro
antibatterico iniziale che risultava essere limitato ai batteri Gram+, escluso l’enterococco ed a pochi
Gram-, inducendo un ampio spettro d’azione anche sui germi produttori di β-lattamasi e sui germi
“difficili”, quali Pseudomonas, Serratia, Bacteroides.[2]
Nelle tabelle seguenti sono riportate le cefalosporine attualmente usate in terapia. [1]
Cefalosporine di 1a generazione
R1
S
R3
R4
O
CH3
H
Cefalotina
H
Cefazolina
H
Ceftezolo
O
S
S
CH3
N
N
N
N
N
S
N
S
N
N
Continua…
2
S
O
CH 3
H
Cefapirina
H
Cefalessina
CH 3
H
Cefadroxil
CH 3
H
Cefradina
N
O
CH 3
H
NH 2
HO
H
NH 2
H
NH 2
Cefalosporine di 2a generazione
R1
R3
R4
HO
S
NH
N
H
Cefatrizina
H
Cefprozil
H
Cefmandolo
H
Cefurossima
H
Cefaclor
N
H
NH2
O
NH2
O
H
OH
O
O
NH2
O
N
OCH 3
Cl
H
NH2
Continua…
3
COOH
S
N
N
N
H
Ceforanide
H
Cefonicid
N
NH2
SO 3H
S
N
N
H
N
N
OH
Cefamicine di 2a generazione (R2=OCH3)
R1
R3
R4
S
O
NH2
H
Cefossitina
H
Cefotetan
H
Cefmetazolo
O
O
S
CH 3
S
S
N
H 2N
N
N
O
N
OH
CH 3
S
N
S
N
N
N
N
Cefalosporine di 3a generazione
R1
R3
R4
O
CH 3
H
Cefotassima
H
Ceftisossima
H
Ceftriaxone
S
O
H2 N
N
H
N
CH3
OCH 3
S
N
NH
N
CH 2
O
Continua…
4
S
H2N
N
N
COOH
CH3
+
H
Ceftazidima
H
Cefodizima
H
Cefissima
H
Cefoperazone
H
Ceftibuten
H
Cefetamet
H
Cefdinir
N
O
CH3
S
COOH
S
S
H2N
N
N
N
OCH 3
CH3
S
H2N
N
N
O
COOH
HO
CH3
S
N
N
H
NH
O
N
N
O
O
N
N
CH3
S
H2N
H
N
COOH
S
H2N
CH3
N
N
OCH 3
S
H2N
CH2
N
N
OH
Cefalosporine di 4a generazione
R1
S
R3
N
R4
O
H 2N
N
NH 2
N
N
H
Cefclidina
+
O
CH3
Continua…
5
H3 C
N
+
H
Cefepima
H
Cefpirone
H
Cefoselis
S
H2N
N
N
O
N
+
CH3
N
+
N
NH 2
OH
2. MECCANISMO D’AZIONE
2.1. INIBIZIONE TRANPEPTIDAZIONE
Il meccanismo d’azione delle Cefalosporine consiste in un’inibizione della sintesi della parete
cellulare batterica per interferenza sul processo enzimatico di transpeptidazione, che catalizza la
formazione delle reticolazioni polimeriche peptidoglicaniche (Fig. 3).[1-2]
Fig. 3 Struttura del Peptidoglicano.[1]
La biosintesi del peptidoglicano può essere schematizzata in 3 stadi:
Il I° stadio, consiste nella produzione di unità base UDP-acetilglucosamina e UDP-muramilpentapeptide, dentro il citoplasma.
Il II° stadio, procede con la condensazione delle unità base e il loro trasferimento sulla struttura
peptidoglicanica in fase di formazione, a livello della membrana cellulare batterica.
Il III° stadio,
coinvolge la formazione di legami trasversali (cross-linking) fra le catene
peptidoglicaniche neoformate (Fig. 4).
6
Fig. 4 Cross-linking.[1]
Quest’ultimo stadio, che si compie al di fuori della membrana citoplasmatica, è catalizzato da una
transpeptidasi, enzima che usa un ossidrile serinico per attaccare la penultima unità di alanina
formando un legame covalente, mentre l’alanina terminale è allontanata. Il complesso enzimapeptidoglicano è attaccato dal gruppo amminico terminale dell’unità pentaglicinica adiacente, che
rigenera il sito attivo della transpeptidasi rendendolo disponibile per l’ulteriore azione catalitica e
produce un nuovo legame ammidico che lega fra loro le due catene adiacenti (Fig. 5).[2]
Fig. 5 Meccanismo catalitico delle Transpeptidasi batteriche.[1]
Continua…
7
Gli antibiotici β-lattamici funzionano da inibitori irreversibili delle transpeptidasi batteriche.
Modelli stereochimici dimostrano che la loro struttura mima il frammento Acetil-D-Ala-D-Ala del
substrato (Fig. 6).[1]
Acido 7-acetilaminocefalosporanico
Acetil D-Ala-D-Ala-OH
Fig. 6 Modelli stereochimici dell’Acido 7-acetilaminocefalosporanico e dell’Acetil-DAla-D-Ala.[1]
Dalle immagini stereochimiche riportate vediamo che i gruppi funzionali importanti per il
binding con la transpeptidasi sono:
1.
Gruppo carbonilico β-lattamico corrispondente al CO-NH interno di D-Ala-D-Ala.
2.
Gruppo carbossilato, per l’interazione con una carica positiva della transpeptidasi.
3.
Gruppi acetossimetilenici della Cefalosporina (metile D-Ala terrminale).
4.
Catena acilaminica in 7 della cefalosporina (R-CO della D-Ala interna).[1]
Gli antibiotici β-lattamici vengono quindi riconosciuti come se fossero il substrato e formano un
addotto reversibile. All’interno dell’addotto reversibile operano un’efficace acilazione della serina
catalitica, con apertura dell’anello β-lattamico (Fig. 7).[1]
8
L’anello β-lattamico in forte tensione è molto più reattivo di un’ammide normale, specialmente se
fuso con un opportuno eterociclo per dare un sistema biciclico. La perdita della tensione dell’anello
che si ottiene è così pronunciata che la reazione non può procedere in senso inverso. Il residuo
eterociclico, legato covalentemente all’enzima, costituisce un ostacolo all’avvicinamento da parte
dell’unità pentaglicinica, in questo modo il sito attivo non è rigenerato e non si forma il legame
crociato. Ne risulta una parete difettosa, con enzima inattivato. [2]
Fig 7 Acilazione
dellatranspeptidasi.[1]
2.2. RELAZIONI STRUTTURA-ATTIVITA’ DELLE CEFALOSPORINE
Le relazioni struttura - attività (SAR) effettuate sulle Cefalosporine hanno chiarito i seguenti punti:
1. L’anello β-lattamico è essenziale per l’attività antimicrobica delle cefalosporine.
2. I gruppi R1, R2, R3 e R4 influiscono sulle proprietà farmacodinamiche e farmacocinetiche
delle cefalosporine (Fig. 2).
R1: sostituenti in C 7 corrispondenti alla posizione R1 (legame amidico) sono responsabili della
suscettibilità all’ambiente acido, delle proprietà farmacocinetiche, delle proprietà antibatteriche e
della resistenza alle β-lattamasi. La catena formata da un anello 2-aminotiazolico e da un gruppo di
O-alchil-ossima, tipico delle cefalosporine di 3a generazione, conferisce, infatti, a tali antibiotici una
maggiore affinità per le transpeptidasi delle enterobacteriacee, una maggiore capacità di superare la
membrana esterna dei Gram- e una maggiore stabilità alle β-lattamasi dei Gram-.[1]
R2: in C7, la sostituzione α-metossi, sul nucleo β-lattamico delle Cefamicine, aumenta la
resistenza all’idrolisi da parte delle β-lattamasi e l’attività nei confronti degli anaerobi. Tale
sostituzione determina però anche una diminuzione dell’attività antistafilococcica.[2]
9
R3: in C3 il gruppo R3 influenza principalmente il metabolismo e le proprietà farmacocinetiche
delle cefalosporine, quali la durata di azione e la potenza. Modifiche in questa posizione
interesseranno anche lo spettro, infatti, l'aggiunta di atomi elettronegativi in R3 migliora la capacità
di attacco e di legame delle cefalosporine alle PBP.
R4: il gruppo carbossilico deve essere libero, infatti, sotto forma di carbossilato stabilisce un
legame ionico essenziale per il binding con la transpeptidasi. La funzione acida si può esterificare
dando origine a dei profarmaci che modificano temporaneamente le proprietà farmacocinetiche
delle cefalosporine (incremento della lipofilia e dell’assorbimento gastroenterico), in vivo R4 è
rimosso per ripristinare le proprietà antimicrobiche.[1]
Tutte le sostituzioni esercitano influenze diverse e l’inserimento di diversi gruppi R1 e R3, ha
portato alla formazione di composti dotati di buon’attività e bassa tossicità. Al tempo stesso, alcuni
sostituenti sono responsabili, in alcun individui, di intolleranza o di difetti dell’emostasi.
Essendo ancora poche le informazioni riguardanti la struttura topografica recettoriale, essendo
essa stessa variabile e specie-specifica, i metodi di “drug design” sono basati principalmente
sull’aspetto farmacoforico (“pharmacophore-based”), approccio classico in cui si modificano
strutture preesistenti, già attive, al fine di trovare la conformazione bioattiva, quella responsabile
dell’effetto biologico.
Tutti gli sforzi attuali sono diretti ad ottenere delle cefalosporine che abbiano una buon’attività
su cocchi Gram+, enterobatteri, Pseudomonas, Acinetobacter, anaerobi. [2]
2.3. I TARGETS MOLECOLARI: PBPs
I targets per le droghe β-lattamiche sono le Penicillin Binding Proteins PBPs. I batteri hanno
diverse PBPs individuali, ognuna con una funzione separata, che convenzionalmente vengono
numerate in base alla dimensione, con PBP-1 come la proteina più grande. Le PBP fanno parte della
famiglia delle proteine serin-trasferasi, come gruppo di proteine specializzate nell’assemblaggio e
metabolismo della parete cellulare batterica. Questa famiglia può catalizzare il trasferimento di un
gruppo R1-CO di un estere, tioestere o ammide carbonil donatore R1-CO-X-R2 ad un accettore, HY,
attraverso la formazione di un legame intermedio serin-estere acil (R1-CO-). Le PBP possono essere
classificate come lmw (basso peso molecolare) o come hmw (alto peso molecolare). Le hmw
possono ancora essere suddivise in quelle di classe A e classe B. Questi due gruppi combinano in
una singola catena polipeptidica i domini di transglicolazione e il dominio transpeptidasico.
Troviamo inoltre un segnale che non viene tagliato (verde in fig. 8), la cui funzione è quella di
ancorare il peptide alla membrana plasmatica. Questo dominio si trova fuso alla porzione N-ter del
10
modulo di transglicosilazione (arancione in fig. 8), che è ancora fuso al modulo di acyl-serin
transferasi (blu in fig. 8). In contrasto alle hmw, che sono enzimi mutlifunzionali, ed essenziali per
la sopravvivenza del batterio stesso, le lmw sono proteine monofunzionali e non essenziali per la
cellula batterica. [3-4]
Fig. 8 Struttura della PBP2a* del
batterio Stafilococcus aureus.[6]
La struttura tridimensionale (Fig. 9,10,11) di molte PBP evidenzia α-eliche che racchiudono
un core formato da 5 β-sheet antiparalleli, supportati da due eliche da un lato e una singola elica
dall’altro con un dominio α-elica centrale. (Fig. 12,13,14,15).[4]
Fig. 9 Struttura di una forma solubile di Fig. 10 Struttura di una forma solubile di
PBP1b di Streptococcus pneumoniae.[3] PBP1b di Streptococcus pneumoniae+
nitrocefin.[3]
11
Fig. 11 Struttura di una forma solubile di
PBP1b di Streptococcus pneumoniae+
ceftisossima.[3]
Fig. 13[4]
Fig. 12[4]
Fig. 14[4]
Fig. 15[4]
Fondamentalmente troviamo tre motivi dominanti in tutte le PBPs, che costituiscono i siti
attivi di queste proteine (Fig. 16). Il primo è dato da una sequenza amminoacidica costituita da
SXXK N-ter della catena 2α, ed assume una posizione centrale nel sito attivo. Nel sito attivo
mostrato in figura, relativo all’enzima R6 PBP2x di streptococcus pneumoniae, la serina e la lisina
assumono la posizione 337/340. Il secondo dominio S/YxN/C è un loop che connette due eliche
dell’α dominio (Ser 452 e Asn 464 in figura) e definisce un lato del dominio catalitico. L’ultimo
motivo è dato da TGS che si trova nel 3β sheet e definisce l’altro lato del centro catalitico.[6-4]
Fig. 16 Sito attivo nella
struttura Nitrocefin-acilSauPBP2a. Il Nitrocefin
è mostrato in viola. [6]
12
La caratterizzazione del sito attivo permette di individuare i siti di interazione e di legame
tra enzima e inibitore e questo potrebbe rappresentare un primo passo verso la sintesi di nuove
molecole altamente specifiche e selettive perché complementari al proprio bersaglio.
3. RESISTENZA BATTERICA
Un altro aspetto importante per questa classe di antibiotici è la resistenza. Quando un antibiotico
è ampiamente usato si sviluppano inevitabilmente sistemi batterici resistenti. I batteri possono
sviluppare resistenza ai β-lattamici mediante tre meccanismi:
1.
prevenzione dell'interazione fra antibiotico e PBP bersaglio,
2.
impossibilità dell'antibiotico di legarsi alla PBP e
3.
idrolisi dell'antibiotico da parte delle β-lattamasi.
1. Il primo meccanismo di resistenza è stato visto solo nei batteri gram-negativi (in particolare in
Pseudomonas species), poiché essi hanno una membrana esterna che copre lo strato di
peptidoglicano. La penetrazione di antibiotici β-lattamici nei gram-negativi richiede il passaggio
attraverso i pori della membrana esterna. Modificazioni delle proteine (porine) che formano le
pareti dei pori possono alterare le dimensioni e la carica di questi canali e provocare l'esclusione
dell'antibiotico (Fig. 17 e 18).[7-8]
Fig. 17 Struttura della parete
cellulare dei Gram+. [8]
Fig. 18 Struttura della parete
cellulare dei Gram-. [8]
2. I batteri possono anche acquisire una PBP modificata che è incapace di legarsi agli antibiotici βlattamici, ma che contribuisce alla sintesi del peptidoglicano. La PBP modificata può aver origine
da una mutazione del gene della PBP o dall'acquisizione di una nuova PBP.[7]
Un esempio è la Meticillina-resistenza in MRSA, dovuta all’acquisizione del gene mecA
mediante trasferimento da una specie indefinita. Il gene mecA è altamente conservato è codifica per
PBP2a, una nuova PBP differente dalle PBPs normalmente prodotte dallo S.aureus. PBP2a è
altamente resistente a tutti gli antibiotici β-lattamici clinicamente usati e rimane attiva per sostenere
13
la sintesi della parete cellulare a concentrazioni normalmente letali di β-lattamici. Vari studi cinetici
hanno dimostrato che la resistenza di PBPs2a è principalmente dovuta all’inefficiente formazione
dell’intermedio covalente acil-PBP(cioè ad una bassa velocità di acilazione k2) e non è causata dal
ridotto accesso del β-lattamico nel sito attivo, visto che i valori delle kd sono relativamente bassi, né
da una più rapida rottura dell’intermedio acil-PBP.
Fig. 19 Sito attivo
nell’Apo-struttura di
SauPBP2a. E’ mostrato il
Nitrocefin, con la stessa
conformazione che
presenta nella struttura
precedente, per notare
meglio la distorsione del
sito attivo.[6]
In seguito, il confronto tra due strutture cristallografiche, quella dell’apo-enzima SauPBP2a (enzima
non legato all’inibitore, fig.19) e quella della SauPBP2a legata al Nitrocefin (un tipo di
cefalosporina) (fig.16), ha rivelato che la scarsa velocità d’acilazione è dovuta ad un distorto sito
attivo che deve subire cambiamenti conformazionali al foglietto β3 e alla porzione N-terminale
dell’elica α2 affinché avvenga l’acilazione. Nell’apo-struttura, infatti, la serina403 è in una cattiva
posizione per l’attacco nucleofilo e c’è un ingombro sterico tra il carbossilato del Nitrocefin e il Cα
della gly599.
Fig. 20 Sovrapposizione delle strutture
Apo-(in blu) e Nitrocefin-acil-SauPBP2a
(in giallo).Il Nitrocefin è mostrato in
viola. [6]
La sovrapposizione delle due strutture (Fig. 20) rivela, inoltre, che
la torsione del foglietto β3 è necessaria per accomodare l’elica α2 nella giusta conformazione in
quanto si può vedere un ingombro sterico tra il carbonile della ser598 nel complesso acil-PBP e il
Cβ della serina403 nell’apo-struttura.
Tutti questi cambiamenti conformazionali, energeticamente sfavoriti, impediscono la formazione
dell’intermedio acil-PBP che si traduce in una ridotta velocità d’acilazione. I valori di kd bassi
indicano invece che il legame iniziale dei β-lattamici in SauPBP2a non è molto impedito e questo
suggerisce che la posizione del Nitrocefin nel complesso iniziale differirà da quell’osservata nella
struttura acil-PBP in modo che il foglietto β non debba distorcersi durante il legame iniziale. Tutto
ciò trova conferma nell’assenza d’equivalenti differenze conformazionali tra le strutture apo- e acilPBP per R61 PBP e R6 PBP2x sensibili ai β-lattamici (Fig. 21), che concorda con una transizione
14
più rapida dal complesso iniziale all’intermedio acil-PBP, come rivelano le loro alte velocità
d’acilazione (k2).
Fig. 21 Sovrapposizione delle strutture apo-enzima
(gialla), cioè non legato al β-lattamico, e complesso
Acil-PBP (viola) di R6 PBP2x di S.Pneumoniae in
corrispondenza della regione del sito attivo.[6]
Si può, quindi, affermare che, essendo l’acilazione uno step chiave nell’inibizione delle PBPs da
parte dei β-lattamici, la proposta che il sito attivo effettivamente bilancia una ritenzione dell’attività
transpeptidasica essenziale (attraverso la conservazione dei residui chiave catalitici) con una
riduzione dell’affinità per i β-lattamici (attraverso la distorsione del sito attivo per ridurre l’efficacia
catalitica) è probabilmente applicabile a PBP2a e alle altre PBPs resistenti in generale. Si è visto,
infatti, che mutazioni nella regione N-terminale dell’elica α2 in R6 PBP2x influenzano
significativamente la velocità d’acilazione in quanto la conformazione della porzione N-terminale
dell’elica α2 gioca un ruolo fondamentale nel collocare correttamente la catena laterale della
serina337 per l’attacco nucleofilo (fig.21).
Dato che la scarsa efficienza d’acilazione è una caratteristica intrinseca della SauPBP2a, un
importante aspetto per il disegno d’inibitori sarà quello di migliorare l’affinità di legame
incrementando il numero d’interazioni non covalenti tra inibitore e sito attivo, in modo da
aumentare l’efficacia complessiva di acilazione e ridurre, quindi, la resistenza.
Le strutture di Nitrocefin-acil-, penicillina G-acil- e meticillin-acil-PBP (Fig. 22 A,B,C)
identificano le caratteristiche responsabili delle differenti affinità di SauPBP2a per questi composti
β-lattamici, che determinano una diversa resistenza. In A si osservano una serie di legami idrogeno
che ritroviamo anche in B; in B sono assenti le interazioni di Van der Waals prodotte dal
sostituente R3 del Nitrocefin., che determinano un aumento dell’affinità di legame (kd bassa)e,
quindi, una più elevata velocità d’acilazione del Nitrocefin rispetto a quella della penicillina G. La
meticillina presenta una velocità d’acilazione ancora più bassa a causa dei limitati legami
all’interno del sito attivo. La figura C mostra, infatti, la meticillina in una posizione che permette
al voluminoso dimetossifenil R1 sostituente di accomodarsi nella regione occupata dal sostituente
R1 del Nitrocefin; ma, questo incrementa la distanza tra l’O terminale della ser462 e l’N dell’anello
β-lattamico rispetto a quanto osservato nel complesso del Nitrocefin, ritardando il trasferimento del
protone dalla ser462 all’N durante l’acilazione. Cosi l’acilazione intrinsecamente lenta di SauPBP2a
15
lavora in associazione con i limitati legami all’interno del ristretto sito attivo (Fig. 22 C) per
produrre una accresciuta resistenza contro β-lattamici ingombranti, come la meticillina.
A
B
C
Fig. 22 A,B,C A. Sito attivo nella struttura Nitrocefin-acil-SauPBP2a. B. Sito attivo nella struttura
Penicillina G-acil-SauPBP2a. C sito attivo nella struttura Meticillin-acil-SauPBP2a. Le tre immagini
mettono in evidenza le interazioni che si instaurano nel sito attivo di queste tre strutture.[6]
Nel disegnare inibitori β-lattamici più efficaci è prevedibile che la riduzione della kd gioca
un grande ruolo nel miglioramento dell’efficacia complessiva dell’acilazione(k2/kd). Coerente con
questo, l’alta affinità di parecchie Cefalosporine sviluppate di recente per SauPBP2a è correlata con
i loro più lunghi sostituenti, che in base alle strutture viste, conferiscono una forma più
complementare al sito attivo e permettono un più largo numero d’interazioni non covalenti
stabilizzanti.[6]
3. Il terzo meccanismo di resistenza batterica consiste nella produzione di β-lattamasi che
inattivano i β-lattamici. Sono state descritte più di 200 diverse beta lattamasi. Alcune sono
specifiche per le penicilline (penicillinasi) e le Cefalosporine (Cefalosporinasi), mentre altre hanno
un ampio spettro d’attività e sono capaci di inattivare la maggior parte degli antibiotici β-lattamici.
Questo ultimo gruppo di β-lattamasi (β-lattamasi a spettro esteso [ESBL]) è particolarmente
problematico, poiché è comunemente codificato da plasmidi e può essere trasferito da un organismo
all'altro. Ciò ha limitato l'uso dei β-lattamici in alcuni ospedali.[7]
Le β-lattamasi a serina derivano per evoluzione dalle PBP batteriche. Esse hanno una serina nella
tasca del sito attivo simile alla serina catalitica delle PBP e presentano un meccanismo catalitico
analogo per la fase d’acilazione; tuttavia, mentre l’acilenzima delle PBP batteriche sensibili è
stabile, l’analogo acilenzima delle β-lattamasi si autoidrolizza velocemente, rigenerando la βlattamasi attiva.[1]
Altre β-lattamasi fanno la stessa cosa, ma usano uno ione zinco anziché un amminoacido della
serina per inattivare le molecole β-lattamiche. In ogni caso, il risultato è che l’agente β-lattamico è
16
rapidamente idrolizzato annullando la sua attività.[1] In generale i batteri Gram+ producono una
gran quantità di β-lattamasi che è secreta a livello extracellulare. Molti di questi enzimi sono
penicillinasi e la loro sintesi è indotta dalla presenza dello stesso antibiotico. Nei batteri Gram- le βlattamasi si trovano in quantità relativamente piccole, ma sono localizzate nello spazio
periplasmatico tra la membrana cellulare esterna ed interna, dove assicurano la massima protezione
al microbo. Nei Gram- le β-lattamasi sono costitutive, cioè sono normalmente prodotte, e
inattivano sia le penicilline che le Cefalosporine.[1-2]
Le Cefalosporine, come abbiamo già detto, si distinguono in generazioni anche sulla base della loro
resistenza alle β-lattamasi. In particolare i composti di 3a generazione mostrano una certa resistenza
all’idrolisi da parte di tali enzimi perchè, oltre ad essere inibitori delle PBP, sono anche inibitori
delle β-lattamasi. Cefalosporine contenenti, nella catena alchilaminica in 7, sostituenti ingombranti
e rigidi oppure il gruppo metossi in 7α inducono nell’acilenzima una conformazione non produttiva
per la fase d’autoidrolisi, orientando il C=O estere in una direzione sfavorevole per l’attacco
dell’acqua.[1]
Cefalosporine contenenti un gruppo uscente nella catena in 3 orientano il C=O estere in una
direzione sfavorevole per l’attacco dell’acqua.[1]
17
4.
CONCLUSIONI
Le Cefalosporine hanno rappresentato e rappresentano uno dei pilastri della terapia
antibatterica, soprattutto quelle di 3a e 4a generazione. Tuttavia anche le Cefalosporine di 1a e 2a
generazione sono attualmente usate nella pratica clinica. Oggi è importante indirizzare le ricerche
farmacologiche e le nuove conoscenze e tecniche per ottenere Cefalosporine che abbiano migliori
caratteristiche farmacocinetiche, ma anche migliore spettro antibatterico.
Tali ricerche sono dirette ad ottenere:
Sintesi di Cefalosporine ancora più stabili alle β-lattamasi recenti.
Ottenere delle Cefalosporine anti-Pseudomonas ed anti-Acinetobacter più attive di quelle
attuali.
Ottenere Cefalosporine con attività rinforzata anti-anaerobi, attive anche su Clostridium
Difficile.
Ottenere delle Cefalosporine ad attività rinforzata sui cocchi Gram+ ed in particolare sugli
Stafilococchi meti-R.
Dott.ssa Gelsomina Ruffolo
I Anno Scuola di Specializzazione in
- Farmacia Ospedaliera Università degli studi di Catanzaro
[A. A. 2004/05]
18
BIBLIOGRAFIA
1. Prof. Carlo Gallina. Appunti delle lezioni di “Chimica Farmaceutica 2”.
Università G. D’annunzio di Chieti e Pescara. Corso di Laurea in Farmacia. AA
2004-2005.
2. http://194.119.197.4/~alcaro/f1/aa_02_03/tesine/cefalosporine.doc.
3. Macheboeuf P, Di Guilmi AM, Job V, Vernet T, Dideberg O, Dessen A. Active
site restructuring regulates ligand recognition in class A penicillin-binding
proteins. Proc Natl Acad Sci U S A. 2005 Jan 18;102(3):577-82. Epub 2005 Jan 6.
4. Ludovico Buti, Giusi Manfredi, Giovanni Passiatore, Alfredo Pezzicoli .
Modeling strutturale di una PBP di Staphylococcus Aureus
5. Michele La Placa. Principi di Microbiologia Medica. 2000
6. Daniel Lim & Natalie C.J. Strynadka. Structural basis for the lactam
resistance of PBP2a from methicillin-resistant Staphylococcus aureus Nature
Structural Biology 9, 870 - 876 (2002) Published online: 21 October 2002.
7. www.microbiologia.unige.it/Pdf/Antibiotici2004.pdf
8. www.med.unifi.it
9. McDonough MA, Anderson JW, Silvaggi NR, Pratt RF, Knox JR, Kelly JA.
Structures of two kinetic intermediates reveal species specificity of penicillinbinding proteins. J Mol Biol. 2002 Sep 6;322(1):111-22.
10. Silvaggi NR, Josephine HR, Kuzin AP, Nagarajan R, Pratt RF, Kelly JA.
Crystal structures of complexes between the R61 DD-peptidase and
peptidoglycan-mimetic beta-lactams: a non-covalent complex with a "perfect
penicillin". J Mol Biol. 2005 Jan 21;345(3):521-33.
19