RFLP
I primi marcatori molecolari ad essere studiati furono gli
RFLP (Restriction Fragment Lenght Polymorphism):particolari
tratti di DNA presenti nella popolazione e trasmessi in
modo ereditario
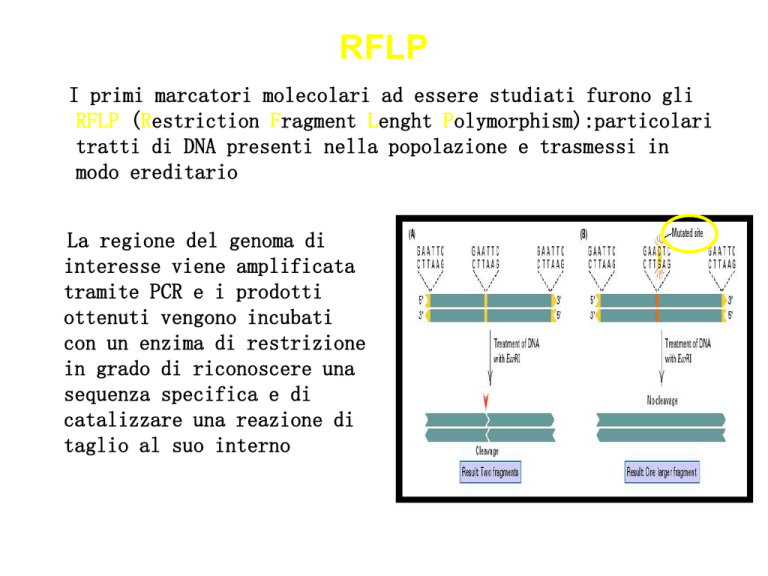
La regione del genoma di
interesse viene amplificata
tramite PCR e i prodotti
ottenuti vengono incubati
con un enzima di restrizione
in grado di riconoscere una
sequenza specifica e di
catalizzare una reazione di
taglio al suo interno
RFLP: Gel di Elettroforesi
Mediante elettroforesi su gel di agarosio si è in grado di
determinare se il frammento amplificato è stato tagliato o
meno, cioè se la sequenza specifica riconosciuta
dall’enzima è presente inalterata oppure no
Direzione
di Migratione
I frammenti
più grandi
migrano più
lentamente
rispetto a
quelli più
corti
The human genome
100%
Extragenic DNA
Gene/gene related sequences
30%
70%
Unique
Coding regions
3%
55%
Repetitive
Introns,promoter,
Pseudogenes,gene fragments
15%
27%
Tandemly
repeated
Satellite
Intersperd
repetitive
SINEs
(<500bp)
Minisatellite
Telomeric
Microsatellite
LINEs
(>500bp)
Hypervariable
Sequenzial breakdown of the genome into component DNA types (Bennett P: Demystified...Microsatellites. J
Clin Pathol:Mol Pathol 2000,53:177-183)
DNA ripetitivo intersperso
• Le singole unità ripetute non sono raggruppate ma sparse in
più punti del genoma. Gli esempi più comuni sono le sequenze
SINEs (Short Interspersed Nuclear Element) e LINEs (Long
Interspersed Nuclear Element)
• Sono associate
perché tendono
povere di geni
mutazionale al
LINEs
prevalentemente a DNA genomico ricco in A/T
a posizionarsi in regioni del cromosoma
allo scopo di imporre il minimo impatto
genoma.
SINEs
• Sono associate prevalentemente a DNA genomico ricco in G/C.
Perché? Sembra che tali sequenze svolgano una qualche
funzione positiva per il genoma: esse sarebbero espresse in
condizioni di stress ed i risultanti RNA legherebbero una
particolare protein chinasi PKR e bloccherebbero la sua
capacità di inattivare la traduzione.
DNA ripetuto in tandem
DNA costituto da blocchi di
sequenze che possono trovarsi in
poche o in molte localizzazioni
cromosomiche differenti. A
seconda delle dimensioni medie
del blocco di unità ripetute è
possibile suddividere questo DNA
non codificante in subclassi:
satellite,minisatellite e
microsatellite.
DNA
satellite
DNA satellite comprende un
grande gruppo di blocchi di
DNA ripetuto in tandem con
singole unità ripetute più o
meno complesse. Il DNA di
questo tipo non è trascritto e
praticamente costituisce il
grosso dell’eterocromatina
del genoma localizzata al
centromero (eterocromatina
pericentrica)
DNA minisatellite comprende
due tipi di sequenze di DNA
ripetuto in tandem, non
trascritto, di modeste
dimensioni e disperso nel
genoma nucleare:
minisatelliti telomerici e
minisatelliti ipervariabili
DNA TELOMERICO
• Questo tipo di DNA è localizzato all’estremità dei cromosomi,
nei telomeri
• Il costituente principale del DNA telomerico è rappresentato da
10-15kb di unità esanucleotidiche ripetute in tandem, in
particolare TTAGGG, che vengono aggiunte da un enzima
specializzato, la telomerasi
• Tali ripetizioni sono responsabili della funzione dei telomeri
in quanto agiscono come protezioni delle estremità dei
cromosomi dalla degradazione e dalle perdite di materiale
• Inoltre forniscono un meccanismo per la replicazione delle
estremità lineari del DNA cromosomico
DNA minisatellite ipervariabile o VNTR
(Variable Number of Tandem Repeat)
• Sono sequenze altamente polimorfiche ed organizzate in oltre
1000 gruppi (lunghi da 0.1 a 20 kb) di corte unità ripetute in
tandem, che variano considerevolmente per dimensioni ma
posseggono una sequenza comune centrale ( core),GGGCAGGAXG
• Molti di questi gruppi si trovano vicino ai telomeri
• La maggior parte di queste sequenze non sono trascritte eccetto
alcuni elementi all’interno di sequenze intrageniche non
codificanti
• Il significato non è ancora chiaro, ma indipendentemente dalla
loro reale funzione nel genoma umano, esiste un utilizzo
pratico di questi gruppi, ma in genere delle sequenze ripetute,
nel DNA fingerprinting (impronta digitale del DNA)


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)








