Prospettive in Pediatria
Aprile-Giugno 2016 • Vol. 46 • N. 182 • Pp. 161-168
Frontiere
Sandro Banfi1 2
Brunella Franco2 3
Genetica Medica, Dipartimento
di Biochimica, Biofisica e
Patologia Generale, Seconda
Università degli Studi di Napoli;
2 Telethon Institute of Genetics
and Medicine (TIGEM), Pozzuoli
(NA); 3 Genetica Medica,
Dipartimento di Scienze Mediche
Traslazionali, Università Federico
II, Napoli
1 Genome Editing
nella medicina del futuro:
opportunità e rischi
Il Genome Editing (GE) è una tecnica di biologia molecolare che permette di modificare in
modo permanente il DNA di una cellula creando sostituzioni, inserzioni o delezioni. Esso
richiede il taglio della doppia elica del DNA in punti specifici del genoma a opera di proteine
chiamate nucleasi (una sorta di “forbici molecolari”). Il taglio è seguito da un processo naturale di riparazione del DNA che può portare o alla inattivazione del gene bersaglio oppure alla
correzione di mutazioni specifiche, a seconda della metodica utilizzata. A causa della sua
complessità sperimentale, il GE è stato poco utilizzato in campo biomedico fino alla recentissima scoperta del sistema CRISPR/CAS9, avvenuta pochi anni fa. Tramite questo sistema,
alla portata anche di laboratori piccoli e relativamente poco attrezzati, è possibile effettuare
studi della funzione genica sia in modelli cellulari che animali. Inoltre, diventa più semplice
disegnare approcci di terapia genica per malattie umane (genetiche e non genetiche). Il
sistema CRISPR/CAS9 sta già rivoluzionando la ricerca biomedica di base e presenta un
enorme potenziale di applicabilità nella pratica clinica in un futuro relativamente prossimo.
Tuttavia, prima che ciò si realizzi, restano da risolvere ancora delle criticità di natura tecnica e
va aperto un serio confronto nella comunità scientifica riguardo alcune implicazioni di tipo etico che possono scaturire da un utilizzo totalmente incontrollato di questa nuova procedura.
Riassunto
Genome editing (GE) is a molecular procedure used to modify the genomic DNA of a cell
by creating substitutions, insertions or deletions. It requires cutting double strand DNA at
desired genomic locations by proteins called nucleases (sort of “molecular scissors”). The
break is followed by a natural process of DNA repair that can either bring about the inactivation of the target gene or correct specific mutations, depending on the method used. Because of its experimental complexity, GE has had a relatively low impact in the biomedical
field until the recent discovery of the CRISPR/CAS9 system just a few years ago. Through
this system, which can be easily adopted even by small and not highly equipped laboratories, it is becoming straightforward to carry out gene functional studies in either cellular or
animal models. Moreover, this method allows the design of, in principle, more effective gene
therapy approaches for human diseases (both genetic and non-genetic). The CRISPR/
CAS9 system is already revolutionising basic biomedical research and has a huge potential
for clinical applications in the near future. However, there are still a number of technical problems that must be solved to allow safe and reliable introduction of CRISPR-CAS9-based
GE procedures in clinical practice. Finally, it will also be necessary to start open discussions
about the ethical implications that may arise from uncontrolled use of this new procedure.
Summary
Metodologia della ricerca
bibliografica effettuata
La ricerca degli articoli rilevanti è stata effettuata tramite la banca bibliografica PubMed (http://www.ncbi.
nlm.nih.gov/pubmed), utilizzando come parole chiave:
“genome editing”, “CRISPR CAS9”, “TALEN”. Sono stati
inclusi solo articoli in lingua inglese. La quasi totalità
degli articoli sull’argomento è stata pubblicata negli
ultimi 3 anni; ad esempio la ricerca in PubMed con la
161
S. Banfi, B. Franco
parola chiave “genome editing” ha prodotto 1539 articoli, di cui solo 93 (6%) prima del 2013. Utilizzando
la parola chiave “Crispr Cas9” sono stati ottenuti 1505
articoli di cui solo 4 prima del 2013.
Introduzione: cosa significa
Genome Editing
Genome Editing (d’ora in avanti abbreviato in GE) è un
termine inglese che indica la possibilità di modificare
in modo permanente il DNA di una cellula. Esso è reso
possibile dall’utilizzo di vere e proprie “forbici molecolari” che determinano un processo di “taglio” della doppia
elica del DNA. Quest’ultimo è seguito da una riparazione che non porterà al ripristino della sequenza di
partenza, ma a una sua variante modificata. Per poter parlare di GE vero e proprio, tuttavia, è necessario
che l’intero processo sia controllato e guidato e che ci
sia la possibilità di effettuare il taglio, e la conseguente
riparazione, in regioni specifiche del genoma che siano predeterminate in partenza. Negli ultimi anni sono
stati pubblicati moltissimi lavori scientifici che discutono di risultati ottenuti con tecniche di GE. L’esplosione
di questo settore delle biotecnologie mediche è stata
causata da due fattori principali. Il primo e più ovvio fattore, relativamente meno recente, è stato rappresentato dalla decodifica della sequenza del genoma dell’uomo e di altre organismi di riferimento per la ricerca sia
di base che applicativa medica, come ad esempio il
topo, il moscerino della frutta (Drosophila) per citarne
solo alcuni, che è stato possibile ottenere a partire dal
1999 fino ai primi anni del ventunesimo secolo (Lander
et al., 2001). Senza la conoscenza della mappa dettagliata dei genomi non sarebbe possibile pianificare
nessun intervento mirato di GE. Il secondo, molto più
recente, è stato rappresentato dalla scoperta di “forbici
molecolari” molto maneggevoli che possono facilmente essere indirizzati a operare in qualsiasi regione genomica desiderata, anche da parte di laboratori piccoli
e non dotati di attrezzature particolarmente sofisticate. Questo è il caso soprattutto del sistema CRISPR/
CAS9, che è alla base dell’esplosione delle applicazioni di GE negli ultimi 3-4 anni e che verrà trattato più
estesamente nella prossima sezione.
Evoluzione tecnologica degli
approcci di GE e descrizione
del sistema CRISPR CAS9
Metodi per modificare il genoma di cellule e organismi
sono state utilizzati già da molti anni dai ricercatori,
soprattutto per effettuare approcci di reverse genetics.
Quest’ultima è una strategia che consiste nello studiare le funzioni di un gene analizzando gli effetti che
una sua alterazione, o addirittura inattivazione, provoca in cellule o in interi organismi. Si contrappone alla
strategia opposta (forward genetics), in cui si parte
162
dalla valutazione di un fenotipo alterato (tipicamente
un quadro patologico) e si cerca di risalire alle sue
basi genetiche identificandone il gene responsabile.
Le procedure che venivano utilizzate per effettuare
GE negli ultimi due decenni del secolo scorso erano però applicabili o a modelli cellulari molto semplici
(es. lieviti) con limitata rilevanza per lo studio di malattie umane oppure, quando applicabili a organismi più
vicini all’uomo (es. gene targeting nel topo, (Bradley,
1991)) richiedevano capacità tecniche elevate e tempi di attuazione molto lunghi che ne limitavano enormente l’utilizzo.
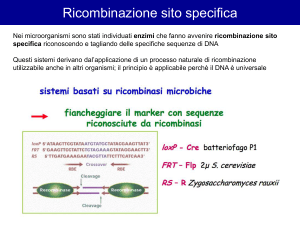
Uso delle nucleasi in GE
Un notevole avanzamento nelle procedure di GE si è
avuto quando è stato introdotto l’utilizzo delle “nucleasi”, vale a dire proteine in grado di rompere il doppio
filamento del DNA in punti specifici (le cosiddette “forbici molecolari” di cui sopra). Ma per quale motivo le
nucleasi possono attivare un processo di GE? Semplicemente perché la rottura del DNA non può essere tollerata dalla cellula, la quale immediatamente attiva un
processo di riparazione (Fig. 1). Il processo di riparazione può avvenire mediante le due seguenti modalità.
1) “Saldatura delle estremità non omologhe” (nonhomologous end-joining). Questa modalità agisce rapidamente e in maniera molto efficace e non si avvale
di una molecola di DNA che funga da stampo. Tuttavia la saldatura delle estremità non-omologhe ha alte
probabilità di introdurre differenze ed errori rispetto
alla sequenza del DNA prima della rottura. Qualora la
rottura si dovesse verificare all’interno di un gene che
produce una proteina, un probabile effetto del processo di saldatura non-omologa sarà la mutazione del
gene e la possibile inattivazione della proteina corrispondente, con importanti applicazioni nel campo
della reverse genetics, come discusso in seguito.
2) Ricombinazione omologa” (homologous recombination). Questa modalità di riparazione del DNA richiede la presenza di una molecola di DNA stampo
identica (o quasi) a quella pre-rottura che può essere
anche fornita esternamente. Questa modalità è meno
efficiente rispetto alla saldatura non-omologa, ma è
in grado di riparare il DNA ricopiando perfettamente,
senza alcun errore, la sequenza della molecola fornita come stampo. È proprio quest’ultima modalità che
fornisce le maggiori prospettive per l’utilizzo del GE a
scopi terapeutici, come sarà discusso in seguito.
Dai primi anni ’2000, sono state introdotti sistemi di
GE basati su tipi diversi di nucleasi, tra cui le nucleasi
a dita di zinco (zinc finger) (Chevalier et al., 2002)
o le nucleasi che mimano gli effettori dell’attivazione
transcrizionale (TALEN, acronimo di Transcription
activator-like effector nucleases) (Boch et al., 2009).
Entrambi questi sistemi presentano un’ottima efficacia, ma hanno lo svantaggio di richiedere la produzione di proteine specifiche per ogni sequenza che si
Genome Editing nella medicina del futuro: opportunità e rischi
Figura 1. Schema raffigurante i sistemi di riparazione del DNA basati su “Saldatura delle estremità non omologhe” (a
sinistra) e della “ricombinazione omologa” (a destra) susseguenti a un taglio della doppia elica di una ragione di DNA
genomico operata da una nucleasi. In indica la comparsa di una mutazione che porta alla inattivazione del gene successiva alla saldatura delle estremità non omologhe. Il rettangolo rosso indica la correzione operata dalla ricombinazione
omologa di una mutazione pre-esistente all’evento di GE, indicata con Mut. Il DNA stampo è la molecola di DNA che
guida il processo di ricombinazione omologa e che contiene la modifica desiderata per realizzare la correzione genica.
voglia tagliare, e comportano quindi dei costi elevati,
che non sono alla portata di laboratori medio-piccoli.
Una vera rivoluzione nel campo si è avuta con la
scoperta del sistema di nucleasi CRISPR/CAS9 e il
suo adattamento a metodiche di GE (Hsu et al., 2014;
Sander e Joung, 2014; Wright et al., 2016).
Scoperta del sistema CRISPR/CAS
CRISPR (abbreviazione di Clustered, Regularly Interspaced, Short Palindromic Repeat, pronuncia crisper)
è una sorta di semplice ma ingegnoso sistema immunitario che i batteri usano per difendersi dai virus. È
costituito da una serie di sequenze genomiche ripetute
intervallate a frammenti di sequenze virali. Questa regione produce dei piccoli RNA noncodificanti per proteine denominati crRNA. Se la cellula batterica viene
infettata da un virus la cui sequenza genomica è riconosciuta da uno di questi crRNA noncodificanti si avrà
la formazione di una molecola ibrida crRNA-DNA virale. La parte ripetuta del crRNA ha un segnale di riconoscimento per una nucleasi CAS (abbreviazione per
CRISPR-associated), così denominata perché il gene
corrispondente è adiacente alla regione CRISPR nel
genoma batterico, che taglia il DNA virale e pertanto
neutralizza l’infezione (Barrangou et al., 2007). L’intero
sistema ha preso il nome di CRISPR/CAS.
Adattamento del sistema CRISPR/CAS9
a procedure di GE
Dalla scoperta del sistema CRISP/CAS e delle sue
modalità di funzionamento al suo adattamento per
utilizzo in GE il passo è stato molto breve. In questo
sistema il riconoscimento del DNA da tagliare non avviene tramite la nucleasi (come nel caso di TALEN e
zinc finger), ma tramite una piccola sequenza di RNA
ed è noto in biologia che le sequenze di RNA sono
oggetto di più facile manipolazione genetica rispetto
alle proteine. Questa caratteristica ha immediatamente aperto delle immense prospettive alle tecniche di
GE. In particolare, il sistema è stato adattato in modo
da sostituire alle sequenze virali contenute nei crRNA
degli RNA sintetici di 18-20 basi (denominati RNA guida o sgRNA) corrispondenti a una qualsiasi sequenza genomica specifica bersaglio. Come nucleasi, si
può utilizzare la proteina CAS9 (prodotta dal batterio
Streptococcus pyogenes) che è molto maneggevole
a causa delle sue ridotte dimensioni. Una volta ingegnerizzato il sistema, ora denominato CRISPR/CAS9
(Fig. 2), si è dimostrato funzionare perfettamente anche in cellule non-batteriche incluse le cellule umane
(Cong et al., 2013; Lander, 2016).
L’attività sperimentale di laboratori in tutto il mondo ha
dimostrato che questo sistema è facilmente introducibile in qualsiasi tipo di cellula utilizzando metodiche
standard di biologia molecolare. Per effettuare GE
tramite CRISPR/CAS9 è necessario semplicemente
disegnare un RNA guida che riconosca la regione genomica bersaglio. Nel caso in cui si voglia produrre
una specifica modifica (es. riproduzione di una data
mutazione) si dovrà anche disegnare e generare un
DNA stampo, operazione anch’essa molto semplice e
alla portata di laboratori non particolarmente attrezzati e dotati di risorse standard per condurre esperimenti di biologia molecolare.
163
S. Banfi, B. Franco
Figura 2. Schema raffigurante l’azione di un sistema basato su CRISPR/CAS9 in una cellula. sgRNA = RNA guida (single guide RNA): si tratta della piccola molecola di RNA che media il riconoscimento della regione genomica bersaglio e
richiama la proteasi CAS9 per il taglio del DNA. Al taglio possono poi seguire i processi di riparazione basati o su “saldatura delle estremità non omologhe” o su “ricombinazione omologa”, a seconda dell’aggiunta o meno di un DNA stampo.
Applicazioni del GE:
studio della funzione di geni
La tecnologia CRISPR/CAS9 consente di generare in
maniera piuttosto veloce modelli biologici cellulari e
animali per lo studio della funzione di geni utilizzando
pertanto strategie di reverse genetics (vedi sopra).
questi approcci in cellule umane (Shalem et al., 2014;
Wang et al., 2014). Un’ulteriore applicazione di tale
tecnologia, considerata la sua rapidità ed efficienza,
potrebbe essere anche, nell’immediato futuro, la possibilità di studiare gli effetti di aplotipi individuati con
tecniche di GWAS in malattie complesse poligeniche
e multifattoriali.
Modelli cellulari
Modelli animali
È ora possibile, tramite il sistema CRISPR/CAS9
creare, per la prima volta, modelli di cellule umane
con inattivazione specifica di un gene d’interesse (ad
esempio un gene responsabile di una malattia genetica). Ciò può essere facilmente ottenuto disegnando
un RNA guida specifico per il gene da studiare e poi
sfruttando il processo di riparazione nonhomologous
end-joining (Fig. 1), che porterà alla sua inattivazione in una linea cellulare. L’efficienza della tecnologia
CRISPR/CAS9 è così elevata da rendere possibili approcci di genomica funzionale che richiedono l’inattivazione completa e simultanea di un numero considerevole di geni, allo scopo di studiare uno specifico
fenomeno biologico. Il trasferimento di RNA guida
diretti contro tutti i geni del genoma umano (insieme
alla CAS9), reso possibile dall’utilizzo di vettori virali
(lentivirus), può essere utilizzato per modificare migliaia di componenti genomiche allo stesso tempo.
Recenti publicazioni hanno dimostrato la fattibilità di
Per la generazione di modelli animali la proteina
CAS9 e gli RNA guida specifici possono essere iniettati direttamente in zigoti fertilizzati di specie animali
comunemente usati nella ricerca medica (es. topo,
zebrafish, Drosophila ecc.), per ottenere la mutazione
(o inattivazione) di uno o più geni che possa poi essere trasmessa anche alle generazioni successive. Tale
tecnologia permette di semplificare notevolmente e
accorciare in maniera significativa i tempi di generazione di modelli animali di malattie genetiche basati
al momento sull’uso della ricombinazione omologa in
cellule staminali embrionali (ES). Come precedentemente riportato, la tecnica è molto versatile e permette anche di ottenere facilmente non solo inattivazioni
di singoli geni, ma anche riarrangiamenti cromosomici più complessi (ad esempio grandi delezioni e/o
duplicazioni del DNA genomico), con significative implicazioni anche per lo studio del cancro. Procedure
basate sul metodo CRISPR/CAS9 sono state infatti
164
Genome Editing nella medicina del futuro: opportunità e rischi
utilizzate con successo anche per riprodurre in vivo
riarrangiamenti genomici alla base di forme tumorali, come ad esempio cancro polmonare (Maddalo et
al., 2014). Infine, come riportato per i modelli cellulari, il sistema CRISPR CAS9 permetterà nel prossimo
futuro anche di generare modelli animali utili per lo
studio di fenotipi complessi, grazie alla sua possibilità
di manipolare più geni contemporaneamente (Niu et
al., 2014).
La facilitazione consentita dalle nuove tecniche di GE
nella generazione di modelli in vitro e in vivo per lo
studio del ruolo biologico di geni e di regioni cromosomiche importanti per il mantenimento dello stato di
salute avrà un sicuro impatto sullo sviluppo di nuove
strategie terapeutiche per le malattie umane.
Potenziali applicazioni
terapeutiche del GE
Il campo però in cui il GE ha il più grosso potenziale di
applicazione nell’ambito delle biotecnologie mediche
è senza dubbio quello terapeutico. Ciò non soltanto
per malattie genetiche causate da mutazioni in singoli geni (malattie monogeniche o mendeliane), che
rappresentano, in linea di principio, il bersaglio più
ovvio ma anche per malattie non genetiche, quali ad
esempio le malattie infettive. Esistono dei dati molto
incoraggianti al riguardo, di cui saranno riportati solo
alcuni casi paradigmatici di seguito (Porteus, 2016).
Naturalmente, trattandosi di un campo giovanissimo,
c’è ancora una lunga strada da percorrere e molti
problemi da risolvere prima di arrivare all’introduzione
ufficiale nella pratica medica di protocolli terapeutici
basati sull’utilizzo del sistema CRISPR/CAS9.
Su quali malattie ha maggiori probabilità
di successo un trattamento basato
su GE?
Il disegno di una strategia terapeutica basata su GE
con le maggiori probabilità di successo deve tenere
conto di una serie di fattori, il primo dei quali è rappresentato dall’accessibilità spazio-temporale del tessuto/organo bersaglio della malattia da trattare. Alcuni
organi e tessuti sono più facilmente accessibili all’introduzione di sistemi CRISPR/CAS9 con obiettivi terapeutici. Tra i tessuti/organi più facilmente “aggredibili”
ci sono la cute, il sangue, l’occhio, il muscolo e il fegato. Sono già stati riportati esempi preclinici (in modelli
murini oppure in sistemi cellulari umani) di trattamento efficace. A questo riguardo, casi esemplari sono
rappresentati da modelli di tirosinemia ereditaria (sul
fegato) (Yin et al., 2016), distrofia muscolare di Duchenne (sul muscolo scheletrico) (Long et al., 2016)
e talassemia (su sangue) (Xie et al., 2014). Al contrario, altri tessuti, incluso quello cerebrale, sono più
difficimente accessibili all’introduzione di un sistema
CRISPR/CAS9 a scopi terapeutici. Per questi tessuti,
è necessario migliorare l’efficacia e la sicurezza delle
metodiche di trasferimento molecolare, prima di potere considerare la fattibilità di un intervento terapeutico
basato su GE. Un altro aspetto molto importante da
considerare è rappresentato dalla finestra temporale
in cui l’intervento di GE può avere delle prospettive di
successo. In questo senso una malattia con esordio
tardivo e decorso più lento ha delle migliori prospettive rispetto a una malattia a esordio precoce (anche
intrauterino) e decorso più rapido.
Uso terapeutico dell’inattivazione
genica mediata da GE
Come precedentemente riportato, il sistema di riparazione del DNA dopo rottura operata da nucleasi,
basato su nonhomologous end-joining (Fig. 1) è il più
rapido ed efficace nella cellula, ma determina molto
facilmente la comparsa di mutazioni con conseguente
inattivazione genica. Tuttavia, vi sono delle malattie in
cui l’inattivazione specifica di un gene o di un frammento genico (es. esone alternativo) può avere degli
effetti terapeutici e questi sono i casi in cui l’applicazione di sistemi di GE può avere delle ottime prospettive di successo.
Un esempio eclatante in tal senso è rappresentato
da una malattia non genetica, vale a dire l’infezione da virus HIV, per la quale l’inattivazione del gene
codificante per la proteina CCR5 sembra avere un
effetto terapeutico. CCR5 svolge un ruolo essenziale
per l’ingresso del virus nella cellula e pertanto la sua
completa inattivazione, mediante GE, svolge un ruolo protettivo/curativo nei confronti della infezione da
HIV. Una procedura di inattivazione di CCR5 basata
sull’utilizzo di nucleasi a dita di zinco è già in fase I di
sperimentazione clinica (Tebas et al., 2014), mentre
altri protocolli basati sull’utilizzo di CRISPR/CAS9
stanno dando risultati molto promettenti (Mandal et
al., 2014).
Un altro esempio di applicazione terapeutica preclinica del sistema di riparazione nonhomologous endjoining è rappresentato da quello praticato in una
delle malattie genetiche più frequenti, vale a dire la
distrofia muscolare di Duchenne (DMD), causata da
mutazioni nel gene codificante per la distrofina e trasmessa con modalità recessiva legata al cromosoma X. Risultati molto promettenti sono stati ottenuti
su un modello murino di DMD (modello dmx) con la
rimozione, mediante un sistema CRISPR/CAS9 degli
esoni contenenti mutazioni deleterie, con conseguente ricostituzione di una distrofina parzialmente funzionante (Long et al, 2016).
Infine, un ultimo e importante campo di applicazione
di procedure di GE basate su nonhomologous endjoining può essere rappresentato da malattie dominanti, in cui solo uno dei due geni è mutato. In questo
caso, la specifica inattivazione della copia mutata del
165
S. Banfi, B. Franco
gene avrebbe un effetto curativo della malattia. Il problema da risolvere in questo caso è trovare il modo
di agire specificamente sulla copia mutata del gene,
lasciando intatta la copia normale.
Uso terapeutico del GE con preciso
ripristino di una sequenza desiderata
Un approccio terapeutico alternativo basato su GE,
e anche concettualmente più ovvio rispetto alla inattivazione genica, prevede la vera e propria correzione”
della mutazione/i (“correzione genica”) alla base della
malattia genetica che s’intende trattare.
Per operare una strategia di questo tipo, è necessario ricorrere al sistema di riparazione del DNA basato
sulla “ricombinazione omologa” (Fig. 1). In pratica si
dovrà generare un RNA guida specifico per la regione
di DNA contenente la specifica mutazione da trattare e
iniettarlo, insieme alla CAS9, nel tessuto bersaglio della malattia provocata dalla mutazione stessa. L’obiettivo è quello di “cancellare” la mutazione ricostituendo la
sequenza genomica normale. Per fare ciò sarà necessario attivare il sistema di riparo della ricombinazione
omologa, che sarà stimolato dalla iniezione contestuale all’RNA guida e alla proteina CAS9 di un frammento
di DNA stampo che conterrà la sequenza di DNA normale e privo della mutazione da correggere. Il risultato
finale sarà una vera e propria cura della malattia nelle
cellule iniettate. Questo approccio potrebbe essere ideale per la terapia di malattie genetiche a trasmissione
dominante, vale a dire quelle causate da mutazioni
in una singola copia di un gene (es. neurofibromatosi
o rene policistico). Ma anche le malattie genetiche a
trasmissione recessiva, in cui entrambe le copie di un
dato gene sono mutate, rappresentano un bersaglio
idoneo per un approccio di GE mirato alla correzione
di una specifica mutazione. Nella quasi totalità delle
malattie recessive è sufficiente la correzione della mutazione di un’unica copia del gene per curare la patologia (come anche dimostrato dal fatto che i portatori di
una sola mutazione non presentano in genere alcuna
anomalia). Esempi incoraggianti che testimoniano l’efficace applicazione preclinica di GE mirato alla correzione di specifiche mutazioni in malattie autosomiche
recessive sono stati riportati per la tirosinemia (Yin H
et al, 2016), l’anemia falciforme (Zou et al., 2011) e l’anemia di Fanconi (Osborn et al., 2015) tra gli altri (vedi
anche Porteus, 2016).
Procedure di correzione genica basate su GE hanno
un più ampio potenziale di applicazione in medicina
rispetto a quelle, pur utili, di inattivazione genica. Il
problema più importante della correzione genica è
al momento rappresentato dalla sua più bassa efficienza rispetto ai sistemi di GE mirati all’inattivazione
genica. Ciò perché il sistema di riparo del DNA della
ricombinazione omologa su cui su basa la correzione
genica è molto più complesso e inefficiente a livello
cellulare, rispetto al sistema di nonhomologous end166
joining. La percentuale media di cellule che acquisiscono la correzione desiderata, utilizzando le procedure oggi correntemente utilizzate, si aggira su valori
insufficienti a generare un significativo effetto terapeutico per una grossa porzione di malattie genetiche
bersaglio (Lombardo e Naldini, 2014). Sono pertanto
richiesti ulteriori miglioramenti tecnologici per le strategie di somministrazione a cellule e tessuti di sistemi
CRISPR/CAS9 per consentire una più diffusa applicazione della correzione genica mediante approccio
GE a malattie umane.
Vantaggi del GE e problemi
da risolvere
Futuri approcci terapeutici di malattie genetiche basati su GE presentano indubbiamente molti potenziali
vantaggi rispetto a procedure di terapia genica basate
sul metodo del trasferimento genico (gene transfer),
sviluppate e applicate con successo negli ultimi 10
anni (Trapani et al., 2015). Queste ultime consistono
nella introduzione nel tessuto bersaglio di un paziente
di una versione normale del gene mutato nel paziente, utilizzando le stesse procedure di trasferimento
applicabili a un sistema di GE. In linea generale il trasferimento genico può essere applicato solo a malattie genetiche in cui si debba sostituire un gene non
funzionante (più frequentemente malattie recessive).
Inoltre il trasferimento genico non riesce a riprodurre
un funzionamento fisiologico del gene introdotto, in
quanto fornito dall’esterno e pertanto sganciato dalla sua normale regolazione all’interno della cellula.
Entrambe queste limitazioni vengono a cadere con
gli approcci GE che quindi presentano un campo di
applicazione esteso sia alle malattie dovute a inattivazione, che a quelle dovute al funzionamento anomalo
di un gene che va inattivato e non sostituito, come
precedentemente descritto. Inoltre, come ultima considerazione, è necessario sottolineare che la correzione mirata del genoma mediante GE permette una
maggiore preservazione del contesto genomico e, in
linea teorica, una migliore conservazione dei meccanismi fisiologici di azione del gene bersaglio.
Tuttavia, sono da considerare numerosi aspetti critici per l’utilizzo terapeutico di GE, e in particolare di
CRISPR/CAS9. Tra i principali sono da annoverare
quelli tecnologici, già precedentemente menzionati,
vale a dire la necessità di: 1) migliorare le modalità
di somministrazione ad alcuni tessuti più difficilmente
accessibili come il cervello e 2) incrementare l’efficacia delle procedure basate sulla correzione genica, al
momento ancora insufficienti.
Un altro aspetto cruciale è rappresentato dalla ancora scarsa conoscenza dei possibili effetti non-specifici (cosiddetti off-target) del GE sul genoma. Infatti,
è stato dimostrato che l’sgRNA può riconoscere in
modo erroneo altre regioni del genoma, oltre a quella
bersaglio, e pertanto causare un taglio da parte della
Genome Editing nella medicina del futuro: opportunità e rischi
CAS9 (Kuscu et al., 2014), che potrebbe avere delle
conseguenze disastrose nel caso in cui colpisse geni
o regioni con ruolo biologico critico nel mantenimento
dello stato di benessere. Purtroppo, a fronte di un’enorme facilità di utilizzo del sistema, non è ancora
semplice determinare preventivamente, in modo attendibile, se l’sgRNA da utilizzare abbia la possibilità
di causare importanti effetti off-target. Sono tuttavia disponibili approcci alternativi di GE basati sull’utilizzo
di sgRNA e proteine CAS modificate che dovrebbero
portare alla minimizzazione della comparsa di effetti
non-specifici operati dai sistemi di GE (Kleinstiver et
al., 2016). In ogni caso, una valutazione più puntuale
e dettagliata della reale incidenza e significato degli
effetti off-target conseguenti all’utilizzo di procedure
basate su GE dovrà costituire un requisito essenziale
prima della loro introduzione nella partica medica.
Implicazioni etiche del GE
Lo sviluppo della tecnologia di GE basato sul sistema CRISPS/CAS9 potrebbe determinare, nell’ambito
della ricerca biomedica, una rivoluzione, paragonabile
a quella provocata alla fine degli anni ’80 del secolo
scorso dalla introduzione della Reazione a Catena della Polimerasi (PCR). La sua caratteristica più notevole
è che si tratta di una tecnica poco costosa, facile da
attuare, alla portata anche di piccoli laboratori di biologia molecolare, senza sofisticate attrezzature scientifiche, né dotati di importanti risorse economiche. Dato
che si tratta di una metodica in grado di modificare in
modo permanente il genoma di qualsiasi tipo di cellula,
si possono aprire degli scenari inquietanti e si stanno
già sollevando dibattiti molto accesi sulle implicazioni
etiche di un suo uso improprio o incontrollato.
Tutte le potenziali applicazioni terapeutiche precedentemente descritte prevedono l’utilizzo del GE su cellule di tessuti già formati, le cui modifiche genomiche
non saranno trasmissibili a generazioni successive.
Tuttavia, la metodica del CRISPR/CAS9 può essere
in teoria applicata a embrioni umani e grande sconcerto ha suscitato la notizia che un gruppo di ricercatori cinesi è stato in grado di modificare il genoma di
embrioni umani proprio con questa tecnica (Liang et
al., 2015). Pur trattandosi di embrioni umani ottenuti
da centri di fertilità, non in grado quindi di sopravvivere a causa di importanti alterazioni cromosomiche,
la notizia ha destato grosse preoccupazioni e ha dato
inizio a importanti discussioni all’interno della intera
comunità scientifica sulla necessità di una regolamentazione dell’uso di queste nuove tecniche di GE
(Cyranoski e Reardon, 2015; Ledford, 2015).
In conclusione, la scoperta del sistema CRISPR/
CAS9 e la sua applicazione a procedure di GE possono potenzialmente rivoluzionare la ricerca biomedica e la pratica clinica in un futuro non molto lontano.
Tuttavia, la risoluzione di alcuni problemi tecnici e una
attenta valutazione delle possibili conseguenze di un
uso improprio di questa metodica sembrano essere
necessari, al fine di un utilizzo più generalizzato.
Box di orientamento
• Cosa sapevamo prima
Prima della scoperta del sistema CRISPR/CAS9, il Genome Editing era considerata una metodica molto
complessa utilizzabile solo da laboratori molto attrezzati e specializzati nel settore.
• Cosa sappiamo ora
Il sistema CRISPR/CAS9 è una procedura molto semplice e poco costosa, alla portata di tutti i laboratori
in grado di fare esperimenti di biologia molecolare. Questa tecnica permette di modificare il genoma sia
per effettuare studi di ricerca volti alla comprensione dei meccanismi di funzionamento dei geni, che per
disegnare potenziali strategie terapeutiche per malattie umane (non esclusivamente di origine genetica).
• Quali sviluppi si possono prevedere per il futuro
Un’estesa applicazione di queste metodiche per la cura di molte malattie, previa però la risoluzione di
alcuni aspetti tecnici e una aperta discussione di alcune possibili implicazioni di natura etica.
Bibliografia
Barrangou R, Fremaux C, Deveau H, et
al. CRISPR provides acquired resistance
against viruses in prokaryotes. Science
2007;315:1709-12.
*** Una delle prime pubblicazioni in cui
viene decifrato il ruolo del sistema CRISPR/CAS9 nella protezione delle cellule
batteriche da infezioni virali.
Boch J, Scholze H, Schornack S, et al.
Breaking the code of DNA binding specificity of TAL-type III effectors. Science
2009;326:1509-12.
Bradley A. Modifying the mammalian
genome by gene targeting. Curr Opin Biotechnol 1991;2:823-9.
Chevalier BS, Kortemme T, Chadsey MS,
et al. Design, activity, and structure of a
highly specific artificial endonuclease. Mol
Cell 2002;10:895-905.
Cong L, Ran FA, Cox D, et al. Multiplex
genome engineering using CRISPR/Cas
systems. Science 2013;339:819-23.
Cyranoski D, Reardon S. Embryo editing
sparks epic debate. Nature 2015;520:593-4.
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-
167
S. Banfi, B. Franco
Cas9 for genome
2014;157:1262-78.
engineering.
Cell
Kleinstiver BP, Pattanayak V, Prew MS,
et al. High-fidelity CRISPR-Cas9 nucleases
with no detectable genome-wide off-target
effects. Nature 2016;529:490-5.
Kuscu C, Arslan S, Singh R, et al. Genome-wide analysis reveals characteristics
of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol 2014;32:677-83.
Lander ES. The Heroes of CRISPR. Cell
2016;164:18-28.
** Dettagliata cronistoria della scoperta
del sistema CRISPR/CAS9 e del suo adattamento a metodiche di GE in cellule animali.
Lander ES, Linton LM, Birren B, et al. Initial sequencing and analysis of the human
genome. Nature 2001;409:860-921.
Ledford H. CRISPR, the disruptor. Nature 2015;522:20-4.
** Importante esempio di uso terapeutico del sistema CRISPR/CAS9 in un modello animale di distrofia muscolare.
Maddalo D, Manchado E, Concepcion
CP, et al. In vivo engineering of oncogenic
chromosomal
rearrangements
with the CRISPR/Cas9 system. Nature
2014;516:423-7.
Mandal PK, Ferreira LM, Collins R, et al.
Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9. Cell Stem Cell 2014;15:643-52.
Niu Y, Shen B, Cui Y, et al. Generation
of gene-modified cynomolgus monkey via
Cas9/RNA-mediated gene targeting in onecell embryos. Cell 2014;156:836-43.
Osborn MJ, Gabriel R, Webber BR, et
al. Fanconi anemia gene editing by the
CRISPR/Cas9 system. Hum Gene Ther
2015;26:114-26.
*** Riassunto delle principali problematiche di natura etica derivanti dall’utilizzo
del sistema CRISPR/CAS9.
Porteus M. Genome editing: a new approach to human therapeutics. Annu Rev
Pharmacol Toxicol 2016;56:163-90.
Liang P, Xu Y, Zhang X, et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell
2015;6:363-72.
** Dettagliata revisione della letteratura sugli iniziali successi, potenziali future
applicazioni e problemi da risolvere per
l’utilizzo terapeutico del sistema CRISPR/
CAS9.
Lombardo A, Naldini L. Genome editing:
a tool for research and therapy: targeted
genome editing hits the clinic. Nat Med
2014;20:1101-3.
Long C, Amoasii L, Mireault AA, et al.
Postnatal genome editing partially restores dystrophin expression in a mouse
model of muscular dystrophy. Science
2016;351:400-3.
Sander JD, Joung JK. CRISPR-Cas
systems for editing, regulating and
targeting genomes. Nat Biotechnol
2014;32:347-55.
Shalem O, Sanjana NE, Hartenian E, et
al. Genome-scale CRISPR-Cas9 knockout
screening in human cells. Science
2014;343:84-7.
Tebas P, Stein D, Tang WW, et al. Gene
editing of CCR5 in autologous CD4 T cells
of persons infected with HIV. N Engl J Med
2014;370:901-10.
** Prima descrizione di risultati positivi ottenuti mediante una strategia di GE
mirata alla terapia dell’infezione dal virus
HIV.
Trapani I, Banfi S, Simonelli F, et al. Gene
therapy of inherited retinal degenerations:
prospects and challenges. Hum Gene Ther
2015;26:193-200.
Wang T, Wei JJ, Sabatini DM, et al. Genetic screens in human cells using the CRISPR-Cas9 system. Science 2014;343:80-4.
Wright AV, Nunez JK, Doudna JA. Biology and applications of CRISPR Systems:
harnessing nature’s toolbox for genome
engineering. Cell 2016;164:29-44.
*** Dettagliata revisione della letteratura sulle applicazioni del sistema CRISPR/
CAS9 nella ricerca di base.
Xie F, Ye L, Chang JC, et al. Seamless
gene correction of beta-thalassemia mutations in patient-specific iPSCs using
CRISPR/Cas9 and piggyBac. Genome Res
2014;24:1526-33.
Yin H, Song CQ, Dorkin JR, et al. Therapeutic genome editing by combined
viral and non-viral delivery of CRISPR system components in vivo. Nat Biotechnol
2016;34:328-33.
Zou J, Mali P, Huang X, et al. Site-specific gene correction of a point mutation
in human iPS cells derived from an adult
patient with sickle cell disease. Blood
2011;118:4599-608.
Corrispondenza
Sandro Banfi
Genetica Medica, Dipartimento di Biochimica, Biofisica e Patologia Generale, Seconda Università degli Studi di Napoli E-mail: [email protected]
Brunella Franco
Genetica Medica, Dipartimento di Scienze Mediche Traslazionali, Universita di Napoli Federico II - E-mail: brunella.
[email protected]
168