Medicina
Genetica
I. Analisi Genetica nell’uomo
II. Genetica Medica
III. Genomica e postgenomica
IV. Citogenetica Medica
V. Genetica Clinica
VI. Genetica in Medicina Interna
☤
1
MEDICINA GENETICA VERSUS GENETICA MEDICA
La Genetica Medica è basata sullo studio di malattie genetiche rare, ereditate in modo “mendeliano”. La Medicina Genetica implica il fatto che la genetica pervade tutta la Medicina, incluse le malattie comuni come l’ipertensione, il cancro, le malattie cardiovascolari, etc. (che vengono a configurarsi come malattie genetiche complesse).
I. ANALISI GENETICA NELL’UOMO
The Human Genome Project
Proposto nel 1985; tra il 1986 e 1989 fu discusso, dibattuto e pianificato. La data di inizio ufficiale fu il 1 Ottobre 1990. Il 30 Settembre 2005 il progetto è giunto a termine.
•
•
•
•
1970
1980
1990
2000
2010
“Gene”: introdotto da Johansson a Copenhagen per 1960
indicare l'“unità ereditaria” di Mendel (1909).
“Genetica”: introdotto da Bateson in Inghilterra ‘Chromosomology’
1956
come scienza dell'ereditarietà (1905).
Somatic Cell Genetics
“Genoma”: usato per la prima volta da Winkler in Molecular Genetics
Germania (1920) per indicare “GENes and chromosOME”, ovvero l'insieme completo dei Transgenic, KO, etc, mice
cromosomi e dei geni che essi contengono.
Database searching
“Genomica”: studio strutturale e funzionale del Microarray technology
genoma (Roderick, Bar Harbor, 1986).
Siamo entrati nell’era post­genomica: finora le tecnologie disponibili permettevano l’analisi di singoli geni, oggi possiamo analizzare il funzionamento di migliaia di geni.
•
2
IL CALCOLO DEL RISCHIO Caratteri AD: la probabilità di trasmettere il carattere è del 50% (1/2). Il gene della malattia è facile da identificare, essendoci delle mutazioni ricorrenti che ci permettono di arrivare al genotipo dell'individuo in esame. ☤ Alessandro G. - 2011/2012
•
•
•
Caratteri AR: i due genitori portatori hanno il rischio del 25% (1/4) di malattia nel figlio. Il rischio del figlio di essere portatore è di 2/3. Caratteri X­linked: Una madre portatrice ha il rischio del 50% di figli maschi malati e del 50% di figlie femmine portatrici. Una femmina con un fratello ed un figlio malati è necessariamente una portatrice.
Nel caso della Distrofia Muscolare di Duchenne (DMD), nei maschi la diagnosi è semplice: con la multiplex PCR vengono amplificati 4 segmenti del gene della distrofina che poi sono fatti migrare in Southern Blot. Dal momento che nella DMD si tratta perlopiù di delezioni, gli individui affetti avranno meno di 4 segmenti all'elettroforesi. La diagnosi nelle femmine è più complicata per la presenza di due cromosomi X. Possiamo ricorrere (PCR quantitativa a parte) all'analisi dei polimorfismi che affiancano il gene: evidenziati i polimorfismi (a due alleli) che affiancano il gene della distrofina, possiamo riscontrarlo o meno nelle femmine in esame. In questo modo si costruisce una mappa degli aplotipi incluso il cromosoma che porta la mutazione. Legge di Hardy­Weinberg: nel caso di geni a due alleli (frequenza biallelica), A e a, la somma delle frequenze dei due alleli (p e q) è pari a 1.
(Le forme alternative di un gene, ad un dato locus, sono dette alleli)
Possiamo risalire alla frequenza dei genotipi pp, pq e qq. Queste considerazioni valgono in una popolazione a incroci casuali: nelle famiglie con mutazioni mendeliane vale la Legge di Mendel. La legge di Hardy­Weinberg è usata per calcolare il rischio in base alla frequenza allelica. N.B.: per le malattie legate all'X la frequenza di maschi malati è uguale alla frequenza dell'allele X mutato. Frequenza allelica p + q = 1 Frequenza genotipica (p + q)2 = 1 p2 + 2pq + q2 = 1
A
a
Genotipo aa q2 A AA aA
Genotipo AA
p2
a aA aa
Genotipo aA
2 pq
Genetica di popolazione: frequenze di alleli e genotipi nella popolazione
Correlazione tra frequenze alleliche e frequenze dei genotipi
corrispondenti nella popolazione secondo la legge di Hardy-Weinberg
p + q = 1 ⇔ p = 1 – q,
dove q è solitamente una piccola frazione (1 su n) approssimabile a 0: p = 1 ­ (1/n) ≃ 1.
3
Esempi:
Nell’albero riportato l’individuo III3 è affetto da una rara malattia autosomica recessiva letale nei primi mesi di vita che presenta una frequenza nella popolazione di 1/40'000 nati. Calcolate la probabilità per la donna V2 di avere un figlio affetto
a) se sposa suo cugino di III grado V1 b) se sposa un individuo della popolazione generale.
I
II
III
IV
V
a) Bisogna calcolare la probabilità che V1 e V2 siano portatori
II1 e II2 = Aa
II3 = 1/2
→ III2 = 2/3
→ III4 = 1/2 × 1/2 = 1/4
→ IV1 = 2/3 × 1/2 = 1/3
→ IV4 = 1/4 × 1/2 = 1/8
→ V1 = 1/3 × 1/2 = 1/6
→ V2 = 1/8 × 1/2 = 1/16
R = 1/6 × 1/16 × 1/4 = 1/384
b)
2
1/40'000 = q
⇔ q = 1/200
(p + q) = 1 ⇔ (p +
1/200) = 1 ⇔
p = 1 – 1/200 ≃1
2pq= 2 × 1 × 1/200 = 1/100 ← frequenza degli eterozigoti (rischio di popolazione)
R = 1/100 × 1/16 × 1/4 = 1/6'400
Se un disordine recessivo legato al cromosoma X colpisce una donna su 1'000'000 in una popolazione, qual è la frequenza attesa dei maschi affetti? E quella delle donne portatrici?
ҲҲ = q2
XX = p2
XҲ = 2pq
XY = p
ҲY = q
2
ҲҲ = 1/1'000'000 ⇔ 1/1'000'000 = q ⇔ q = 1/1'000
ҲY = 1/1'000
XҲ = 2 × 1 × 1/1'000 = 1/500
POLIMORFISMI DEL DNA Polimorfismo è definito come la presenza, nella popolazione, con frequenza maggiore all'1%, di 2 o più forme (“alleli” o “varianti”) di un gene o di una data sequenza di DNA. I polimorfismi possono essere silenti, ovvero non avere alcun effetto sul fenotipo. Possono essere dovuti a sostituzione, mutazione, o delezione, di una singola base, o alla variazione del numero di “ripetizioni tandem”.
Migliaia di siti polimorfici sono stati identificati nell'ambito dell'Human Genome Project.
4
☤ Alessandro G. - 2011/2012
Lo sviluppo dei marcatori genetici nell'uomo
Tipo di marcatore
N° di loci
Caratteristiche
Gruppi sanguigni
(1910­1960)
~ 20
Può esserci bisogno di sangue fresco, antisieri rari. Il genotipo non può essere desunto dal fenotipo a causa della dominanza. Non facile la localizzazione fisica.
Varianti della mobilità elettroforetica delle proteine del siero
(1960­1975)
~ 30
Può essere necessario siero fresco, saggi specialistici. Localizzazione fisica non facile. Spesso polimorfismo limitato.
Tipi tissutali HLA
(1970)
> 1
(aplotipo)
Un gruppo di associazione. Altamente informativo. Si può analizzare solo il linkage con 6p21.3
RFLP del DNA
(1975)
> 105
2 alleli marcatori. Inizialmente (potenzialmente) richiesto il Southern­blotting, ora la PCR. Facile la localizzazione fisica.
VNTR del DNA
[minisatelliti]
(1985)
>104
Molti alleli, altamente informativi. (potenzialmente) Tipizzazione con Southern­
blotting. Facile localizzazione fisica. Tendono a raggrupparsi vicino alle estremità dei cromosomi.
> 104
Molti alleli, altamente informativi. VNTR del DNA
[microsatelliti (ripetizioni di­, tri­ e (potenzialmente) si possono tipizzare mediante PCR Facile localizzazione fisica. tetra­nucleotidiche)]
Distribuiti lungo tutto il genoma.
(1989)
Ci occupiamo in particolare delle applicazioni dei polimorfismi genetici nello studio delle famiglie affette da malattie genetiche. I polimorfismi usati a questo scopo sono: • RFLP (Restriction­Fragments Length Polymorphisms) Gli enzimi di restrizione tagliano il doppio filamento di DNA a livello di specifiche sequenze palindromiche. Le sequenze palindromiche possono venire interessate da mutazioni che alterano il sito riconosciuto dall'enzima per cui alla digestione vengono generati frammenti di lunghezza diversa da quella canonica.
Tramite il Southern Blot (elettroforesi su gel) i frammenti generati vengono separati in base alla loro lunghezza in bande di migrazione differenti. Gli RFLP sono sistemi a due alleli (il sito di restrizione può esserci o non esserci). 5
•
VNTR (Variable Number of Tandem Repeats) I microsatelliti sono ripetizioni, in numero variabile, di 2 ­ 4 basi che sono sparsi nel genoma.
Il numero di ripetizioni è variabile e quindi questi sistemi sono a più alleli e sono molto informativi; sono utili per le analisi di linkage e di segregazione. Le sequenze ripetute sono affiancate da sequenze uniche che permettono l'identificazione delle ripetizioni stesse.
I microsatelliti più usati sono i CA­repeats (...CACACACA...). Amplificandoli con la PCR e facendoli scorrere in elettroforesi otteniamo bande di migrazione diverse in base al numero delle ripetizioni, cioè in base alla lunghezza del Famiglia Informativa
frammento. Attualmente la lunghezza dei frammenti può essere determinata automaticamente con il sequenziatore. Studiando in una famiglia un polimorfismo situato vicino al gene di nostro interesse possiamo determinare il genotipo dell'individuo. Se il gene malattia nella madre è vicino ad una ripetizione di lunghezza nota, e troviamo la stessa lunghezza di ripetizione nel figlio, vuol dire che il gene della madre è stato *
*
ereditato dal figlio. • SNPs (Single Nucleotide Polymorphisms) Sono variazioni di singole basi e sono sistemi a 2 alleli, ma sono molto frequenti nel genoma (circa 3'000'000 di SNPs). Sono utili per le analisi di linkage e di segregazione; inoltre recentemente si è formato un consorzio con l'obiettivo di tracciare una mappa degli SNPs per cercare come le loro variazioni incidano sulla suscettibilità a diverse malattie.
~15 kb
M = allele di un marker
sullo stesso aplotipo
dell’allele mutato (∗)
6
☤ Alessandro G. - 2011/2012
Linkage disequilibrium mapping: le varianti in una determinata regione genica possono essere non indipendenti: sono in linkage disequilibrium. Markers che sono fisicamente vicini tendono a rimanere associati alla mutazione ancestrale (ovvero: non ricombinano).
Geni e malattie: una nuova dimensione
Variazione “A”: Mutazione → Malattia monofattoriale
Variazione “B”: Polimorfismo → Sano, ma con una proteina che funziona di più o di meno: Suscettibilità
Utilizzo ai fini clinici degli SNP: identificare le varianti geniche che conferiscono un rischio di malattia.
Limiti: il rischio relativo per ogni singolo SNP è modesto; è difficile pesare l’effetto combinato di tutti i polimorfismi di un individuo.
Una “SNPs­map” potrà essere usata per fornire la cura appropriata al paziente appropriato.
ANALISI DI LINKAGE (CONCATENAZIONE)
L'analisi di linkage permette di determinare la posizione cromosomica di un locus responsabile di un determinato carattere rispetto a marcatori polimorfici la cui localizzazione è nota; questo grazie alla co­trasmissione di geni fisicamente vicini sullo stesso cromosoma (eccezione alla seconda legge di Mendel sull'assortimento indipendente). Inoltre permette di seguire, nella famiglia, la segregazione di un allele contenuto nel tratto di genoma tipizzato con i marcatori.
L'analisi di linkage è un approccio molto utile per il mappaggio e l’identificazione di geni responsabili di malattie genetiche Mendeliane (oltre 1'200 geni identificati) e per la diagnosi genetica indiretta (patologie di cui è nota la posizione, ma non la sequenza, del gene malattia), mentre è più difficoltoso il suo uso per lo studio di caratteri complessi come la suscettibilità (malattie complesse).
L'analisi si basa sulla co­segregazione, alla meiosi, di 2 o più loci più frequentemente di quanto ci si aspetti nella segregazione indipendente. Se 2 loci vengono frequentemente ereditati assieme, è probabile che siano vicini sullo stesso cromosoma. La frazione di ricombinazione (ricombinanti/totali) non può mai superare il 50%.
7
Sono un esempio di co­segregazione il locus del gene della cecità per colori (daltonismo) e quello del G6PD (favismo) che si trovano sul cromosoma X. Analisi di linkage di caratteri mendeliani
L'analisi del linkage determina la frequenza (θ) con cui 2 loci ricombinano tra loro alla meiosi, ed è la misura della distanza genetica tra i due loci.
Se due loci sono su cromosomi diversi segregano indipendentemente. La probabilità che vengano ereditati insieme è di ½: θ = 50%.
Se due loci sono vicini fra loro sullo stesso cromosoma, saranno ereditati insieme più frequentemente: θ < 50%. Tanto più sono vicini, tanto più piccola è la probabilità che avvenga un crossing­over. Un riscontro di frazione di ricombinazione < 50% suggerisce la concatenazione. Le frequenze di ricombinazione fra loci piuttosto distanti non sono additive (a causa dell’effetto dei crossing­over multipli).
La distanza genetica è quindi il numero atteso di crossing­overs fra 2 loci per meiosi. L’unità di misura della distanza genetica è il centiMorgan (cM): 1 Morgan = 100 cM.
Per θ < 10% c’è una corrispondenza ~1:1 fra θ e cM: 1 cM ≈ θ = 1%.
Per θ >10% la corrispondenza di 1:1 si perde, ed allora si usano delle funzioni di mappa (relazioni fra frazione di ricombinazione e distanza genetica) per convertire θ in cM e viceversa: si adopera la funzione di Haldane per calcolare la distanza. ln ( 1 − 2θ )
1 − e −2 ω
ω =−
⇔θ=
2
2
L'eterozigosità (H) rappresenta la probabilità che un individuo preso a caso sia eterozigote
H = 1 ­ (p12 + p22 + p32 + p42 + … + pi2)
Informatività
Aa
11
aa
22
Aa
12
aa
12
Aa
Aa
12
12
NON informativa NON informativa
Aa
12
aa
12
Aa
22
Informativa
Aa
1 2
aa
34
Aa
14
Informativa
Per misurare la frequenza di ricombinazione fra due marcatori è necessario che gli eventi 8
☤ Alessandro G. - 2011/2012
meiotici siano informativi, cioè che ci permettano di ricostruire le fasi (le disposizioni degli alleli di due loci adiacenti sullo stesso cromosoma) e di identificare il marcatore concatenato, ed a questo scopo i marcatori parentali devono essere doppi eterozigoti.
A
1
B
1
Ricombinazione
Non ricombinazione
A
1
binazio
Ricom
B
2
Non
ri
c om
bi na
ne
zion
e
A
1
B
1
A
2
B
1
A
1
B
2
MEIOSI
NON
INFORMATIVA
MEIOSI
INFORMATIVA
Una serie di alleli per loci adiacenti che vengono ereditati insieme sullo stesso cromosoma formano un aplotipo.
La tipizzazione di famiglie ampie ed informative, dove segrega una specifica malattia genetica, consente di stabilire se esiste una “concatenazione” tra un marcatore specifico ed il locus malattia.
L’esistenza di un rapporto di concatenazione viene dimostrata tramite vari metodi, tra cui quello del rapporto di massima verosimiglianza (o metodo dei lod score). La base logica di questo metodo è molto semplice, ed è dovuta al fatto che in una progenie si possono osservare due situazioni: o quella del riassortimento indipendente di due geni (o di un marcatore con un gene) o quella della concatenazione degli stessi. I lod scores vengono così calcolati prendendo in considerazione una meiosi alla volta e confrontando la probabilità dei genotipi osservati nelle ipotesi alternative di concatenazione (linkage) o di assortimento indipendente.
Tuttavia anche quando due loci sono localizzati sullo stesso cromosoma bisogna tener conto che si possono osservare fenomeni di ricombinazione per il “crossing­over” che è dovuto alla distanza dei due loci. La percentuale di ricombinazione è tanto maggiore quanto più distanti sono i loci. La percentuale di ricombinazione viene indicata con la lettera greca θ, e l’1% di ricombinazione corrisponde all’unità di mappa o centiMorgan (cM) (dal nome di Morgan che scoprì la concatenazione genica nel 1911).
50% di ricombinazione → θ = 0,5 → assenza di concatenazione
Il metodo del Lod Score calcola l'indice Z, che è un rapporto tra la probabilità che i due loci siano concatenati (L0) e la probabilità che siano indipendenti (L1): Z = Log(L1/L0). Si calcolano cioè due probabilità:
1. la probabilità L0 che la progenie di quella famiglia sia ottenuta in assenza di concatenazione ( θ = 0,5 )
2. la probabilità L1 che la stessa progenie si sia generata in presenza di concatenazione ( θ < 0,5 )
Queste probabilità vengono più esattamente definite come verosimiglianza delle osservazioni nelle due ipotesi di indipendenza e di linkage.
Si calcola quindi il rapporto di verosimiglianza (L = L1/L0 ) e si cerca il valore di θ per cui tale rapporto risulta massimo. Per semplicità di calcolo si usa il logaritmo in base 10 del rapporto 9
di verosimiglianza L, ovvero l'indice Z, che in inglese viene denominato lod score.
La mappatura di una malattia si ottiene così quando il valore di Z è uguale o superiore a 3. In effetti questo valore sta ad indicare che l’ipotesi di concatenazione per un determinato valore di θ è 1000 volte più probabile di quella di indipendenza.
Limiti di significatività:
• Z > 3: evidenza di linkage significativa
• Z < ­2: si può escludere la presenza di linkage
Esempio di conteggio dei ricombinanti
dd
25
Pedigree a fase nota
1. Modello di ereditarietà: deduco i genotipi per il locus malattia
2. La fase è nota 3. Individuo i ricombinanti
Dd
11
dd
34
Dd
12
Dd
13
Dd
13
dd
24
Dd
14
dd
24
Dd
23
NR
NR
NR
NR
NR
R
Pedigree a fase non nota
1. Modello di ereditarietà: deduco i genotipi per il locus malattia
2. La fase non è nota 3. Individuo i ricombinanti
?
Dd Dd
21 12
dd
34
Dd
13
Dd
13
dd
24
Dd
14
NR
NR
NR
R
R
R
10
dd
24
Dd
23
NR
NR
R
R
R
NR
Dd
12
Dd
Se la fase è 2 1
Se la fase è
☤ Alessandro G. - 2011/2012
II. GENETICA MEDICA
ECCEZIONI ALL'EREDITARIETÀ MENDELIANA
“Un gene <≠> una malattia”
Eterogeneità genetica (o di locus)
Il concetto classico di “Eterogeneità genetica” implica che la stessa condizione possa essere causata da mutazioni in geni differenti (Eterogeneità di locus). Per alcune patologie, il grado di eterogeneità genetica è tale che più di 100 geni possono essere responsabili dello stesso fenotipo.
L'Eterogeneità allelica consiste invece nel fatto che mutazioni alleliche in un singolo gene possano produrre un fenotipo simile.
Sordità: circa 1/1'000 nati vivi presentano sordità; il 50% dei casi è dovuto a mutazioni di singoli geni: 2/3 AR, 1/3 AD, 1 ­ 2% X­linked. Esistono delle forme sindromiche, ma la maggior parte delle sordità AR non sono sindromiche. L'eterogeneità genetica è molto ampia: ci sono più di 40 loci per le forme AR e un numero simile per le forme AD, nonché diversi loci per le forme X­
Linked. Circa 2/3 delle sordità AR non sindromiche sono dovute a mutazioni del locus DFNB1 in 13q11­q12 (gene della connessina 26) nelle popolazioni dell'area mediterranea. L'80% di queste presentano la stessa mutazione delG35 all’interno di una sequenza contenente 5 G. Lo studio della connessina nei genitori (individuare le forme mutate del DFNB1) permette di definire meglio il rischio per i figli. Sindrome di Noonan: ha una prevalenza di 1/2'000. È una condizione monogenica a trasmissione autosomica dominante, espressività variabile, eterogeneità genetica. La mutazione coinvolge la via di SOS1 rendendola costantemente attiva avviando cascate di effettori cellulari. All'analisi mutazionale si rilevano PTPN11 (50%), SOS1 (10­13%), K­RAS (< 5%), RAF1 (3%).
La sindrome comprende dismorfismi facciali (fronte alta, ipertelorismo, fessure palpebrali rivolte verso il basso, orecchie ad impianto basso e retroruotate, filtro con disegno accentuato, bordi del labbro superiore sollevati, collo corto con cute nucale ridondante, attaccatura posteriore dei capelli bassa), cardiopatia congenita nel 50­80% dei casi (stenosi valvolare polmonare (20­50%), cardiomiopatia ipertrofica (20­30%), difetti settali, tetralogia di Fallot, etc.; bassa statura (40­50%) per ritardo di crescita post­natale; ritardo mentale/difficoltà di apprendimento (25­35%); anomalie oculari (95%): strabismo, ambliopia, nistagmo; diatesi emorragica (30%); anomalie linfatiche pre­natali (igroma cistico, poliidramnios, idrope fetale) e post­natali; anomale renali (11%): idronefrosi, doppio distretto, agenesia/ipoplasia renale, ectopia, anomalie di rotazione; anomalie cutanee (cheratosi follicolare). I maschi affetti sono fenotipicamente indistinguibili dalle femmine affette dalla sindrome di Turner.
Molti affetti hanno una mutazione de novo, pertanto il rischio per i fratelli è trascurabile (< 1%): il mosaicismo germinale è teoricamente possibile ma non è mai stato riportato in letteratura. Il 30­75% degli affetti ha invece un genitore 11
affetto, con un rischio per i fratelli del 50%.
L'eterogeneità genetica è anche una caratteristica di certe patologie neurodegenerative come l'Atassia spinocerebellare (SCA) o la Paraplegia spastica ereditaria (HSP). Espressività variabile e Penetranza incompleta
L'eterogeneità sul piano clinico configura l'Espressività Variabile: mutazioni differenti nello stesso gene causano differenti fenotipi. Uno stesso locus può essere responsabile di quadri clinici completamente diversi. Differenti membri della stessa famiglia possono mostrare spettri fenotipici differenti della stessa malattia; le cause sono comuni a quelle responsabili della penetranza incompleta: l’influenza di altri geni, o di fattori ambientali, può agire sullo sviluppo dei sintomi di una malattia. Morbo di Hirschprung (HSCR): l'incidenza è di circa 1/5'000 nati vivi. La malattia è caratterizzata dall'assenza delle cellule dei gangli parasimpatici dei plessi sottomucoso e mioenterico nel colon. Clinicamente si presenta con severa distensione addominale, difetti dell'accrescimento ed ostruzione intestinale. La causa molecolare è l'arresto prematuro della migrazione cranio­caudale delle cellule di derivazione della cresta neurale durante la gestazione. L'espressività variabile si manifesta colpendo segmenti del colon progressivamente più distali. Il gene RET (REarranged during Transfection) codifica per una famiglia di recettori tirosin­kinasi (GFR­α1, α2, α3, α4), i cui ligandi (GDNF, NTN, ART, PSP, ...) inducono dimerizzazione ed autofosforilazione, interazione con gli effettori del segnale, attivazione di RAS e delle MAPK. Le mutazioni di RET sono associate all'HSCR, alla MEN2A­FMTC ed alla MEN2B. Tutte le malattie sono AD e la penetranza è completa entro i 70 anni. La Penetranza Incompleta è una frequente complicazione di un carattere dominante: la penetranza di un carattere, per un determinato fenotipo, è definita come la probabilità che una persona, che presenta l’alterazione a livello genotipico, manifesti il carattere. Un carattere con penetranza completa (100%) deve manifestarsi in tutte le persone eterozigoti per quel carattere. La penetranza incompleta sì può descrivere come la presenza di “portatori sani” di una mutazione dominante.
Un indice di penetranza incompleta è il salto di generazione per certi caratteri quando si costruisce un albero genealogico.
È facile spiegare la penetranza incompleta se pensiamo ad un “locus genetico” non isolato, ma in un contesto differente da persona a persona (background genetico), sotto l’influenza di geni che possono modificare il fenotipo (Geni Modificatori).
Oloprosencefalia: è la più comune alterazione dello sviluppo dell'encefalo, un difetto di sviluppo della linea mediana, con incompleta separazione encefalica (dalla completa continuità dei due emisferi, alla separazione parziale, o anche solo una leggera continuità); colpisce 1/16'000 neonati.
La patologia è molto variabile sia geneticamente che fenotipicamente, con presentazioni cliniche che vanno da casi lievi, con solamente un dente incisivo unico, fino a quadri estremi di ciclopia e anoftalmia. La patologia è più spesso sporadica dovuta ad anomalie cromosomiche o mutazioni sporadiche, ma esistono 12
☤ Alessandro G. - 2011/2012
anche forme AD con genitori che presentano quadri molto lievi e difficilmente evidenziabili della malattia. Quindi lo stesso genotipo può avere espressione fenotipica diversa (espressività variabile).
La penetranza incompleta si riferisce al fatto che il genotipo malattia può non essere manifesto fenotipicamente. Penetranza del 100% significa che tutti i portatori del genotipo malattia hanno il fenotipo malattia. Un indice di penetranza incompleta in alberi genealogici è il salto di generazione. La spiegazione di questi due fenomeni risiede nel contesto genico dell'individuo, cioè la presenza di geni modificatori che influenzano il fenotipo.
Nell'oloprosencefalia il gene più frequentemente coinvolto è SHH (Sonic Hedgehog): 3,7% nei casi sporadici, 18 % nei casi familiari; meno frequentemente altri geni sullo stesso pathway di segnalazione (ZIC2: 5%, SIX3: 1%, TGIF: 1%, ecc). Interazioni differenti (più mutazioni sullo stesso pathway) possono influenzare il quadro della malattia (espressività variabile). Penetranza Incompleta ed Espressività Variabile possono occasionalmente essere osservate in difetti genetici ad ereditarietà recessiva (alterazione in omozigosi, più variabile perché coinvolge il bilanciamento dell’effetto di due alleli).
Malattie ad insorgenza tardiva, ed anticipazione
L'insorgenza tardiva è un caso particolare di penetranza incompleta: le malattie genetiche non necessariamente devono essere “congenite”. In alcune malattie, sebbene l’alterazione genetica sia presente fin dalla nascita, il quadro clinico può manifestarsi nell’età adulta. Il ritardo può essere dovuto a: • Lento accumulo di sostanze nocive • Incapacità di riparare certi danni ambientali • Morte rallentata di certi tessuti • Geni oncosoppressori nel modello a doppio hit • Cause sconosciute Il fenomeno dell'anticipazione è un caso di espressività variabile: descrive la tendenza da parte di alcune A : probabilità di un individuo che ha il gene
malattie genetiche a trasmissione autosomica dominante malattia di sviluppare i sintomi a quella età.
di divenire più gravi nelle generazioni successive. B : rischio di un figlio di un affetto di portare il gene
L’anticipazione può essere mascherata da variazioni malattia ad una determinata età.
casuali della severità; in molti casi può essere riferita ad errori della diagnosi. Il fenomeno dell’anticipazione è stato spiegato con l’instabilità di certi trinucleotidi repeat che possono espandersi in certe malattie (X­fragile, Distrofia Miotonica, Malattia di Hungtington). La severità e l’età di insorgenza in queste malattie correlano con la lunghezza del repeat, e questa tende ad espandersi attraverso le generazioni.
Corea di Hungtington (malattia da poliglutammine): malattia autosomica dominante, a penetranza completa ed insorgenza tardiva, presenta il fenomeno dell'anticipazione. Nel 1873 Huntington descrisse la “coreoatetosi” (korea = “danza”, athetosis = “senza posizione”), caratterizzata da demenza.
La tripletta ripetuta è CAG (che codifica per una glutammina) nella porzione 13
codificante del gene IT15 (cromosoma 4), per la hungtingtina, una proteina di 330kD. Un numero normale di ripetizioni è 9 ­ 32, sopra le 37 ripetizioni si manifesta la malattia. Da 32 a 37 triplette ripetute non provocano la malattia ma conferiscono un rischio alla prole. Il numero di CAG­repeats correla anche con l'età di insorgenza: • 40­50 triplette: esordio tardivo • 60­80 triplette: esordio giovanile IRREGOLARITÀ DELLA TRASMISSIONE AUTOSOMICA DOMINANTE
Non paternità
Tra le cause del riscontro di “anomalie” genetiche va sempre considerata la condizione (non conosciuta od occultata) di non paternità!
Mutazione de novo
Mutazione che avviene, durante la meiosi, a livello delle cellule gametiche parentali, o nelle prime divisioni dello zigote. Se avviene più tardi darà il quadro del Mosaicismo Somatico. Una mutazione post­zigotica produce un “mosaicismo” con due (o più) linee cellulari geneticamente distinte; il mosaicismo può colpire tessuti somatici o germinali.
Mosaicismo germinale
Una mutazione ad un gene causativo per una malattia dominante o X­linked, se colpisce la linea germinale, può essere ereditata. La malattia mima una ereditarietà recessiva: più figli affetti da genitori sani.
Si eseguirà lo studio molecolare di più tessuti:
• fibroblasti
• sperma
Acondroplasia: trasmissione AD. La maggior parte dei bambini nascono da genitori sani, quindi da mutazioni sporadiche, ma alcuni casi sono imputabili al mosaicismo germinale (coesistenza nel genitore di due o più linee cellulari geneticamente distinte). In questo caso la linea cellulare che ha dato origine alle cellule germinali porta la mutazione acondroplasica (FGFR­3), mentre il tessuto osteocartilagineo dell'individuo ha la variante normale del gene. Quindi, anche se il fenotipo è sano, la trasmissione segue il modello AD. Il rischio di mutazioni è proporzionale all'età paterna.
Il recettore FGFR3 è espresso ad alti livelli sulla membrana cellulare dei condrociti negli abbozzi cartilaginei delle ossa. Normalmente il legame con il ligando stimola la maturazione della cellula, facendone cessare la proliferazione. Le mutazioni di FGFR3 responsabili di acondroplasia fanno sì che il recettore sia sempre attivato, anche quando non c’è il suo ligando, compromettendo la normale maturazione delle ossa.
In omozigosi la mutazione è incompatibile con la vita:
◦ 25%: sano (AA)
◦ 50%: acondroplasico (Aa)
◦ 25%: abortito (aa)
14
☤ Alessandro G. - 2011/2012
Malattie oligogeniche:
1. Eredità digenica
I due geni coinvolti possono avere un effetto sinergico (necessarie le mutazioni di entrambi perché si manifesti il quadro) oppure le mutazioni del secondo gene coinvolto possono modificare l'espressione clinica del quadro. Alcuni casi di retinite pigmentosa necessitano di due mutazioni per manifestarsi: geni RDS e ROM1 (eredità digenica); in altri casi è sufficiente la sola mutazione di RDS. Nei casi di glaucoma la seconda mutazione modifica il quadro clinico ­ provoca un'insorgenza precoce della malattia. 2. Trasmissione triallelica
Sindrome di Bardet­Biedl (BBS): forma sindromica di retinite pigmentosa con varie associazioni (polidattilia, ipogonadismo, obesità, ritardo mentale, insufficienza renale). La malattia è geneticamente eterogenea: sono stati individuati 12 geni, denominati BBS. Di recente si è visto che gli alleli devono interagire tra di loro per provocare la malattia: la presenza di un terzo allele mutato, potrebbe suggerire il ruolo di “modificatore di penetranza”.
La maggioranza dei casi di BBS si trasmette con modalità AR classica, bbs1 / bbs1 BBS1 / bbs1 bbs4
però in più di 10% di casi di BBS la trasmissione è triallelica, cioè è necessario un terzo allele (di un altro locus) perché si manifesti. In altre parole, un individuo bbs1/bbs1 può non essere malato a meno che non bbs1 / bbs1
bbs4
abbia contemporaneamente anche un altro allele mutato (bbs4, ad esempio). Ereditarietà X­linked
I caratteri legati all'X presentano due fenomeni non canonici: la lyonizzazione e le traslocazioni X­autosomiche. • L'inattivazione del cromosoma X, detta anche lyonizzazione ҲX ҲY
ҲX XY
(questo fenomeno fu studiato da Mary Lyon), è volta a bilanciare il dosaggio genico: negli individui normali, sia i maschi che le femmine avranno un solo X attivo per cellula. Questo processo di compensazione di dose minimizza gli effetti di copie multiple di geni XY ҲҲ ҲY XY ҲX ҲY
legati all'X (“compensazione di dose”). Responsabile dell’inattivazione della X è il gene XIST, che viene espresso esclusivamente dal cromosoma X inattivo.
Se si osservano nuclei di cellule femminili normali (XX), si nota una massa di cromatina fortemente condensata, che non è presente nei nuclei delle cellule maschili normali (XY): si tratta del corpo di Barr, e rappresenta un cromosoma X altamente condensato e quindi inattivo. Il corpo di Barr è un cromosoma geneticamente inattivo. L’inattivazione avviene circa al 16° giorno dopo la fecondazione. Il cromosoma X che viene inattivato è scelto a caso tra i cromosomi X materno e paterno, secondo un processo indipendente da cellula a cellula: una volta che un cromosoma X è inattivato in una cellula, tutta la progenie eredita lo stesso tipo di inattivazione.
Dal momento che l'inattivazione è casuale, l'X rimanente può essere portatore del gene mutato, per cui la femmina diventa emizigote, e manifesta la malattia. Si ricorre al Test di Humara (HUMan Androgen­Receptor) per analizzare se l'inattivazione della X è casuale:
◦ MspI: metilasi­resistente
15
◦ HpaII: metilasi­sensibile
☞ Sito non metilato sull’X attivo → digestione HpaII → la PCR non amplifica
☞ Sito metilato sull’X inattivo → NO digestione HpaII → la PCR amplifica
•
Le traslocazioni X­autosomiche, anche se bilanciate, comportano la fissa inattivazione dell'X normale per bilanciare il dosaggio genico, dal momento che sull'X con la traslocazione c'è materiale genetico autosomico, che non deve essere inattivato, e viceversa sull'autosoma con la traslocazione c'è materiale dell'X. Anche se il punto di rottura della traslocazione interrompe una sequenza genica (ad esempio il gene della Distrofia Muscolare di Duchenne) la femmina avrà attivo solo l'X con l'interruzione, e quindi potrà manifestare anche malattie X­linked recessive altrimenti osservabili solo nei maschi (emizigoti).
L'espressione fenotipica di un carattere X­linked recessivo è possibile nei casi:
• ҲY + XҲ → ҲҲ; oppure ҲY; oppure Ҳ0 (sindrome di Turner)
• XY + XҲ → XҲ con lyonizzazione sbilanciata
La letalità maschile si può manifestare per certi caratteri X­linked dei quali la mancanza di un allele normale non è compatibile con la vita. La caratteristica degli alberi genealogici è di maschi abortiti e solo femmine affette. Sono esempi di malattie legate all'X con letalità maschile l'Incontinentia Pigmenti, la sindrome di Goltz (Focal Dermal Hypoplasia) e la sindrome di Rett. Incontinentia pigmenti: è una malattia X­linked dominante, letale nei maschi. Dà suscettibilità alle infezioni da micobatteri.
Si presenta con un eritema neonatale seguito da vesciche su tutto il corpo tranne la faccia. Le vesciche guariscono in qualche settimana. Residuano poi anomalie della pigmentazione. L'eritema compare allo stadio I (dalla prima settimana al 4° mese). Sono poi presenti ipo­/anodontia, microdontia, alopecia/stempiatura, alterazioni ungueali e retiniche.
La proteina IKBKG (NEMO) normalmente attiva NF­кB. Le cellule senza NF­
кB attivo sono più sensibili ai segnali pro­apoptotici.
Test genetico prenatale: • Ricerca della delezione
• Analisi di segregazione con marcatori microsatellitari del cromosoma X
Per alcune malattie legate all'X il quadro è più grave nelle femmine: nell'albero genealogico le femmine sono affette, mentre i maschi sono sani o con gradi lievi di malattia.
Un esempio di questo fenomeno è la displasia craniofrontonasale, una disregolazione dell'equilibrio della crescita: la caratteristica di questo tipo di eccezione alle regole 16
☤ Alessandro G. - 2011/2012
mendeliane è di essere trasmessa dal padre a tutte le figlie femmine, ma a nessun figlio maschio; le femmine vengono colpite più severamente dei maschi, che sono portatori. La spiegazione del fenomeno sta nel fatto che a volte non avere un gene funzionante è meglio che averne un 50% mutato che interferisce con il funzionamento del 50% normale: per cui gli eterozigoti sono più gravi degli omozigoti (o degli emizigoti: i maschi, in questo caso). Imprinting
Imprinting è un termine mutuato dall’etologia (K. Lorenz); l’imprinting genomico descrive differenze nell’espressione di alleli paterni e materni di certi geni autosomici nei mammiferi.
Per la grandissima maggioranza dei geni, l’espressione di un allele non dipende dall’origine materna o paterna dello stesso (1a legge di Mendel ed incrocio reciproco); tuttavia per alcuni geni l’espressione di un allele dipende dalla sua origine parentale. Questo fenomeno è conosciuto come imprinting genomico. Si possono allora avere delle malattie:
• Quando l’allele normalmente espresso porta una mutazione.
• In presenza di una disomia uniparentale. Se sono ereditate le 2 copie del cromosoma che porta l’allele inattivo, la funzione del gene sarà abolita.
Si sapeva che l’espressione di un dato gene può dipendere dal background genetico, o dall’influenza dell’ambiente, ma non si era mai pensato che essa potesse anche dipendere dall’origine parentale. Alcune osservazioni hanno fatto pensare a tutto questo:
• Embrioni di topo manipolati in modo da possedere una copia del genoma materno o paterno non si sviluppano, sebbene posseggano un numero diploide di cromosomi.
• Aborti umani triploidi sono fenotipicamente differenti, e questa differenza dipende dall’origine materna o paterna del genoma in più.
• Certi caratteri autosomici dominanti si manifestano solo quando ereditati dal padre o dalla madre.
• Delezioni di certe regioni cromosomiche causano un fenotipo differente, se presenti sul cromosoma ereditato dal padre o dalla madre.
• La perdita allelica di numerosi tumori spesso coinvolge l’allele paterno.
L’imprinting sembra agire a livello trascrizionale. Il meccanismo sembra coinvolgere la metilazione del DNA, ma i dettagli sono complessi e non ancora compresi fino in fondo. Potrebbe variare da tessuto a tessuto, ed anche durante lo sviluppo, suggerendo un livello differente del controllo dell’espressione genica.
Ci devono essere dei meccanismi capaci di distinguere tra gli alleli ereditati dal padre e quelli ereditati dalla madre: dato che un cromosoma passa attraverso la linea germinale maschile e femminile, esso deve acquisire un imprinting che “marchi” gli alleli in modo diverso 17
nell’organismo maschile o femminile in cui il gamete si sviluppa. Ci deve essere inoltre un meccanismo che cancella l’imprinting per esempio quando un uomo trasmette un allele, ereditato da sua madre, con imprinting materno.
Imprinting Materno → malattia trasmessa attraverso il padre
Imprinting Paterno → malattia trasmessa attraverso la madre
Disomia uniparentale: corredo cromosomico diploide normale (46XX o 46XY), ma con ineguale contributo paterno e/o materno.
Il caso estremo di disomia uniparentale è la diploidia uniparentale: tutti i cromosomi derivano da un singolo genitore. Una diploidia uniparentale comporta nell’uomo un mancato sviluppo embrionale. La “mola idatiforme” rappresenta uno zigote con apparente corredo cromosomico 46XX, che non sviluppa l’embrione. L’epitelio trofoblastico può trasformarsi in coriocarcinoma. Dipende da una diploidia parentale paterna. Il “teratoma ovarico” è rappresentato da una massa disorganizzata di tessuti embrionali senza presenza di annessi extra­embrionali. Dipende da una diploidia uniparentale materna. Più spesso i casi di disomia riguardano un singolo cromosoma, e si ha allora la disomia uniparentale, che può essere:
• Eterodisomia: una copia dei 2 cromosomi paterni o materni •
Isodisomia: 2 copie dello stesso cromosoma paterno o materno La disomia uniparentale può causare malattia con diversi meccanismi: • Caratteri soggetti ad imprinting
• Due copie di un allele alterato (AR) La disomia può accadere in diversi scenari: • Embrione trisomico per un cromosoma, che espelle un cromosoma di essi per restaurare la diploidia • Pressione selettiva su un embrione monosomico, che duplica il cromosoma unico per ristabilire il corredo diploide.
Malattie dovute a disomia uniparentale o da imprinting:
• 7 materno → Ritardo della crescita
• 11 paterno → sindrome di Beckwith­Wiedenman
• 15 materno → Sindrome Prader­Willi
• 15 paterno → Sindrome di Angelman
S. di Angelman
18
S. di Prader-Willi
☤ Alessandro G. - 2011/2012
Sindrome di Angelman: grave ritardo mentale, assenza della parola, microbrachicefalia, prognazia, bocca larga con lingua protrudente, denti spaziati, capelli biondi (65%), atassia e movimenti stereotipati, crisi epilettiche, tipico pattern all'EEG. Sindrome di Prader­Willi: ampia variabilità di espressione e severità; normale altezza alla nascita che diminuisce già a partire dai primi due mesi di vita, obesità, ritardo mentale (63% ritardo mentale lieve, 31% ritardo mentale medio, 6% ritardo mentale grave), rima palpebrale rivolta verso il basso, strabismo, ipotonia alla nascita, problemi comportamentali soprattutto riguardo al cibo (eccessivo appetito, mancato senso di sazietà), ipogonadismo.
Nel 70% dei casi circa, in entrambe le sindromi si riscontra una delezione 15q11.1. La ragione è che in quel locus ci sono due geni soggetti ad imprinting: uno ad imprinting materno e l'altro ad imprinting paterno (espressione/soppressione allelica in base all'origine parentale materna o paterna del gene). Questi due geni hanno pertanto un'espressione monoallelica. La malattia quindi si può avere quando: ◦ l'allele non imprintato manca (delezione: 70%), o è mutato (0 ­ 10%)
◦ due copie dello stesso cromosoma sono ereditate dallo stesso genitore (disomia uniparentale): 30%
◦ difetto di imprinting per alterazioni del Centro di Imprinting (IC): 2 ­ 5%
In breve quindi, la presenza della sindrome di Angelman o della sindrome di Prader­Willi dipende dall'origine del cromosoma che ha subito la delezione 15q11.1: nel primo caso manca il contributo materno della regione 15q11.1 mentre la stessa regione sul cromosoma paterno è inattivata.
•
DNA mitocondriale ed Eredità matrilineare
Poliplasmia: in ogni cellula (eccetto piastrine e ovulo non fertilizzato) sono presenti molti mitocondri, ed ogni mitocondrio contiene multiple copie del suo genoma, pertanto esistono migliaia di copie di mtDNA per cellula. Durante la divisione cellulare i 19
•
•
•
•
mitocondri vengono distribuiti casualmente alle cellule figlie e quindi la genetica mitocondriale è più simile alla genetica di popolazione che alla genetica mendeliana.
Eteroplasmia: nei tessuti normali tutte le copie di mtDNA sono identiche (omoplasmia). Nel caso di una mutazione del mtDNA, questa può colpire tutte le copie, oppure essere presente solo in una percentuale di genomi (eteroplasmia). Generalmente i polimorfismi neutrali sono omoplasmici, mentre la maggior parte delle mutazioni­
malattia sono eteroplasmiche: l'eteroplasmia è pertanto cellulare o mitocondriale?
Effetto soglia: l’espressione clinica delle mutazioni del mtDNA è determinata dalla relativa proporzione “wild type / mutato” in un determinato tessuto; è necessario un numero minimo di copie per danneggiare il metabolismo energetico di un determinato organo o tessuto (SNC, cuore, muscolo, rene, ghiandole esocrine), principalmente in base al bilancio energetico (valore relativo, e non assoluto).
Segregazione mitotica: durante la divisione cellulare, la proporzione di genomi mutati può variare, per deriva nelle cellule figlie, con conseguente cambiamento fenotipico.
Eredità materna: virtualmente tutti i mitocondri dello zigote derivano dall’oocita, e perciò la modalità di trasmissione delle mutazioni mitocondriali differisce dalla trasmissione mendeliana classica:
◦ madre portatrice → trasmissione a tutta la progenie (ma solo le figlie femmine possono ritrasmettere la mutazione ai loro figli)
◦ eteroplasmia + effetto dose → eccezioni fenotipiche all’eredità matrilineare
I mitocondri sono presenti in tutti i tessuti, quindi le malattie mitocondriali possono colpire qualsiasi organo.
Sostituzioni nucleotidiche: generalmente associate a patologie neurologiche ed oftalmologiche.
• Leber’s Hereditary Optic Neuropathy (LHON)
Cecità ad insorgenza tardiva dovuta alla morte del nervo ottico. È generalmente determinata da un cambio Arg­His (np 11778) nel gene ND4 presente in omoplasmia nella maggioranza dei pazienti, ma può essere determinata da numerose altre mutazioni a carico dei complessi per il trasporto degli elettroni, talora anche in combinazione: la probabilità di cecità aumenta negli individui con mutazioni più gravi o nella combinazione di mutazioni diverse.
Rischio di sviluppare LHON nei portatori di mutazione:
◦ 30 ­ 50% nei maschi
20
☤ Alessandro G. - 2011/2012
•
◦ 8 ­ 28% nelle femmine
Neurogenic muscle weakness, Ataxia and Retinite Pigmentosa (NARP)
Retinite pigmentosa, atassia, convulsioni, demenza, debolezza muscoli prossimali di origine neurogena, neuropatia sensitiva e ritardo nello sviluppo. È determinata da un cambio Leu­Arg (np 8993) nell’ATPasi6; tale mutazione è sempre presente in eteroplasmia e la gravità dei sintomi è correlata alla percentuale del DNA mutante.
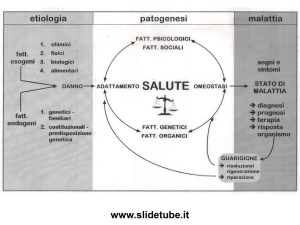
L'EPIGENETICA
L'insieme di quei fattori esterni ai geni/cromosomi, ma che, agendo senza modificarli, ne influenzano l'espressione.
21
III. GENOMICA E POSTGENOMICA
STRATEGIE PER L'IDENTIFICAZIONE DI GENI MALATTIA Esistono quattro metodi per identificare un gene patologico: 1. Clonaggio funzionale: alcune informazioni sulla funzione del gene vengono sfruttate per isolare un clone del gene; se le basi biochimiche della patogenesi sono conosciute (è noto il prodotto genico), questo può essere purificato e sequenziato in amminoacidi. Perciò sarà possibile identificare il gene con alcuni accorgimenti tecnici. Alternativamente, può essere usato un saggio funzionale per controllare la presenza del gene. Questo approccio è stato utile solo in pochi casi.
2. Gene candidato: si può sospettare che un gene sia responsabile di una patologia umana senza sapere niente della sua localizzazione cromosomica. Ciò può avvenire se un certo fenotipo assomiglia a un altro fenotipo, in animali o esseri umani, per il quale si conosca il gene responsabile, o se la patogenesi molecolare suggerisce che il gene possa essere membro di una famiglia genica nota. Tali approcci hanno avuto successo solo raramente e sono stati superati dalle strategie con il gene candidato di cui si conosce la localizzazione.
3. Clonaggio posizionale: si isola il gene conoscendo solo la sua localizzazione subcromosomica, senza utilizzare alcuna informazione riguardante la patogenesi o la funzione biochimica. Conoscendo la posizione cromosomica del locus si costruisce la mappa fisica e la mappa genetica della regione, ed il gene malattia viene identificato dal ristretto numero di trascritti di questa regione per la presenza di mutazioni. Il clonaggio posizionale rimane arduo e sta diventando sempre meno necessario per l’accumularsi di informazioni che permettono un approccio posizionale al gene candidato.
Geni e patologie identificati con Clonaggio posizionale:
1986: Distrofia muscolare di Duchenne, Retinoblastoma
1989: Fibrosi cistica
1990: Neurofibromatosi di tipo 1, Tumore di Wilms
1991: Aniridia, Poliposi familiare del colon, Sindrome dell’ X fragile, Distrofia miotonica
1993: Malattia di Huntington, Sclerosi tuberosa, Malattia di von Hippel­Lindau
1994: Acondroplasia, Cancro mammario/ovarico a esordio precoce, Malattia del rene policistico
1995: Atrofia muscolo spinale
4. Gene candidato per posizione: è il metodo più usato. Una volta che una patologia sia stata mappata, sta diventando possibile utilizzare sempre più spesso la ricerca nelle banche dati per identificare i geni candidati. Con il continuo aumento del numero di geni umani mappati in specifiche regioni subcromosomiche questi approcci posizionali al gene candidato sono pronti a dominare il campo. La regione cromosomica del locus viene identificata tramite l'analisi del linkage (meiosi informative!) studiando una famiglia grande o più famiglie piccole con la malattia. Tutti gli individui della famiglia vengono tipizzati per più di 400 marcatori polimorfici (microsatelliti a più alleli) sparsi in tutto il genoma a distanza media di 10cM l'uno dall'altro; questa tappa è detta Wide Genome Scan (per tutti i cromosomi sono state create mappe genetiche di marcatori polimorfici). Per tutti i 400 polimorfismi viene fatta l'analisi del linkage rispetto al locus malattia; così si mette in evidenza la concatenazione della malattia ad alcuni marcatori la cui posizione cromosomica è nota: viene identificata così la regione critica che porta il locus malattia. Con la ricostruzione di aplotipi della famiglia la regione critica viene ulteriormente ristretta (definizione della regione critica) e vengono 22
☤ Alessandro G. - 2011/2012
studiati i trascritti dei geni della regione alla ricerca di mutazioni per identificare il gene responsabile.
Marcatori molecolari:
• RFLPs (Restriction Fragment Length Polymorphisms): presenza/assenza del sito di taglio per un enzima di restrizione → Biallelici
Southern blot/PCR
• Microsatelliti: ripetizione in tandem di 2/3/4 nucleotidi (CA) n → Molti alleli, molto informativi
Distribuiti in modo uniforme nel genoma (ogni ~ 100 Kb)
PCR/marcatura con fluorescenza
• SNPs (Single Nucleotyde Polymorphisms): differenze di singola base, non necessariamente riconosciuti da enzimi di restrizione → Biallelici
Molto frequenti
Sviluppo di tecniche di genotyping automatizzate e in larga scala
Marcatori genetici: polimorfici → presenza di 2 o più alleli alternativi.
L'allele più raro ha frequenza di almeno 1% nella popolazione. Facilmente tipizzabili e stabili di generazione in generazione. Posizione nota nel genoma.
L’approccio statistico alla mappatura dei caratteri mendeliani consiste nel calcolare mediante opportuni software le probabilità che i loci analizzati in un albero genealogico:
• siano concatenati, cioè in linkage (L1)
• siano indipendenti (L0)
Il rapporto tra le due probabilità depone a favore o contro il linkage: se è in favore del linkage, la frazione di ricombinazione più probabile è quella che dà il rapporto più favorevole.
La significatività statistica (p > 0.05) si raggiunge quando il logaritmo del rapporto tra le probabilità (LOD score) è uguale o maggiore di 3.
Multipoint Lod Score (MLS): spesso i dati ricavati da una solo famiglia non sono sufficienti per stabilire presenza /assenza di linkage. I lod score ottenuti da famiglie indipendenti (per lo stesso valore di θ) si possono sommare fra loro. Il valore di θ per cui il LOD è massimo è la stima più probabile della frazione di ricombinazione.
Data una mappa di markers con posizione nota, si calcola la likelihood per ogni posizione del locus malattia lungo il cromosoma. Permette di estrarre il massimo dell’informazione data da tutti i markers sul cromosoma.
Identificazione di un gene e correlazione con uno specifico fenotipo
La correlazione gene­malattia può essere confermata con diverse metodiche: • Ricerca di mutazioni causative, all’interno di un gene candidato, per una specifica malattia genetica, in un gruppo di pazienti.
• Riacquisizione del fenotipo normale in fibroblasti in vitro mediante transfezione con il gene candidato. • Ricostruzione della malattia in modelli animali.
Costruzione di mappe genetiche di riferimento
Famiglie CEPH: costituiscono un pannello di 40 famiglie di riferimento, selezionate per la loro struttura ideale per l’analisi di linkage (3 generazioni, con i 4 nonni, 2 genitori, e almeno 6 figli).
23
FARMACOGENETICA E FARMACOGENOMICA
La farmacogenetica studia l'influenza dei fattori genetici ereditari sulla risposta ai farmaci a livello cellulare, tissutale, di individuo e di popolazione.
La farmacogenomica consiste nella determinazione e nell’analisi del genoma e dei suoi prodotti allo scopo di:
• correlare queste informazioni con la risposta al farmaco presente a livello cellulare, tessutale, di individuo o di popolazione
• individuare nuovi bersagli terapeutici
• sviluppare nuovi farmaci mirati alla malattia o all'individuo.
A livello di popolazione esiste una grande variabilità nell'efficacia dei farmaci, negli effetti collaterali e nelle reazioni avverse (normalmente aumentano all'aumentare della dose del farmaco).
Si riscontrano nella popolazione molti individui con scarsa risposta ai farmaci più usati: • Anti­ANG.II: 10 ­ 25% • ACE­inibitori: 10 ­ 25% • β­bloccanti: 15 ­ 25% • Antidepressivi triciclici: 20 ­ 50% • Agonisti β2­adrenergici: 40 ­ 70% La stessa grande variabilità si riscontra a livello degli effetti collaterali, che possono diventare inaccettabili, insorgendo, in alcuni individui, già a piccole dosi del farmaco. Il 2 ­ 14% dei pazienti ospedalizzati vengono ricoverati a causa di ADR (Adverse Drug Reactions); si stima che il 5 ­ 20% dei pazienti ospedalizzati soffra di ADR, che causano fino al 3% dei decessi nella popolazione e fino al 6% dei decessi ospedalieri. Le reazioni avverse ai farmaci sono la quarta causa di morte dopo malattie cardiovascolari, tumori e ictus.
La risposta a farmaci è governata da diversi fattori tra cui: • Fattori fisiologici: età, sesso, peso corporeo • Fattori patologici: malattie, funzionalità epatica e quella renale • Ambientali: dieta, alcool, tabacco, uso di altri farmaci • Fattori genetici: polimorfismi (sopratutto SNPs) L'attività di un farmaco è influenzata da due meccanismi: 1. Farmacocinetica: fattori che influenzano la concentrazione ematica del farmaco (“Cosa fa il corpo con il farmaco”), i cosiddetti A­D­M­E:
◦ Assorbimento ◦ Distribuzione ◦ Metabolismo: conversione in metaboliti inattivi o attivazione di pre­farmaci ◦ Escrezione 2. Farmacodinamica: interazioni tra il farmaco ed il suo bersaglio molecolare (“Cosa fa il farmaco al corpo”). Ci sono polimorfismi genici che alterano la farmacocinetica (geni codificanti per trasportatori, enzimi del metabolismo), ed altri che agiscono sulla farmacodinamica (geni di recettori, di canali ionici, di enzimi, di proteine regolatrici):
1. Polimorfismi farmacocinetici (del metabolismo): i farmaci sono generalmente lipofili, per facilitarne l'assorbimento, e quindi per essere eliminabili necessitano di un processo (nel fegato) che li renda idrosolubili. Questo processo di detossificazione è 24
☤ Alessandro G. - 2011/2012
Concentrazione
ematica del farmaco
svolto in due passaggi successivi: ◦ Reazioni di fase I: trasformazione dei gruppi funzionali della molecola del farmaco mediante ossidazione, riduzione, idrolisi. Il 75% delle reazioni di questo tipo sono svolte dal Citocromo P­450 e dai suoi componenti: ▪ CYP2D6: metabolizza il 25­30% dei farmaci più comuni (beta­bloccanti, antiaritmici, antidepressivi triciclici, neurolettici, antitussivi, narcotici)
▪ CYP2C19: diazepam, imipramina, omeprazolo
▪ CYP2C9: losartan (anti­ipertensivo), fenitoina (antiepilettico), warfarin, glipizide (antidiabetico)
◦ Reazioni di fase II: rendono il metabolita solubile, mediante reazioni di coniugazione (transferasi). I polimorfismi più importanti interessano: ▪ NAT2 (N­acetil­transferasi 2): metabolizza i cancerogeni del tabacco, i sulfonamidici, la caffeina ▪ COMT (catecol­O­metiltransferasi): catecolamine ▪ TPMT (tiopurin­metiltransferasi): chemioterapici impiegati nella terapia di Leucemie Linfoblastiche Acute ▪ UGT (UDP­glucuronato­transferasi): cancerogeni vari I polimorfismi di questi geni cambiano l'emivita ed i livelli sierici del farmaco (dose effettiva, durata di azione terapeutica del farmaco, reazioni avverse e tossicità), spiegando la differente risposta dell'individuo. 0
0
0
tempo
tempo
Metabolismo
lento
tempo
Metabolismo
rapido
Metabolismo
normale
Identificazione del fenotipo metabolico:
[ Farmaco ]
Farmaco sonda ( probe drug) ⇒
(Rapporto Metabolico)
[ Metabolita ]
Distribuzione normale
popolazione
polimorfica
popolazione
polimorfica
rapporto metabolico
Veloce
Metabolismo
normale
lento
Polimorfismo di CYP2D6 (Fase I)
La CYP2D6 metabolizza circa il 25% dei comuni farmaci, tra cui β­bloccanti, antiaritmici, antidepressivi triciclici, neurolettici, narcotici. Salvo la codeina, 25
tutti gli altri vengono inattivati dal citocromo (la codeina viene trasformata nel suo principio attivo: la morfina). Circa il 5­10% della popolazione appartiene al gruppo polimorfico dei Poor Metabolizers (PM), mentre un altro 5­10% sono Ultrarapid Metabolizers (UM).
Metabolismo della sparteina in base al suo rapporto metabolico
UM = Ultrarapid metabolizer (metabolismo ultra­rapido): 5 ­ 10%
EM = Extensive metabolizer (metabolismo normale): 65 ­ 80% IM = Intermediate metabolizer (metabolismo intermedio): 10­15%
PM = Poor metabolizer (metabolismo lento): 5­10%
Distribuzione etnica dei fenotipi metabolici di CYP2D6:
◦ Metabolismo lento
▪ Europei: 5 ­ 10 %
▪ Africani: 2 ­ 5 %
▪ Orientali: <1 %
◦ Metabolismo ultra­rapido
▪ Scandinavi: 1,5 %
▪ Spagnoli: 7 %
▪ Etiopi : 20 %
Il locus 22q13.1 ha più di 75 varianti alleliche: quasi tutti i polimorfismi nel gene CYP2D6 sono SNPs (che possono dare un enzima instabile o con alterazioni dell'affinità); le delezioni causano una assenza dell'enzima, uno splicing defettivo dà un enzima alterato, le ripetizioni (fino a 13 copie del gene) un aumento della quantità di enzima. Il fenotipo correla strettamente con il genotipo: si è visto che i PM non hanno nessuna copia del gene, mentre gli UM ne hanno 3 o più copie.
La concentrazione sierica del farmaco a sua volta è inversamente proporzionale al numero di copie del gene, per cui i PM subiscono più facilmente gli effetti tossici del farmaco (cardiotossicità, tachicardia, costipazione, debolezza per gli antidepressivi triciclici; parestesie, disturbi visivi, vertigine, nausea, vomito, aritmie per gli antiaritmici), mentre per gli UM il farmaco non è efficace alle dosi abituali. Con la codeina il discorso è speculare: i PM non beneficiano dal farmaco, mentre gli UM sperimentano euforia, nausea ed altri effetti collaterali. Polimorfismo della TPMT (Fase II)
Le tiopurine sono analoghi delle purine; inibiscono la sintesi del DNA e bloccano la divisione cellulare. Vengono metabolizzate in nucleotidi della tioguanina, metaboliti attivi incorporati nel DNA, causando arresto del ciclo cellulare ed effetto terapeutico antileucemico e citotossico.
Mercaptopurina e tioguanina sono chemioterapici per la Leucemia Linfoblastica Acuta; l'azatriopina è un immunosoppressore per trapianti e per trattare 26
☤ Alessandro G. - 2011/2012
malattie autoimmuni.
Le tiopurine sono caratterizzate da un indice terapeutico (rapporto fra dose tossica e dose terapeutica) ristretto; in alcuni pazienti, le tiopurine provocano mielosoppressione con conseguente leucopenia e trombocitopenia, che può essere letale: questi pazienti devono essere trattati con dosi di tiopurine 5 ­ 10% delle dosi standard.
Delle TPMT esistono 21 varianti alleliche con attività enzimatica ridotta: la 3A è la variante più frequente nella popolazione europea, la 3C la più frequente nella popolazione africana e asiatica; entrambe hanno attività enzimatica ridotta all'1%. Lo 0,3% della popolazione è omozigote per la mutazione ed ha attività enzimatica assente; l'11,1% è eterozigote, con attività intermedia; l'88,6% presenta gli alleli wild type ad attività elevata.
2. Polimorfismi farmacodinamici (del target): l'inefficacia della risposta, nel 40­70% dei casi, si osserva per i farmaci β2­agonisti impiegati nel trattamento delle crisi asmatiche in quanto broncodilatatori. La scarsa risposta è in parte dovuta alla desensibilizzazione dei recettori (down­regulation), come risultato di un trattamento protratto, ed in parte è da attribuire ai polimorfismi del gene del recettore (4 SNPs biologicamente attivi nella regione codificante ed 1 SNP nella regione promotore 5'). Gli studi in vitro con cellule transfettate hanno rivelato che i polimorfismi della regione codificante riducono la risposta al farmaco stimolando la down­regulation del recettore, mentre il polimorfismo del promotore innalza l'efficacia del farmaco. Questi risultati però non hanno avuto conferma in vivo.
Ultimamente si è visto che per predire la risposta in vivo non sono tanto importanti i singoli SNPs, quanto l'aplotipo dell'individuo (l'insieme di più SNPs ereditati in blocco).
Polimorfismo del recettore β2­adrenergico
La variabilità individuale nell'efficacia e nella durata di azione dei farmaci broncodilatatori (agonisti del recettore adrenergico β2) presenta un 40 ­ 70% dei pazienti che hanno una scarsa risposta, dovuta, in parte, alla desensibilizzazione del paziente al farmaco nel tempo (causata dalla riduzione del numero di recettori β2­adrenergici).
Si conoscono 9 SNPs nella regione codificante del gene (5 silenti, 4 con sostituzione aminoacidica in 16, 27, 34 e 164) e più di 10 SNPs nella regione a monte del gene (1 in una sequenza che codifica il peptide BUP ­ Beta Upstream Peptide, di 19aa, che regola l’espressione del recettore β2).
◦ Effetto farmacologico del polimorfismo Arg164Ile del recettore β2AR: riduzione nell’attivazione dell’enzima adenilato ciclasi
◦ Effetto farmacologico del polimorfismo Arg19Cys del peptide regolatore BUP: aumento nel numero di recettori β2­adrenergici L’attività complessiva di un gene riflette la somma degli effetti di tutte le 27
variazioni: la correlazione tra l’attività di un gene e la risposta a un farmaco è più forte per gli aplotipi che per le singole varianti.
Aplotipi del recettore β2­adrenergico:
◦ 13 SNPs → 213 (ovvero 8'192) possibili aplotipi
◦ 12 aplotipi sono presenti nella popolazione
Gli aplotipi del recettore β2­AR sono predittivi della risposta broncodilatatoria in vivo.
Complessità poligenica
La risposta al farmaco non è un tratto monogenico, ma risulta dalla interazione tra diversi fattori genetici e fattori ambientali:
• genotipo farmacocinetico: enzimi del metabolismo, trasportatori
• genotipo farmacodinamico: bersaglio del farmaco
• fattori ambientali
• altri geni?
L'AmpliChip CYP450 Test è il primo test farmacogenetico approvato dal FDA (Foods and Drugs Administration) per uso diagnostico, al fine di prevedere il metabolismo di un farmaco.
Esegue un microarray per identificare polimorfismi nei geni del metabolismo: 27 varianti del CYP2D6, 2 varianti del CYP2C19.
Sono anche nati algoritmi per la terapia personalizzata con Warfarin basata sul genotipo del paziente (CYP2C9: metabolismo epatico ­ farmacocinetica; VKORC1: vit.K­epossido­reduttasi ­ farmacodinamica).
Scopo della farmacogenomica quindi è, partendo da soggetti con la stessa diagnosi, trattare con farmaci e dosi standard i “genotipi favorevoli” (responders e soggetti non predisposti a tossicità), trattare con dosi o farmaci alternativi i “genotipi non favorevoli” (non­responders e soggetti predisposti a tossicità). Essa permette di attuare una Medicina predittiva (prevedere la risposta dell'individuo al farmaco in base al profilo genetico) e una Medicina personalizzata (terapia basata sul profilo genetico di ogni individuo), rispondendo al motto “il farmaco giusto alla dose giusta al paziente giusto”.
28
☤ Alessandro G. - 2011/2012
IV. CITOGENETICA MEDICA
LA PATOLOGIA CROMOSOMICA
Lo studio citogenetico del cariotipo è il metodo classico per identificare grossolane aberrazioni cromosomiche. Un prelievo di sangue viene cresciuto in un terreno di coltura, poi le cellule vengono bloccate in metafase con la colchicina, dopodiché i cromosomi vengono liberati dalle membrane usando una soluzione ipotonica, vengono fissati con metanolo, lavati e colorati con diversi metodi (a bande G; a bande R, negativo delle bande G; a bande Q) in modo di renderli visibili. A partire dai cromosomi sparsi viene costruito il cariogramma. L'analisi del cariotipo mette in evidenza alterazioni di dimensioni maggiori a 5Mb. La gravità delle anomalie cromosomiche è correlata al tipo di cromosoma e alla quantità di geni interessati. Le anomalie cromosomiche possono essere classificate in base a diversi aspetti: • Bilanciate: di solito senza anomalie fenotipiche • Sbilanciate: di solito si manifestano con ritardo mentale ed anomalie di sviluppo (la gravità è spesso proporzionale all'entità di sbilanciamento) In base alla natura della traslocazione: • Traslocazioni robertsoniane: tra cromosomi acrocentrici (13, 14, 15, 21, 22); i due cromosomi coinvolti si fondono a livello del centromero perdendo i bracci corti (senza significato clinico per il soggetto ma rischio per la prole). • Traslocazioni reciproche: mediante punti di rottura; di solito il fenotipo non presenta anomalie, a meno che il punto di rottura non interrompa una sequenza genica (trasmissione AD). In base al coinvolgimento delle linee cellulari le anomalie sono distinte in: • Anomalie costituzionali: presenti in tutte le cellule del corpo. • Anomalie somatiche: presenti in alcune linee cellulari (mosaicismo) In base alla natura dell'anomalia distinguiamo: • Anomalie di numero: ◦ Aneuploidie: le trisomie e le monosomie. Le cause dell'aneuploidia possono essere due: ▪ Non­disgiunzione ▪ Ritardo nella migrazione cromosomica in anafase La frequenza delle anomalie cromosomiche è direttamente proporzionale all'età della madre e inversamente proporzionale all'età gestazionale. Frequenza globale alla nascita: 0,65%.
Le anomalie più frequenti: ▪ + 21 → sindrome di Down: 0,12% (1 su 833) ▪ + 18 → sindrome di Edward: 0,013% 29
•
30
▪ + 13 → sindrome di Patau: 0,004% Nelle anomalie bilanciate il problema non si pone per il portatore (a meno che il punto di rottura non interrompa una sequenza genica) ma per la sua prole. Infatti una traslocazione bilanciata è sospettata tipicamente quando in una famiglia nascono 2 figli con ritardo mentale e anomalie di sviluppo che sono fenotipicamente diverse. La non­disgiunzione è l'incapacità di separarsi, dei cromosomi appaiati, durante l'anafase della meiosi I, oppure dei cromatidi fratelli, durante l'anafase della meiosi II. Essa dà origine a gameti con 24 cromosomi, che dopo la fecondazione con gamete normale (n = 23) produce uno zigote trisomico (2n+1 = 47). I fattori che influenzano la non­disgiunzione non sono noti, ma si sa che il fenomeno avviene più frequentemente nella meiosi I materna. Quando il cariotipo non porta alla diagnosi dell'anomalia, la citogenetica ricorre alle tecniche di biologia molecolare, tipicamente la FISH (Fluorescent In Situ Hybridization), con la quale si riescono ad identificare riarrangiamenti piccoli nascosti alla citogenetica classica. La sonda nucleotidica marcata con fluorescina si combina con la sequenza complementare. Ci sono molte sonde diverse e specifiche con le quali possiamo studiare segmenti cromosomici a noi noti: ad esempio l'alterazione cromosomica della sindrome di Williams (delezione di una regione del cr. 7) che non è evidenziabile con il cariotipo, è agevolmente diagnosticata con FISH impiegando sonde specifiche per la regione deleta. Altra metodica per identificazione di aneuploidie che si basa sul FISH utilizza marcatori per le regioni subtelomeriche (ricche di CpG con alta densità genica spesso interessate in queste patologie) per l'inquadramento di ritardi mentali sindromici non evidenziabili con il cariotipo classico. Questi riarrangiamenti criptici subtelomerici incidono per il 7­8% dei ritardi mentali sindromici non diagnosticati. Un'altra metodica ancora è l'Array­CGH (Comparative Genomic Hybridization) è capace di identificare riarrangiamenti in tutto il genoma. Frammenti marcati del genoma del probando vengono fatti ibridizzare su un supporto di segmenti genomici. L'intensità di segnale da un certo tratto del genoma può essere anormale indicando delezioni o duplicazioni di quel tratto. Questo metodo riesce a definire riarrangiamenti piccoli (più piccoli sono i frammenti del supporto, più alta è la risoluzione della tecnica) nel circa 20% dei ritardi mentali non diagnosticati. ◦ Poliploidie: triploidia, tetraploidia ◦ Mixoploidie: anomalie diverse in linee cellulari differenti Anomalie di struttura: ◦ Traslocazioni: reciproche, robertsoniane ◦ Inversioni: ▪ Paracentriche: sullo stesso braccio cromosomico ▪ Pericentriche: coinvolgono il centromero. ◦ Delezioni ◦ Duplicazioni ☤ Alessandro G. - 2011/2012
V. GENETICA CLINICA
LA DIAGNOSI DI LABORATORIO DI MALATTIE GENETICHE
dalla “probabilità” al dato genetico
Test genetici: il test genetico è definito come analisi del DNA, RNA, proteine o metaboliti per rilevare, a fini clinici, genotipi, mutazioni, fenotipi o cariotipi correlati a malattie ereditarie. Le indicazione all'esecuzione di un test genetico sono: • Conferma di diagnosi di una malattia • Identificazione di portatori sani di una malattia ereditaria • Diagnosi prenatale di malattie ereditarie • Diagnosi presintomatica di malattie ereditarie ad insorgenza tardiva suscettibili a trattamento • Ricerca della suscettibilità di ammalarsi di malattie complesse Tipi di test genetico:
• Test diagnostico: in presenza di un sospetto clinico di una malattia, permette di confermarne o precisarne la diagnosi
• Test presintomatico: in una famiglia con un difetto genetico noto, permette di identificare i portatori prima dell’esordio della malattia conclamata (malattie a penetranza completa con insorgenza tardiva)
• Test predittivo: in una famiglia con un difetto genetico noto che conferisce suscettibilità a una malattia, permette di identificare i soggetti a rischio
• Test prenatale: fornisce informazioni sullo stato genetico del nascituro
Ogni test genetico che non ha una sensibilità del 100% presenta un rischio residuo (RR), ovvero la possibilità di falsi negativi: se la sensibilità è del 75, il 25% delle mutazioni non sarà evidenziato. Pertanto, in chi risulti negativo ad un test, il Rischio di Popolazione sarà moltiplicato per il Rischio Residuo.
Diagnosi molecolare:
• Diretta: la sequenza del gene è nota, per cui si possono confrontare gli alleli del paziente con la sequenza normale per evidenziare eventuali variazioni
• Indiretta: la sequenza del gene non è nota, ma è nota la posizione cromosomica del gene; si utilizzano marcatori del DNA di quella regione e si studiano molti membri della famiglia per identificare il tratto di genoma che contiene il gene mutato
( = Analisi di linkage )
Carattere autosomico dominante: Sindrome di Cowden
Criteri patognomonici
• Malattia di Lhermitte­Duclos (gangliocitoma displastico del cervelletto) • Lesioni Mucocutanee: ◦ trichilemmomi
◦ cheratosi acrale ◦ papillomi cutanei ◦ papillomi mucosi
Criteri Maggiori
• Carcinoma mammario • Carcinoma tiroideo (non­midollare)
31
• Macrocefalia (circonferenza cranica > 97° percentile) • Carcinoma endometriale
Criteri minori: Lesioni tiroidee benigne, ritardo mentale, polipi amartomatosi intestinali, Mastopatia fibrocistica, Lipomi, Fibromi, Tumori Urogenitali (soprattutto carcinoma renale), malformazioni urogenitali, fibromatosi uterina
Analisi di sequenza del gene PTEN → 303delA (L112X)
Carattere autosomico recessivo: Fibrosi Cistica
> 1000 mutazioni descritte
Test genetico per Fibrosi Cistica:
• Screening delle mutazioni più frequenti: sensibilità 70­80%
• Analisi di sequenza dell’intero gene: sensibilità fino a 98.7%
Si può ridurre il rischio residuo ma non si può escludere completamente la presenza di mutazioni.
Rischio di Fibrosi Cistica nella prole: coppia senza storia familiare, non testata: 1/3'000
→ Test genetico (con sensibilità del 70%):
• Negativo in entrambi: 1/40'000
• Positivo solo in uno: 1/370
• Positivo in entrambi: 1/4 → Diagnosi prenatale (ricerca specifica delle mutazioni dei genitori)
Estensione analisi (tempi lunghi, eseguibile in pochi centri, sensibilità comunque incompleta).
Test genetico prenatale:
• Specifico: ricerca delle mutazioni identificate nei genitori → sensibilità completa
• Test standard su CVS o amniocentesi → sensibilità incompleta
CONSULENZA GENETICA La consulenza genetica è il «processo di comunicazione che affronta i problemi umani associati all'insorgenza, o al rischio di insorgenza, di un disordine genetico nella famiglia». La consulenza può servire a:
• Diagnosi di patologie genetiche
• Indicazioni di specifici esami citogenetici o molecolari da effettuare
• Calcolo del rischio eventuale per altre gravidanze
La maggioranza delle informazioni mediche è rivolta al singolo individuo e fornisce indicazioni standard; le informazioni genetiche sono invece rivolte alla famiglia intera.
Gli obiettivi della consulenza sono spiegare alla persona la malattia diagnosticata, la sua patogenesi e definire il rischio in modo che la persona possa formulare la sua scelta liberamente basandosi sulla conoscenza fornita, mettendo il paziente in condizione di compiere scelte informate e consapevoli. La consulenza aiuta a:
• Comprendere gli aspetti medici, comprese la diagnosi e la probabile evoluzione del disordine, così come le sue possibilità di controllo
• Comprendere il modo in cui l’eredità contribuisce al disordine e il rischio di ricorrenza nei familiari
• Comprendere le opzioni per affrontare il rischio di ricorrenza • Scegliere i comportamenti che sembrano più appropriati in considerazione del rischio e degli obiettivi della famiglia, agire quindi in armonia con tali decisioni
• Raggiungere il migliore adattamento possibile al disordine nel familiare affetto, al 32
☤ Alessandro G. - 2011/2012
rischio di ricorrenza nel non affetto, oppure entrambe le cose.
I motivi di ricorso alla consulenza più frequenti sono: • Identificazione ed inquadramento delle sindromi malformative
• Rischio di ricorrenza per famiglie con malattie ereditarie a trasmissione nota
• Inquadramento clinico di pazienti con cromosomopatie
• Problemi di consanguineità tra genitori
• Assunzione di farmaci o altre sostanze teratogene in gravidanza
I Professionisti coinvolti nell’attività clinica di genetica sono:
• Medici specialisti in genetica medica
• Medici specialisti in altre discipline (per i disordini appartenenti ad una determinata branca)
• “Genetic Counselors”: figure centrali nei servizi di genetica dei Paesi Anglosassoni, hanno per lo più un background di infermiere/ostetrica
Tipi di consulenza genetica: • Prenatale: per sospette o documentate malattie del feto. Sebbene le questioni relative alla genetica siano rilevanti in qualsiasi fase della vita, molte famiglie richiedono la consulenza genetica durante la gravidanza o nelle prime settimane dopo la nascita. In molti casi, del resto, la famiglia viene a conoscenza del rischio genetico proprio durante il periodo prenatale, in seguito ad un risultato abnorme delle indagini sul nascituro. • Neonatale: sospetta o documentata malattia del neonato • Riproduttiva o preconcezionale: consulenza nella programmazione della famiglia • Teratologica: stima il rischio di danno fetale per esposizione materna a possibili sostanze teratogene • Postnatale
• Oncologica
• Associata a Test Genetici di Screening
• Preconcezionale: nelle situazioni in cui il feto presenta un eccesso di rischio rispetto alla popolazione generale, ha lo scopo di: ◦ quantificare il rischio
◦ dare alla coppia la possibilità di elaborare le informazioni relative al rischio e valutare le possibili opzioni
◦ porre in tempo le basi per un’eventuale diagnosi prenatale Come si svolge la Consulenza Genetica
1. Raccolta delle informazioni
2. Costruzione dell’albero genealogico
3. Esame obiettivo del probando e dei suoi familiari
4. Richiesta ulteriori informazioni ed accertamenti specifici
5. Relazione scritta
6. A distanza di tempo: controllo della comprensione dei consultandi di quanto discusso
7. Aggiornamento diagnostico e terapeutico
Approccio non direttivo ma informativo, con neutralità del consulente nell’esposizione e nella scelta delle possibili opzioni: non direttività della Consulenza Genetica (termine mutuato dalla psicoterapia, indicante «la capacità del consulente di astenersi dall’esprimere giudizi personali che possano influenzare la persona nella propria scelta, impedendone l’autonomia decisionale»).
33
•
•
•
•
•
•
•
Consulenza Genetica Oncologica
risponde ai “perché” di chi appartiene a famiglie con ricorrenza di tumori
favorisce una più corretta percezione del rischio
informa sulle opzioni disponibili per la prevenzione
promuove una graduale presa di decisione riguardo l’eventuale test genetico e riguardo le opzioni diagnostiche e preventive
valuta le implicazioni psicologiche, limitando il rischio di un impatto negativo della comunicazione del rischio
permette la corretta interpretazione di risultati negativi o ambigui del test
utilizza metodi aggiuntivi per la stima del rischio
Consulenza genetica e Diagnosi fetale
Elevato rischio procreativo a priori:
• genitore portatore di anomalia cromosomica
• genitori portatori di mutazioni geniche
Basso rischio:
• età materna e precedenti anomalie cromosomiche
• aumentato rischio sulla base di test su siero materno
Riscontro inaspettato:
• malformazioni fetali evidenziate ecograficamente
La diagnosi genetica delle patologie più comuni (beta­talassemia, fibrosi cistica, alcune forme di sordità congenita ereditaria, ritardo mentale legato al sesso, distrofia muscolare di Duchenne) richiede la consulenza genetica per predisporre appropriate analisi molecolari o biochimiche che appurino lo stato di portatore sano. La ricerca delle mutazioni responsabili può essere effettuata anche direttamente in caso di villocentesi o di amniocentesi, purché la coppia sia sufficientemente informata su limiti e modalità tecniche delle indagini.
Diagnosi prenatale
Già all'11 settimana è possibile effettuare alcune indagini sul feto: la translucenza nucale (l'ispessimento depone per anomalie cromosomiche) e l'analisi di villi coriali. L'ecografia morfologica non è possibile prima della 20a settimana. Le indicazioni alla diagnosi prenatale: • Madre di età superiore a 35 anni • Precedente figlio con aneuploidia • Genitori portatori di anomalie cromosomiche strutturali o di aneuploidie sessuali • Patologia fetale ecoevidenziata • Indicazioni biochimiche Se il gene responsabile di malattia è noto, possiamo eseguire un test diretto per riconoscere se il gene in questione è mutato.
Se il gene non è conosciuto, ma è nota la sua localizzazione, possiamo eseguire un test indiretto per riconoscere se il feto ha ereditato la malattia.
L'analisi di villi coriali dà risultati definitivi già per la 13a settimana, mentre i risultati dell'amniocentesi non giungono prima della 19a settimana. Lo svantaggio del prelievo di villi sta in una più alta frequenza di falsi positivi rispetto all'analisi del liquido amniotico, a causa di mosaicismi o chimerismi. Mosaicismo: un mosaicismo è da considerarsi vero quando più cellule del a
34
☤ Alessandro G. - 2011/2012
prelievo mostrano le alterazioni. I falsi positivi (1­2%) possono derivare dal solo interessamento dei villi con il feto normale, pertanto il riscontro di trisomie del cromosoma 1 o 2 sono generalmente dei falsi positivi (per il feto sono anomalie letali). Nello stesso modo ci possono essere rari falsi negativi (0,04%) per interessamento del feto ma non dei villi. Riscontro di trisomie come 18, 17, ecc. devono essere confermate all'amniocentesi successivamente. Il mosaicismo può causare fenotipo malattia, o meno, in base alla distribuzione tissutale della linea cellulare interessata dall'anomalia ed in base alla percentuale di cellule anomale nel tessuto. Chimerismo: il riscontro di genotipo fetale con cellule 46XX e cellule 46XY fa sospettare un chimerismo che può essere falso (contaminazione da cellule materne) o vero chimerismo originato per fusione di due gameti (uno XY e uno XX). Il dubbio è risolto con l'analisi dei polimorfismi e la riconduzione degli alleli X ereditati. Riarrangiamenti bilanciati: il riscontro di riarrangiamenti impone di stabilire se questi riarrangiamenti sono di nuova insorgenza o sono ereditati. In questo ultimo caso il rischio di patologie fetale è minore rispetto a riarrangiamenti de novo.
Il basso rischio degli riarrangiamenti ereditati deriva dalla rarità di condizioni predisponenti: • Meno dell'1% ereditano un gene interrotto dal riarrangiamento ed un omologo mutato (malattie a trasmissione AR). In questi casi bisogna escludere riarrangiamenti complessi nei genitori. • In rarissimi casi, il riarrangiamento nel figlio è solo apparentemente bilanciato, ma durante la meiosi viene persa una parte nel materiale genico. Questi casi possono essere identificati con array­CGH. • Riarrangiamenti che coinvolgono cromosomi 7, 11, 14, 15 sono rari (<1%) ma in questi casi bisogna escludere la disomia uniparentale. I riarrangiamenti de novo presentano maggiori rischi per il feto (6% circa) con meccanismi diversi: • Traslocazioni apparentemente bilanciate • Traslocazioni robertsoniane • Inversioni • Inversioni e traslocazioni reciproche combinate La malattia in tal caso può essere causata da rotture del gene (AD), da effetto posizione (gene traslocato lontano dai suoi enhancers), e da disomie uniparentali. La diagnosi prenatale ha i suoi limiti. Innanzitutto formula solo un rischio empirico, inoltre non può fornire il fenotipo possibile del feto. La diagnosi può solo escludere una disomia uniparentale. Nel futuro si prospetta un ruolo sempre maggiore dell'array­CGH. Marcatori sovrannumerari: sono frammenti genomici sovrannumerari che si palesano alla FISH. I marcatori vengono distinti in: • Marcatori satellitari: bracci corti di cromosomi acrocentrici, di solito non attivi trascrizionalmente. Conferiscono un rischio del 10% di anomalie fetali. • Marcatori non satellitari: tutti gli altri segmenti cromosomici. Rischio di anomalie fetali pari al 20%. • Marcatori ad anello: cromosoma senza estremità libere richiuso su se stesso: fino al 50% di anomalie fetali. 35
Il significato della presenza di marcatori sovrannumerari varia in base a: • Origine del frammento: identificata con FISH. I più pericolosi sono i frammenti appartenenti ai cromosomi 13, 18, 21 che possono dare quadri trisomici. • Attività trascrizionale del materiale genetico sovrannumerario: eucromatina o eterocromatina, identificati con colorazioni particolari. I frammenti inattivi sono meno pericolosi. • Eccesso ereditato o de novo: la nuova insorgenza comporta un maggiore rischio. Li si distingue analizzando il cariotipo dei genitori. • Rappresentatività e percentuale: presente in tutte le cellule oppure un mosaicismo. In presenza di marcatori sovrannumerari bisogna escludere un'eventuale disomia uniparentale. Più del 50% dei marcatori originano dal cromosoma 15. le situazioni più frequenti che possono essere specificatamente ricercate sono: • Inv dup 15 • Isocromosoma 12p • Isocromosoma 16p GENETICA ONCOLOGICA
La predisposizione genetica a sviluppare tumori può essere legata ad alterazioni di uno dei seguenti gruppi di geni: • Oncogéni: mutazioni attivanti. Per manifestare il fenotipo basta un allele mutato. Gli oncogéni sono la causa principale dei tumori sporadici, e solo raramente si riscontrano come coinvolti nei tumori familiari (i rari esempi sono il gene Ret della MEN2 e il gene Met del ca. papillare renale familiare). • Oncosoppressori: le mutazioni inattivanti (secondo il modello a doppio hit proposto da Knudson nel 1971) sono implicate nella maggior parte di forme tumorali familiari. Alcuni esempi: ◦ VHL: sindrome di Von Hippel­Lindau (associazione di tumori renali ed emangiomi retinici e cerebellari. ◦ APC: FAP (Familial Adenomatous Polyposis)
◦ WT1: tumore di Wilms ereditario ◦ RB1: retinoblastoma ereditario ◦ P53: sindrome di Li­Fraumeni ◦ BRCA1/2: ca. mammario/ovarico ereditario ◦ MEN1: MEN1 • Geni di risposta al danno al DNA: mutazioni inattivanti di entrambi gli alleli determinano un accumulo di mutazioni e l'insorgenza di condizioni sindromiche complesse con tendenza allo sviluppo di neoplasie (anche se questa tendenza non domina il quadro clinico). Ne sono esempi: ◦ XP­A,C,D,F: xeroderma pigmentoso ◦ BLM: sindrome di Bloom, caratterizzata da alterazioni ematologiche (anche in senso neoplastico) ◦ hMSH­2,6/hMLH/hPMS­1,2/hEXO­1: ca. colorettale non associato a poliposi (HNPPC ­ sindrome di Lynch) ◦ ATM: atassia­teleangectasia
La trasmissione può seguire le regole dei caratteri autosomici dominanti in caso che la presenza di una mutazione (di un allele) sia sufficiente a conferire la predisposizione al 36
☤ Alessandro G. - 2011/2012
cancro, secondo il modello a doppio hit, ed è questo il caso della sindrome di Lynch, della FAP, del ca. mammario ereditario, della sindrome di Li Fraumeni, ecc., oppure la trasmissione può essere autosomica recessiva. La modalità di trasmissione recessiva è seguita dalle sindromi complesse come XP, AT, sindrome di Bloom, anemia di Fanconi, in cui sono necessari mutazioni di entrambi gli alleli per manifestare il fenotipo. Bisogna sottolineare che l'assenza di familiarità non esclude l'eventuale ereditarietà della neoplasia.
La penetranza, per tutte le forme di trasmissione, non è completa, grazie a influenze genetiche e ambientali. Per sviluppare il cancro sono necessarie più mutazioni oltre a quelle ereditate, e l'insorgenza di nuove mutazioni è favorita dal fumo, dai fattori ormonali, dalla capacità di riparazione del DNA, dai geni modificatori. Tutti questi fattori modificano la penetranza e influenzano il rischio del soggetto di sviluppare una patologia neoplastica anche se questa ha basi ereditarie. La consulenza genetica oncologica ha come obiettivo di valutare il rischio genetico individuale in base alle conoscenze disponibili e programmare eventualmente misure di sorveglianza adatte al caso specifico. Gli strumenti a disposizione del consulente sono: • Ricostruzione dell'albero genealogico che permette di apprezzare l'esistenza o meno delle caratteristiche di ereditarietà della patologia (non solo in base al pedigree): ◦ Presenza di casi multipli nella stessa famiglia ◦ Più generazioni colpite ◦ Associazioni di tumori specifici (geneticamente associati come mammella e ovaio) anche in assenza di familiarità ◦ Esordio in età più giovane del consueto ◦ Caratteristiche non neoplastiche delle specifiche sindromi • Con un fondato sospetto, si possono effettuare dei test genetici per ricercare la mutazione specifica del gene sospettato (in caso sia disponibile un tale test). Il limite del test genetico sta nell'incapacità di identificare tutti i casi di alterazioni: la negatività del test non esclude la presenza di una mutazione responsabile del quadro. 37
VI. GENETICA IN MEDICINA INTERNA
CARCINOMA DEL COLON RETTO (CCR) Il CCR rappresenta circa il 15% di tutte le neoplasie; ha un rischio del 5% nell’arco della vita, se si considerano individui della popolazione generale. È un ottimo esempio di come l’accumulo di mutazioni genetiche porti al cancro.
Ne esistono delle forme sporadiche (90%) e forme familiari (HNPCC, FAP, Peutz­Jagers e altre). Le forme sporadiche e la FAP (simile agli sporadici) esordiscono come polipi/adenomi, ed evolvono in carcinomi, mentre la HNPCC ha una progressione particolarmente accelerata e può presentare carcinomi senza polipi precedenti.
Si registrano 1'000'000 di nuovi casi/anno al mondo, dei quali 32'000 in Italia, dati che negli ultimi anni hanno registrato un aumento considerevole. Nelle forme sporadiche la sopravvivenza a 5 anni è del 50% circa, nonostante la terapia. Insorge tipicamente dopo i 50 anni, l'età di massima insorgenza è la 6 a ­ 7a decade di vita, senza differenze tra maschi e femmine; nell'80% dei casi la diagnosi è tardiva (neoplasia infiltrante la sottomucosa) per l'assenza di sintomatologia.
L'origine della forma sporadica è quindi una combinazione di fattori ambientali e fattori genetici.
Polipi e CCR
Di solito il carcinoma del colon retto origina da un polipo adenomatoso (dimostrato da vari studi), in seguito a mutazioni che attivano oncogéni e inibiscono geni oncosoppressori, anche se solo < 1% dei polipi è destinato a degenerare in senso maligno. Il sangue fecale è positivo in < 5% dei polipi, e può essere positivo in svariate situazioni (dalle lesioni gengivali alle emorroidi) pertanto l'utilità clinica del sangue occulto è molto dubbia. Le diverse conformazioni polipose hanno un diverso rischio di degenerazione maligna; il rischio di cancerizzazione è stimabile a seconda delle caratteristiche anatomopatologiche. I criteri di maggior rischio di trasformazione maligna sono: • Forma: sessili > peduncolati • Istologia: villosi > tubulo­villosi > tubulari • Dimensioni: più grandi le dimensioni più alto il rischio ◦ ≥ 2,5 cm → 10% ◦ 1,5 ­ 2,5 cm → 2­10% ◦ < 1,5 cm → < 2% Spesso i polipi non sono isolati, ma associati ad altri polipi o al CCR (30%), pertanto la diagnosi deve essere fatta con pancolonoscopia e non la sola rettosigmoidoscopia. 1. Fattori ambientali: ◦ IBD ­ Inflammatory Bowel Diseases (Rettocolite ulcerosa, morbo di Crohn, morbo celiaco senza dieta priva di glutine): il rischio aumenta con la durata della malattia e con la sua estensione anatomica. ◦ Fibre nella dieta: il consumo abbondante di vegetali riduce il rischio di cancro, viceversa i grassi animali lo aumentano leggermente (RR = 1,5). La fibra agisce da agente protettivo grazie a: ▪ Diluizione dei cancerogeni aumentando il volume fecale e trattenendo acqua
38
☤ Alessandro G. - 2011/2012
▪ Accorciamento del tempo di contatto ▪ Lega acidi biliari (irritanti) ▪ Facilita la fermentazione di acidi grassi a catena corta ▪ Può contenere agenti anticancerogeni (antiossidanti) ◦ Ca2+: in roditori il calcio previene la formazione di adenomi; nell'uomo è noto l'effetto inibente sulla proliferazione dell'epitelio intestinale. ◦ FANS: il consumo regolare di aspirina ha un effetto protettivo. 2. Fattori genetici: La familiarità è molto importante: i fratelli di un pz. con CCR hanno una probabilità di ammalarsi pari a quella della popolazione se in lui la patologia è insorta dopo i 50 anni, ma maggiore di 5 volte, se è insorto prima dei 50 anni. Se si ha un solo parente con cancro il RR è 2; se i parenti sono di più il RR diventa 7.
I geni coinvolti: ◦ Aumento dell’espressione degli oncogéni (basta anche una mutazione puntiforme su un solo allele): K­RAS (cr. 18, mutazioni puntiformi nel 40­70%), c­myc (sovraespresso nel 60­70%) ◦ Diminuzione dell’espressione degli oncosoppressori (inattivazione di entrambi gli alleli per delezione: concetto del doppio colpo o LOH ­ Loss of Heterozygosis): APC (cr. 5, perdita all’elica del 20­50%), p53 (cr. 17, perdita all’elica nel 75%, rara negli adenomi), MMG (cr. 5)
◦ Metilazione del DNA I geni APC e k­RAS sono frequentemente mutati in forme di cancerogenesi diretta che non passano attraverso polipi. La cancerogenesi è un risultato di accumulo di mutazioni specifiche non necessariamente in un preciso ordine temporale e non si riscontrano queste mutazioni specifiche nel 100% dei casi. Nonostante questo, la cascata degli eventi può essere così sintetizzata (ed idealizzata): 1. inattivazione oncosoppressore APC → piccolo adenoma
2. attivazione oncogéne RAS → grande adenoma con displasia
3. inattivazione p53 → cancro
4. anomalie a carico dei Mismatch Repair Genes → metastasi
APC: il gene APC è stato scoperto deleto dal cromosoma 5 in una famiglia di pazienti con FAP. Tutti i membri avevano una delezione di un braccio lungo del crom. 5 mentre l'altro portava la mutazione del gene codificando per una proteina tronca: si era realizzata una Loss of Heterozygosis (LOH). In tutti i malati di famiglie in cui vi è un’alta frequenza di CCR si è osservata la presenza di mutazioni a carico di un allele di APC e la perdita di parte dell’altro 39
allele. APC è stato clonato nel 1991 mediante identificazione della delezione interstiziale sul cromosoma 5q in pazienti con FAP e studi di analisi del linkage; studi successivi hanno dimostrato che APC è fondamentale anche in CCR sporadici (>70%, caratteristica peculiare dei CCR). Più del 90% delle mutazioni del gene APC determinano un segnale di stop prematuro e di conseguenza un prodotto genetico troncato ed inefficace. Tale proteina troncata tipica di APC mutati viene impiegata per lo screening genetico.
Il gene APC è un oncosoppressore gatekeeper. Codifica per una proteina di trasporto al proteasoma di proteine intracitoplasmatiche più piccole (β­ e γ­catenina, GSK­3β, tubulina, axina, GB1, hDLG, ecc.), espressa dalle cellule enteriche delle cripte della mucosa del colon, ovvero gli enterociti germinativi. APC mutato non è capace di indirizzare alla degradazione, veicolandole ai lisosomi, le proteine da eliminare: ne consegue il loro accumulo citoplasmatico.
La β­catenina stabilizza il legame tra caderine e alfa­catenina all’actina del citoscheletro (adesione intercellulare); trasloca il segnale della via Wiuglens­Wint (proteine WNT) durante lo sviluppo embrionale (la mutazione di WNT nella Drosophila ne causa la mancata crescita delle ali; le proteine WNT si legano alle proteine FRZ/Fz, che sono proteine G, attivando le proteine DSH); funziona da trasportatore delle proteine senescenti ai lisosomi per la degradazione legando il complesso WNT­FRZ­DSH. La β­catenina accumulata non lega più il complesso, e si accumula fino a penetrare nel nucleo, dove si lega al TCF (T­cell Factor) attivandolo. Il complesso attivo funge da fattore di trascrizione di vari oncogéni, tra cui c­myc, c­jun, PPAR­δ, ciclina D1. Qualunque mutazione o meccanismo che provochi l'accumulo di β­
catenina provoca lo stesso effetto!
Almeno alcuni di questi geni (sicuramente il PPAR­δ) sono repressi dai FANS o NSAIDS. I FANS hanno effetto analgesico grazie al blocco, tramite l’inibizione delle ciclossigenasi, della via dell’acido arachidonico: COX­1 è la ciclossigenasi costitutiva, e svolge azione “protettiva”, poiché tramite la PgI2 induce la produzione di muco gastrico, aumenta il flusso ematico e favorisce l’aggregazione piastrinica; COX­2 è la ciclossigenasi proinfiammatoria, attivando l'infiammazione (che è un meccanismo comunque protettivo, di difesa estrema) producendo PgE2, PgD2, PgF2α: è l’inibizione di quest’ultima isoforma che produce gli effetti dei farmaci antinfiammatori. La progressione del CCR nei pz. in cura con FANS è rallentata, probabilmente per interferenze di tali farmaci nella via di segnalazione che dalla β­catenina porta alla trascrizione degli oncogéni: i bloccanti delle prostaglandine sono sufficienti a bloccare il cancro!
La β­catenina interviene precocemente nella cancerogenesi (la mutazione di APC è presente già nei polipi), ed è un fattore fondamentale nella progressione della cascata di cancerizzazione. Metilazione del DNA: meccanismo forse residuo di infezioni virali, che i virus usavano per riconoscere il proprio DNA self da quello cellulare.
K­RAS: gene localizzato sul cromosoma 18, fa parte di una famiglia (H­, K­ e N­RAS) che codifica proteine con 7 domini transmembrana e funge da recettore per fattori di crescita cellulare. Funziona da “interruttore” legando GTP o GDP: attaccato dalla GTPasi, si inattiva.
Mutazioni a livello di K­RAS (anche solo puntiformi) possono rendere il sito di clivaggio inattaccabile alla GTPasi, con il risultato di mantenere sempre attiva la proteina.
È presente mutato in CCR sporadici (< 50%), nel 50% degli adenomi > 1 cm; la mutazione è rara negli adenomi piccoli.
Un possibile test di screening potrebbe essere la ricerca di RAS mutato nelle feci.
40
☤ Alessandro G. - 2011/2012
P53: gene localizzato sul cromosoma 17, il cui prodotto interviene nella modulazione di più di 20 diversi oncogéni. È un oncosoppressore­attivatore: in condizioni normali codifica per un attivatore trascrizionale dei geni inibitori della crescita, e funziona nelle condizioni di stress cellulare (ovvero quando sono più probabili gli errori di replicazione) favorendo da un lato la riparazione del DNA e, qualora questa non fosse possibile, promuovendo la morte cellulare.
È più frequentemente mutato nelle neoplasie maligne, solo raramente in quelle benigne.
Geni MMR (Mismatch Repair): le mutazioni di queste proteine, incaricate di individuare gli accoppiamenti sbagliati di basi, tagliarli e sistemare la base giusta, aumentano la probabilità di accoppiamenti di basi sbagliati e non riconosciuti; insorgono più frequentemente nelle aree microsatellitari e sono coinvolti in HNPCC (mutazioni; perdita allelica) e nel 20% di CCR sporadici (metilazione della regione promoter; Loss of Imprinting ­ LOI).
1. hMSH2, hMSH6 (human MutS Homolog 2 e 6): formano un dimero a forma di “mani in preghiera” che, scorrendo lungo il DNA, individua gli accoppiamenti sbagliati grazie alla loro diversa distanza tra le catene di DNA (normalmente larghe ~11 Å); un appaiamento di 2 pirimidine (singolo anello azotato) o di 2 purine (doppio anello azotato) ingombra diversamente rispetto alle normali coppie purina­pirimidina: una coppia di purine sarà più “larga” e bloccherà lo scorrimento, una coppia di pirimidine invece più “stretta” e fermerà lo scorrimento facendo perdere il contatto enzima­DNA.
2. hPMS1, hPMS2 (human PostMeiotic Segregation 1 e 2), hMLH1, hMLH3 (human MutL Homolog 1 e 3): hMLH1 si unisce a PMS2 e, sotto l’effetto di ATP, legano hMSH2­
hMSH6 al complesso precedente.
3. hEXO1 (human EXOnuclease 1): attivato dal complesso tetraproteico e dai 2 ATP si inserisce nel complesso e taglia la base sbagliata.
FORME FAMILIARI
Sindrome poliposica ereditaria ­ FAP (Familial Adenomatous Polyposis) La sua frequenza è di circa 1/10'000 nati vivi e la prevalenza è di 2­3/100'000 nella popolazione generale; i due sessi sono colpiti equamente, come nella forma sporadica. La diagnosi di FAP viene posta se viene soddisfatta una delle seguenti condizioni: • > 100 polipi all'endoscopia • Adenomi multipli (anche < 100) ed un parente affetto da FAP In realtà il 75­80% ha un genitore affetto. Ormai nella FAP la diagnosi precoce ha fatto calare a < 1% l'incidenza del CCR, grazie alla prevenzione mediante colectomia totale (con ileo­ano, ileo­
rettoanastomosi o ileostomia) prima dei 30 anni; la progressione maligna in cancro sarebbe infatti del 100%. Dal punto di vista genetico la cancerogenesi segue il modello classico a doppio hit, con un allele del gene APC ereditato non funzionante. I polipi compaiono quando viene persa la seconda e ultima copia del gene sul braccio lungo del cromosoma 5 (5q). I polipi hanno una prevalente localizzazione colorettale, ma interessano tutto il distretto gastroenterico, come lo stomaco (specie il fondo), il duodeno con la papilla di Vater ed il tenue. La poliposi esordisce mediamente a 16 anni, 41
comunque entro i 36 anni. Il CCR insorge a 39 anni in media, e l'incidenza cresce vertiginosamente con l'età. Nella FAP ci possono essere lesioni extraintestinali associate: • Malformazioni e tumori di SNC, tiroide, ossa • Cisti epidermiche • Tumori desmoidi (cicatrici)
• Dentizione anormale • Ipertrofia dello strato pigmentario della retina I test genetici per la FAP sono disponibili, e sono indicati per: • FAP incerte: molti polipi (>20), ma meno di 100, in assenza di familiarità • FAP manifesta, ma senza familiarità (può esserci un caso di paternità dubbia)
• Screening dei parenti di affetti da FAP con mutazione genetica nota, per riconoscere tra essi i portatori/affetti
I test genetici hanno un 10% di falsi negativi: la mancata evidenza di mutazioni genetiche non esclude la presenza di FAP.
In trattamento con FANS il numero di polipi si riduce significativamente già dopo pochi mesi di trattamento; ricompaiono però alla sospensione. HNPCC (Hereditary Non­Polyposic Colorectal Cancer)
o Sindrome di Lynch
Rappresenta lo 0,8 ­ 1% dei CCR; nella maggioranza dei casi nasce de novo, senza passare per la fase del polipo, che comunque è raro, pertanto la prevenzione è molto difficile; colpisce il colon. Nel 45% dei casi insorgono altre neoplasie a carico del colon, o di endometrio, mammella, ovaio, entro 10 anni dal carcinoma.
Il cancro della HNPCC è caratterizzato dal punto di vista genetico da: • Instabilità microsatellitare: le zone microsatellitari sono serie ripetitive di basi uguali (10 ­ 100 volte), che per questa caratteristica predispongono facilmente ad errori di accoppiamento di basi; sono quindi pericolose nei geni che controllano la crescita cellulare: si trovano anche all’interno di geni importanti, quale Bax (pro­apoptotico). Le sequenze dei microsatelliti diventano particolarmente lunghe nelle cellule cancerose.
• Mutazioni dei geni MMR (Mismatch Repair): presenti in più del 95% dei casi di HNPCC.
La mancanza del meccanismo di riparazione attuato dai MMR fa sì che le instabilità microsatellitari, non venendo riparate, si accumulino, con perdita della funzione degli oncosoppressori ed accelerazione della malignità del cancro. Alterazioni di microsatelliti e MMR (non dipendenti tra loro) diventano quindi pericolose quando sono concomitanti.
La diagnosi segue i criteri di Amsterdam o “Regola del 3­2­1­1­0”:
• 3 (o più) membri della famiglia affetti da CCR • 2 (o più) generazioni consecutive affette da CCR
• 1 (o più) affetto è parente di 1° grado degli altri due affetti
• 1 (o più) caso diagnosticato prima dei 50 anni • esclusione (0) della presenza di FAP Per porre la diagnosi “classica” di HNPCC devono essere soddisfatti tutti i 42
☤ Alessandro G. - 2011/2012
criteri. Circa l'85% delle diagnosi rispetta appieno questi criteri; il restante 15% è costituito da forme particolari che eccedono dalla norma.
È caratterizzato da esordio precoce, localizzazioni primitive multiple, pochi od assenti adenomi, predominanza nel colon destro; i tumori extracolici associati possono essere: • cheratoacantoma (tumore delle ghiandole sebacee)
• tumori epatobiliari • tumori gastrici e dell'intestino tenue
• tumori dell'apparato urogenitale: carcinoma della vescica, dell'endometrio e dell'ovaio
Questa malattia ha un rischio così elevato di sviluppare tumori, che il carcinoma dell'endometrio ha le stesse probabilità di insorgere di quello del colon, e paradossalmente potrebbe manifestarsi per primo!
Le linee guida per l'esecuzione dei test genetici sono: 1. Forte anamnesi di CCR familiare 2. Risultati interpretabili adeguatamente 3. Possibilità di influenzare la gestione del paziente o dei familiari EMOCROMATOSI
L'emocromatosi è un insieme di patologie caratterizzate da un abnorme accumulo di ferro a carico di diversi organi. Può avere origine genetica. In Italia colpisce 1/2'000 nati, ma in Nord Europa l’incidenza è maggiore, circa 1/200­350 (addirittura in Irlanda 1/83); è più rara nei non caucasici.
Omeostasi del Ferro
OMEOSTASI DEL Fe IN CONDIZIONI NORMALI
L'assorbimento del ferro nell'organismo è per via Eritrociti circolanti
intestinale, regolato da ipo­ od ipersideremia; l'escrezione 1500 - 2500 mg
non è correlata alla sideremia (desquamazione degli Emolisi
Eritropoiesi
enterociti, emorragie, mestruazioni, gravidanza). La (monociti/macrofagi)
(midollo osseo)
20 mg/die
20 mg/die
regolazione dell'omeostasi non si attua tramite l’escrezione del ferro (che infatti non dipende dalla concentrazione Pool plasmatico
Eliminazione
4 mg
bensì dal turnover) ma modificando l’assorbimento: Assorbimento
(intestino)
(intestino)
1-2 mg/die
1-2 mg/die
nell’emocromatosi viene a mancare il feedback di regolazione.
Il ferro è assunto tramite la dieta in quantità di 1­2 mg/die: Deposito corporeo totale: 4g
macrofagi tessutali
epatociti (300mg - 1g)
tramite gli enterociti, perlopiù duodeno­digiunali, il ferro passa nel pool plasmatico (4 mg), che scambia Fe con l’eritropoiesi (20 mg/die).
• L'enterocita presenta sul versante luminale Dcyt­B1 (citocromo­B1 duodenale; Fe­
reduttasi: ferrico Fe3+ → ferroso Fe2+), DMT1 (Trasportatore Metallico Divalente: assorbe lo ione ferroso), HPC1 (Proteina Trasportatrice di Eme: internalizza l’Eme di emoglobina e mioglobina); nel citoplasma OH (Eme­ossigenasi) degrada l’Eme liberando ferro Fe2+. Sul versante basale vi sono l’efestina (Fe­ossidasi: ossida il ferro ferroso, Fe2+, a ferrico, Fe3+) e la FP (Ferroportina: uscita nel plasma dei vasi della sottomucosa); nel plasma il ferro Fe3+ è trasportato dalla transferrina (Tf), da NTBI (Non­Transferrina Legante Ferro) e da altre proteine. La regolazione della maturazione degli enterociti è attuata anche da HFE che lega il recettore TfR1 basocellulare, 43
attivato da Tf­S, facendo diminuire i DMT1 sul versante luminale.
• Nei globuli rossi sono contenuti 2'500 mg di Fe e, quotidianamente, 20 mg sono liberati per l’eritrocateresi; il Fe è in equilibrio tra tale circuito e il deposito nei macrofagi variamente distribuiti (4'000 mg complessivi di cui 300 ­ 1'000 mg nel fegato). I macrofagi fagocitano i globuli rossi senescenti; al loro interno i fagolisosomi li lisano, l’emoglobina viene degradata, il Fe 2+ liberato dall’eme ed immagazzinato nei depositi di ferritina o riversato tramite ferroportina o ceruloplasmina nel plasma (nel versante extracellulare della membrana il Fe2+ viene ossidato a Fe3+) e legato dalla transferrina.
• All’interno dell’epatocita il Fe2+, rilasciato dalla degradazione intraepatocitaria di ferritina, eme, Hb, Tf e NTBI viene immagazzinato nei depositi di ferritina o riversato dalla FP nel plasma (nel versante extracellulare il ferro Fe2+ viene ossidato a Fe3+) e legato dalla transferrina o da NTBI.
Gli epatociti inoltre producono epcidina (attraverso la fase di pre­proepcidina e pro­
epcdina), che diminuisce l'immissione di Fe nel plasma; l’espressione dell'epcidina è modulata da HFE, TfR2 (Recettore della Transferrina) e HJV (emo­juvelina) che fungono da sensori del Fe. In condizioni normali un aumento del Fe plasmatico induce l'espressione di HFE, TfR2 e HJV, che a loro volta stimolano la trascrizione di HAMP (Hepcidin Antimicrobial Peptide), che porta infine alla trascrizione di epcidina: tale proteina, immessa in circolo, va a legarsi alla ferroportina e porta alla sua internalizzazione e successiva degradazione, inibendo così l'immissione di Fe nel plasma ad opera di enterociti e macrofagi. Ogni giorno, inoltre, 1­2 mg sono escreti con le feci, così da mantenere in equilibrio il pool complessivo.
L'emocromatosi è un disordine del metabolismo del Fe con progressivo accumulo intra­
parenchimale e potenziale danno multiorgano.
Esistono emocromatosi secondarie, che consistono in un sovraccarico di ferro secondario ad una patologia organica, sistemica, metabolica o iatrogena ed emocromatosi primarie o ereditarie, per mutazione ereditaria di uno o più geni coinvolti nel metabolismo del Fe.
Emocromatosi ereditarie
•
•
•
•
•
•
•
•
HFE mutato
C282Y omozigoti, 95% delle emocromatosi ereditarie
C282Y­H63D eterozigoti, 5%
H63D omozigoti
Non­HFE mutato
Emocromatosi da variante HJV
Emocromatosi da variante epcidina
Emocromatosi da variante TfR2
Emocromatosi da variante ferroportina
Emocromatosi secondarie
Anemie da carico di Fe:
Eritropoiesi inefficace (s. talassemiche, anemia sideroblastica, mielodisplasia, diseritropoiesi congenita); Aumentata eritropoiesi (anemia emolitica cronica)
Terapia marziale o emotrasfusioni multiple.
Sindromi metaboliche:
Obesità/insulino­resistenza, ipertensione
Epatopatie croniche:
Epatiti, alcol, NASH, porfiria cutanea tarda
Accumulo di Fe delle popolazioni africane
Esistono poi altre forme ereditarie, come la sindrome da iperferritinemia­cataratta congenita, il deficit di eme­ossigenasi (OH), l'accumulo neonatale di Fe, l'aceruloplasminemia congenita, l'a­/ipo­transferrinemia congenita, le varianti di DMT1.
44
☤ Alessandro G. - 2011/2012
Il ferro si accumula prevalentemente nel fegato causando epatomegalia fino a fibrosi, cirrosi ed infine HCC: fino alla fibrosi la patologia è reversibile con la terapia (salasso); la cirrosi epatica è pressoché assente con ferritina < 1 g/L. Le manifestazioni cliniche interessano anche l'adenoipofisi (ipogonadismo ipogonadotropo, infertilità, impotenza), il cuore (cardiomiopatia, scompenso, aritmie), il pancreas (diabete mellito), le articolazioni (artropatie), la cute (iperpigmentazione).
FORME EREDITARIE
L'emocromatosi ereditaria è un disordine ereditario del metabolismo del Fe con progressivo accumulo intra­parenchimale e potenziale danno multiorgano.
Caratteristiche fondamentali sono la natura ereditaria, in quasi tutti i casi autosomica recessiva a penetranza incompleta (tranne il tipo 4 che è AD), con aumento del Fe prima nel distretto plasmatico (aumento di sideremia e saturazione della transferrina) poi anche nelle cellule parenchimali (aumento della ferritinemia, danno d’organo) e l'eritropoiesi generalmente non alterata, con una buona risposta al salasso.
Il danno multiorgano è potenziale e scatenato da fattori secondari (ad esempio i maschi sono più propensi a sviluppare sintomi non avendo la perdita mensile di sangue mestruale).
Classificazione Genetica (OMIM)
Tipo 1. Emocromatosi HFE (o “classica”)
Autosomica recessiva, cr. 6p21; gene adiacente all'MHC di classe I, con i quali condivide la necessità di legarsi ad una β2­microglobulina per funzionare esprimendosi sulla superficie cellulare.
L'HFE epatica lega TfR2, inducendo HAMP e quindi l'epcidina, per ridurre la ferroportina; l'HFE enterocitario lega TfR1 (basocellulare) riducendo DMT1 (DcytB 1) e quindi l'assorbimento nei villi di Fe.
La mutazione è nel 95% dei casi una omozigosi C282Y, nei restanti casi una omozigosi H63D (od una eterozigoti C282Y­H63D).
La forma normale, presentando una cisteina in posizione 282, forma ponti S­S che consentono il legame tra HFE e la β2­microglobulina; se tale legame non è permesso, l’HFE diviene instabile ed è degradato bloccando così la via di feedback.
Coinvolge principalmente il fegato, l'apparato endocrino, il cuore. L'esordio è dopo i 40 anni, con astenia, artralgie, impotenza, infertilità, epatomegalia, diabete mellito, pigmentazione cutanea, cirrosi, scompenso cardiaco, aritmie; aumentano la transferrina e la ferritina. La 45
manifestazione clinica principale è l'epatopatia, per l'accumulo predominante negli epatociti. Il decorso clinico è moderato, la risposta alla salassoterapia è buona.
Step patogenetici e
alterazioni biochimiche
Fattori non correlati
al gene HFE
STORIA NATURALE DELL’OMOZIGOSI C282Y
Fattori ambientali
Fattori genetici
Mutazione HFE
Crescita, mestruazioni,
gravidanza, dieta,
perdite ematiche
Abuso etilico, malattie con
sovraccarico di Fe: talassemia,
porfiria, infezioni, HCV, NASH
Geni codificanti per epcidina, TfR2,
aptoglobina, emopessina,
ceruloplasmina, eme-ossigenasi 1
Geni implicati in
antiossidanti, fibrogenesi,
riparazione tissutale
↑ Fe
plasmatico
↑ Fe
tessutale
Danno
d’organo
↑ Saturazione
transferrina
↑ Ferritina
Ferritina > 1000 ng/ml
Alterazione test epatici,
endocrini e glicemici
Tipo 2a. Emocromatosi giovanile
Autosomica recessiva, cr. 1q21, gene per la HJV (emojuvelina), omozigosi G320V nel 50% dei casi.
HJV è un corecettore per la trascrizione di epcidina.
La patologia dà segni clinici prima dei 30 anni con interessamento cardiaco, endocrino, epatico (cardiopatia, ipogonadismo, diabete). L'esordio avviene dopo i 10 anni, con dolore addominale, ipogonadismo ipogonadotropo, aritmie e insufficienza cardiaca intrattabile, ridotta tolleranza glucidica; aumentano la transferrina e la ferritina. L'accumulo di Fe avviene negli epatociti, nel cuore, nelle ghiandole endocrine, nel muscolo scheletrico. Il decorso clinico è estremamente severo, la risposta alla salassoterapia aggressiva è buona.
Tipo 2b. Emocromatosi da epcidina
Autosomica recessiva, cr. 19q13, gene HAMP.
La pre­proepcidina è una proteina sintetizzata nel fegato in risposta al sovraccarico sideremico (la sovraespressione induce anemia o previene l'emocromatosi in caso di mutazioni C282Y). L'epcidina causa la ritenzione del Fe nei macrofagi, negli enterociti e nella placenta.
La clinica è analoga a quella della 2a. Tipo 3. Emocromatosi da TFR2
Autosomica recessiva, cr. 7q22, gene per TfR2: tale proteina è espressa perlopiù nel fegato, e ha affinità per il ferro superiore a TfR1.
Nelle cripte intestinali sembra possa interferire con HFE nel rilevamento della concentrazione di ferro. TfR2 è responsabile della captazione di transferrina coniugata con Fe ed è un corecettore per la trascrizione di epcidina.
La clinica è analoga all’emocromatosi di tipo 1, ma insorge prima (comunque dopo i 40 anni) ed è più grave.
Tipo 4. Emocromatosi da ferroportina
Autosomica dominante, cr. 2q32, gene per la ferroportina: se mutata, il Fe rimane bloccato nei macrofagi, negli enterociti, epatociti, nella placenta, con saturazione della transferrina a livelli normali o diminuiti ma ferritina enormemente aumentata (accumulo di Fe nei macrofagi con relativo risparmio degli epatociti), diminuzione del Fe circolante, ed aumentato 46
☤ Alessandro G. - 2011/2012
assorbimento intestinale.
Nel tipo I aumenta la saturazione di transferrina, con deposito di Fe nella ferritina epatocitaria; nel tipo II, più benigno e frequente, aumenta la ferritina macrofagica, con relativo risparmio degli epatociti.
La localizzazione coinvolge la milza ed in parte il fegato. Esordisce dopo i 40 anni con sintomi simili al tipo 1, accompagnati da anemia lieve; l'aumento della ferritina è notevole.
La manifestazione clinica principale è l'epatopatia. L'accumulo di Fe è principalmente nelle cellule reticolo­endoteliali. Il decorso clinico è lieve ma la risposta al salasso è scarsa.
ALGORITMO DIAGNOSTICO NEL SOSPETTO DI EMOCROMATOSI EREDITARIA (EE)
↑ Sat. Transferrina (>45%)
↑ Ferritina
Escludere malattie epatiche o
ematologiche ed altre cause
(flogosi cronica, infezioni)
Test genetici per
mutazione C282Y e H63D
• Omozigosi C282Y
• C282Y / H63D
EE tipo 1
• C282Y eterozigosi
• H63D eterozigosi/omozigosi
• No mutazioni HFE
Ferritina < 500 μg/L
Ferritina > 500 μg/L
Follow-up
RM (Fe epatico)
NON-HFE EE
↑ Fe
Nella norma
Ratio epcidina/ferritina
Ferritina >1000 μg/L
e/o ↑ GOT-GPT
Biopsia epatica: ↑ Fe
CEF > 4000 μg/g di peso secco
●
IEF > 1,9 (CEF/età)
●
NO EE
↓
↑
↓↓
●
Salasso ( → ferritina < 50 μg/L)
Valutazione genetica
Studi sulla famiglia
Follow-up
Test tipo 4
Test tipo 3
Test tipo 2a/2b
NO-EE tipo 2-4
●
EE tipo 2-4
●
AMILOIDOSI
Le amiloidosi sono patologie perlopiù autosomiche dominanti a penetranza variabile, causate da un disordine del folding (ripiegamento tridimensionale) di proteine che, divenute insolubili, precipitano sotto forma di fibrille. Sono un gruppo di malattie ad espressione clinica sistemica o localizzata, caratterizzate dalla deposizione extracellulare di aggregati proteici in forma fibrillare, con invasione lungo il polo neurovascolare, con alterazioni funzionali e strutturali degli organi coinvolti.
L'incidenza è di 8 casi per milione all'anno, senza distinzione di sesso, con un'età media di 50 ­ 70 anni. Pur essendo patologie sistemiche, ciascuna proteina invade preferenzialmente un organo: reni (74%), cuore (60%), fegato (27%), SNA (18%), apparato gastrointestinale (8%), SNC/SNP, articolazioni, pancreas, milza, cute. La prognosi è infausta a 12 anni dalla diagnosi nell’80% dei pazienti.
Il sospetto clinico si ha in qualsiasi paziente con una malattia sistemica multiorgano senza causa apparente (anamnesi familiare per forme ereditarie ­ autosomica dominante, ma con penetranze variabili). Entra in diagnosi differenziale con coronaropatie, neuropatie (amiloidosi TTR), IRC (amiloidosi Apo­A1, Apo­A2).
47
La diagnosi è data dalla colorazione con Rosso Congo del frustolo bioptico, così da evidenziare l’amiloide per la colorazione verdognola; se non è possibile ottenere una biopsia dell’organo interessato si procede biopsiando il grasso periombelicale o la mucosa rettale (che ha una sensibilità del 70 ­ 80% per TTR).
Le conseguenze degli accumuli amiloidi sono la compressione dei tessuti e l'ostacolo agli scambi tra capillari ed interstizio, causando atrofia, sclerosi ed infine insufficienza d'organo.
Calore
Stress
Mutazioni
Altri fattori
Aggregati amiloidosici
Lisi cellulare
Folding fisiologico delle proteine
Il folding delle proteine di nuova Catena
polipeptidica
sintesi è spontaneo e reversibile, e nascente
comprende la formazione di ponti Chaperon
disolfuro (S­S), catalizzata da disolfuro­isomerasi, e la formazione Prodotti intermedi
Chaperon, S-S
parzialmente ripiegati
di legami prolil­peptidici. Queste reazioni sono facilitate da molecole Proteina matura
chaperon che si legano alle proteine Chaperon
inducendo uno stato di avvolgimento Associazione ad ubiquitina
parziale in cicli di folding­unfolding­
R.E. → Golgi → secrezione
folding.
proteasoma
Le molecole chaperon inibiscono l’aggregazione di proteine con folding degradazione
parziale.
Classificazione
•
•
•
•
Anatomica
◦ Amiloidosi sistemiche (l'80 ­ 90% delle amiloidosi)
◦ Amiloidosi localizzate (il 10 ­ 20%)
Eziologica
◦ Amiloidosi secondarie
◦ Amiloidosi primarie
Genetica
◦ Amiloidosi ereditarie
◦ Amiloidosi sporadiche
Biochimica
◦ Tipo di proteina (>20 tipi diversi)
AMILODOSI SISTEMICHE
Caratteristiche cliniche delle amiloidosi sistemiche in SNC, SNP, SNA, SNE
• Alterazioni sensoriali simmetriche, centripete
• Neuropatia dolorosa, termica
• Neuropatia sensoriale­motoria progressiva (esordio di TTR)
• Disautonomia (es. ipotensione ortostatica)
• Sindrome del tunnel carpale
• Disfunzioni vescicali
• Impotenza
• Manifestazioni gastrointestinali
• Assenza di diabete
48
☤ Alessandro G. - 2011/2012
•
Presenza di presenza di gammapatia monoclonale (prim/sec)
SEDE
Discrasie immunocitiche B (plasmacitoma)
Rene, intestino, cuore, milza, cute, articolazioni, vie aeree
FIBRILLA PROTEICA
AL (amiloide a catene leggere)
Amiloidosi reattiva sistemica Rene, fegato, milza, surrene
(flogosi croniche; LH)
Da emodialisi (insufficienza renale)
AA (amiloide associata)
Osso, cute, legamenti, articolazioni
Aβ2m (β2­microglobulina)
Familiari Febbre mediterranea (AR)
Peritoneo, pleure, membrane sinoviali
Polineuropatia (AD)
Nervi periferici
Senile
Cuore, grandi arterie, polmone, prostata, pancreas
AA (amiloide associata)
ATTR (transtiretina Met/Val30 o Iso/Val122)
Amiloidosi AL (catene leggere): Incidenza: 8/1'000'000 per anno. Le fibrille sono costituite dal frammento N­terminale di un'immunoglobulina
monoclonale (plasmocitoma a catene leggere), comprendente la regione variabile ed una porzione della regione costante.
ATTR (prevalentemente normale)
DISCRASIE IMMUNITARIE
REATTIVE
Cause ignote
Infiammazione cronica
Proliferazione LB
Attivazione macrofagi
Plasmacellule
IL 1-6
Catene leggere Ig
Proteina SAA
STIMOLO
PRECURSORE
SOLUBILE
Amiloidosi AA (infiammazione cronica): Proteolisi parziale
le infiammazioni in causa sono le Malattie infiammatorie croniche: Malattia reumatica, FIBRILLE
AL
AA
infezioni croniche, febbri periodiche INSOLUBILI
ereditarie, neoplasie (Hodgkin’s, Ca. renale). Le fibrille sono costituite dal frammento N­
terminale di una HDL (SAA = Acute­Phase Reactant Serum Amyloid).
AMILOIDOSI LOCALIZZATE
SEDE
FIBRILLA PROTEICA
Tiroide
Pancreas
Pancreas
ACal (calcitonina)
AIAPP (amilina)
AIAPP (amilina)
Atri
AANF (fattore natriuretico atriale)
Neocorteccia, ippocampo, amigdala
Aβ (APP)
Neuroni cerebellari
AprP
HCHWA I (AD)
Arterie/arteriole cerebrali e leptomeningee
Cistatina C (Glu/Leu68)
HCHWA O (AD)
Arterie/arteriole cerebrali e leptomeningee
Aβ (APP)
Endocrine
Carcinoma tiroideo
Insulinoma
Diabete Tipo II
Amiloidosi isolata atriale
Cerebrali
M. Di Alzheimer
Encefalopatia spongiforme
49
AMILOIDOSI EREDITARIE
Sono una serie di patologie (ad ereditarietà autosomica dominante e penetranza variabile) ciascuna delle quali è la conseguenza di una o più mutazioni in una proteina specifica che, pur manifestando distribuzione sistemica, esprime tendenza a depositarsi in tessuti preferenziali, che ne condizionano le caratteristiche cliniche.
PRECURSORE PROTEICO
CLINICA
FORME SISTEMICHE
AF
ATTR
APO­A1,2
Afib
Agel
Alys
ACyst
Transtiretina
Apolipoproteina A1,2
Fibrinogeno
Gelsolina
Lisozima
Cistatina C
Amiloidosi familiare e senile (polineuropatia)
Amiloidosi familiare renale (sd. Nefrosica)
Amiloidosi familiare renale (Ostertag)
Amiloidosi familiare
Amiloidosi familiare
Amiloidosi familiare
FORME LOCALIZZATE
AF
Abri
Adan
Aker
ALac
AbriPP
AdanPP
Cheratoepitelina
Lattoferrina
Demenza, familiare
Demenza, familiare
Cornea, familiare
Cornea, familiare
Amiloidosi TTR: la transtiretina è una proteina plasmatica a struttura tetramerica formata da 4 subunità identiche di 127 aminoacidi; viene sintetizzata dal fegato, è presente anche nel cervello (plessi corioidei) e nell’occhio (epitelio pigmentoso della retina). Trasporta la proteina­legante il retinolo e un 1/4 della tiroxina sierica (sito centrale).
Essa è codificata da un gene di 4 esoni sul cr. 18:
• esone 1: 20 AA (solo 3AA nella forma matura)
• esone 2: AA 4 ­ 47
• esone 3: AA 48 ­ 92
• esone 4: AA 93 ­ 127
Le mutazioni amiloidosiche identificate sono più di 80 e vengono trasmesse come carattere autosomico dominante; sono mutazioni di singoli nucleotidi nel gene TTR: non si conoscono mutazioni nell'esone 1, ce ne sono 37 per l'esone 2, 45 per l'esone 3, 16 per l'esone 4. Le mutazioni accelerano la tendenza amiloidosica spontanea di TTR a depositarsi (si ritrova anche non mutata nei depositi di amiloide). L'unica terapia nota è il trapianto di fegato (esistono rari casi di progressione anche dopo trapianto).
Essa è la resposabile della Familial Amyloid Polineuropathy (FAP 1), che ha prevalenza del 3 ­ 5%, penetranza del 50%. La progressione è centripeta (verso il SNC) ed inizia dal SNP prevalentemente dagli arti inferiori con sintomi sensoriali; prosegue poi con una polineuropatia sensoriale e motoria progressiva (parestesie, dismotilità ingravescenti), disfunzioni gastrointestinali e urinarie (alvo alterno, CIP, ritenzione), ipotensione ortostatica e più tardivamente interessamento cardiaco, che è la causa di morte (cardiomiopatia restrittiva). I depositi sono visibili nel fundus oculi. C’è interessamento articolare con progressiva distruzione delle sinovie. Raramente vi sono depositi di amiloide meningei e 50
☤ Alessandro G. - 2011/2012
cerebrovascolari. Amiloidosi non­TTR
• ApoA1: proteina normalmente di 243 AA, l’amiloide è formata da residui tra l’AA 83 e il 93; 12 mutazioni (generalmente sostituzioni di singoli nucleotidi) sul cr. 11 inducono la deposizione di amiloide ottenuti da una degradazione incompleta. Ad ereditarietà autosomica dominante, variano la penetranza, l'età d’insorgenza e la severità del quadro clinico a seconda della mutazione: mutazioni del terminale carbossilico (Dominio C­terminale, CTD) alterano il metabolismo della proteina con deposizioni renali (interstiziali), epatiche e cardiache; mutazioni del terminale amminico (Dominio N­terminale, NTD) causano deposizioni cutanee e laringee.
• Gelsolina: gene sul cr. 9, è sintetizzata nel muscolo scheletrico, che aiuta a rimodellare, e nei macrofagi: è coinvolta nel clivaggio dell’actina (essenziale per il metabolismo e la riorganizzazione citoscheletrica). Si conoscono due mutazioni (Asp187Asn e Asp187Tyr) che portano alla deposizione di amiloide, composta da un segmento interno della gelsolina costituito da 71 AA comprendenti il residuo mutato; la clinica è caratterizzata da distrofia corneale, neuropatia craniale, lassità cutanea del volto, mentre la patologia renale ha progressione più rapida nella mutazione Asp187Asn.
• Lisozima: è una proteina di 14'500Da con proprietà batteriolitiche (è un indice di flogosi, aumenta anche in linfomi/leucemie); si conoscono 4 mutazioni associate a deposizione di amiloide, composta di vari segmenti, ma anche della proteina intatta. La deposizione, nei glomeruli, causa una tardiva insufficienza renale cronica (insorgenza nella 3a e 4a decade di vita). Il trapianto di rene è frequentemente efficace.
• Cistatina: inibitore della proteasi, Leu68Gln, insorge a 30­40 anni con emorragie cerebrali recidivanti.
• Fibrinogeno: muta la porzione sensibile alla proteasi, con nefropatia recidivante dopo il trapianto entro 10 anni; il trapianto di fegato blocca la progressione della malattia.
• ApoA2: si deposita il C­term (21AA) con insufficienza renale cronica che in taluni casi non recidiva dopo il trapianto.
LA MALATTIA DI ALZHEIMER
È una malattia neurologica del gruppo delle demenze (di cui è la principale causa: seconda ad essa troviamo il Parkinson), caratterizzata da eterogeneità genetica (mutazioni in geni diversi che portano allo stesso fenotipo). Per demenza si intende una «alterazione globale e spesso irreversibile e progressiva delle funzioni corticali più alte, includenti apprendimento di abilità percettivo­
motorie, corretto uso di abilità sociali, controllo delle reazioni emotive senza evidente ottundimento della coscienza».
Forme di Alzheimer:
• Sporadico (75%): insorgenza tardiva (dopo i 65 anni), malattia multifattoriale
• Familiare (25%): insorgenza precoce, autosomica dominante
◦ ad esordio precoce (< 65 anni):
▪ APP → AD1 (Alzheimer Disease type 1)
▪ PS­1 → AD3
▪ PS­2 → AD4
51
◦ ad esordio tardivo (> 65 anni):
▪ APO­E → AD2
L'Alzheimer colpisce più di 4 milioni di persone negli USA e circa un milione in Italia, con una spesa sociale che si stima intorno ai 50 miliardi $ e 10 miliardi € all'anno. Insorge generalmente dopo i 40 anni, e risulta essere il 75% delle demenze oltre i 60 anni, con una prevalenza del 10% tra gli ultra­70enni.
È caratterizzata dalla deposizione di ammassi fibrillari extracellulari ed intracellulari che coinvolgono i Nuclei della base (neuroni contenenti acetilcolina, importante per l’acquisizione della memoria e l’apprendimento), l'ippocampo (essenziale per la conservazione della memoria), la corteccia cerebrale (formazione del pensiero e del linguaggio).
Le fasi precoci della malattia interessano l'ippocampo, con una progressiva perdita della memoria recente e della capacità di svolgere compiti routinari; avviene poi il coinvolgimento della corteccia cerebrale: impoverimento del ragionamento, esplosioni emotive, alterazione del linguaggio. La morte di numerosi neuroni porta ad alterazioni del comportamento (agitazione), e nelle fasi finali sopraggiungono l'incapacità di riconoscere le facce, di comunicare, la perdita di controllo degli sfinteri anale e vescicale. Il tempo medio dalla diagnosi alla morte è di 4 ­ 8 anni, sebbene la malattia talora possa durare più di 20 anni.
La diagnosi di certezza è solo anatomopatologica (biopsia/autopsia), che rivela placche neuritiche “senili” ed ammassi fibrillari in ippocampo, corteccia temporale, nuclei della base. La clinica (deterioramento progressivo) comunque consente di fare una diagnosi di possibilità o probabilità e lo stesso valga per le analisi laboratoristiche nonché strumentali (alterazioni inizialmente del metabolismo della corteccia parietale, atrofia dell'ippocampo nelle fasi avanzate).
È possibile attuare analisi tramite RMN, TC, SPECT (TC a Emissione di Fotone Singolo), PET (Tomografia a Emissione di Positroni); verranno evidenziati riduzione del flusso di sangue, assottigliamento dei tessuti, riduzione della massa, anomalo utilizzo del glucosio.
I principali tipi di lesione istologica sono le placche amiloidi o senili (extra­
neuronali) e la degenerazione neurofibrillare (intra­neuronale): la sostanza amiloide, componente essenziale delle placche senili, è principalmente composta da una proteina (βA4) che deriva dalla proteolisi di un precursore di maggiori dimensioni, PPA (Amyloid Protein Precursor); i neuroni appaiono raggrinziti, le fibrille partono a livello del citoplasma perinucleare e spesso si estendono sino ai dendriti apicali, come masse di filamenti abnormi, di forma globosa o a fiamma, che al microscopio ottico presentano una struttura fibrillare (NFTi, NeuroFilamenti intraneuronali).
I fattori di rischio sono sia non genetici, quindi potenzialmente modificabili, che genetici e pertanto non modificabili. I fattori non genetici sono:
• Fattori di rischio vascolari:
◦ ipertensione sistolica > 160 mmHg ◦ colesterolo sierico > 6,5 mmol/L • Fattori di rischio dovuti allo stile di vita: ◦ fumo di tabacco
◦ scarsa attività fisica 52
☤ Alessandro G. - 2011/2012
◦ nessuno/eccessivo consumo di alcool ◦ traumatismi cranici
• Fattori di rischio socio­demografici: ◦ età avanzata
◦ sesso femminile
◦ periodo di istruzione < 15 anni
◦ lavoro che espone a tossine ambientali (es. metalli pesanti)
• Infezioni virali
• Depressione • PAN (anamnestica)
• Ipertiroidismo • Familiarità per sindrome di Down I geni coinvolti sono:
• gene della Proteina Amiloide (APP), cr. 21
β­APP (Amyloid­Beta Precursor Protein) è un recettore tipo G, che favorisce adesione cellulare, crescita sinaptica, riparazione neurale; la mutazione causa incapacità di formare l’intera proteina β­APP, con formazione di peptidi Aβ, più corti, frammenti di 40 ­ 43 AA (Amiloide Aβ), che si depositano in placche extraneuronali insieme ad APO­E, proteoglicani, anti­chimotripsina portando alla morte dei neuroni interessati.
→ AD1: le mutazioni sono rare (20 famiglie nel mondo); l'esordio è precoce (tra i 35 e 50 anni).
Evidenze a favore del ruolo del gene APP come sede delle mutazioni responsabili di Alzheimer:
◦ βA4 (Aβ peptide di 42 AA, prodotto del clivaggio proteolitico di β­APP) è il principale componente della placca amiloidotica neuronale caratteristica della malattia di Alzheimer.
◦ La sede del gene per l'Alzheimer è il cromosoma 21 (da studi di linkage genetico).
◦ I pazienti con trisomia 21 (doppia coppia del gene β­APP) sviluppano la malattia entro i 40 anni.
◦ Mutazioni del gene β­APP sono state identificate in pazienti con emorragie cerebrali ereditarie ed amiloidosi vascolare (abbondante deposito perivasale di Aβ; sporadiche placche diffuse a tutto il parenchima cerebrale).
• gene della Presenilina 1 (PSEN1), cr. 14 Le preseniline sono una famiglia di proteine di transmembrana che svolgono funzioni di proteasi (γ­secretasi). PSEN1 è molto simile a PSEN2; codificano per proteine simili, probabilmente costituite da 7 domini transmembrana (anche se studi recenti avrebbero dimostrato la presenza di 8 domini). Mutazioni missense causano la produzione di βA4 con 42 AA.
→ AD3: la causa genetica più comune di Alzheimer familiare ad esordio precoce; forma grave. Il gene PS­1 codifica per una proteina denominata S182; la mutazione ha trasmissione autosomica dominante ed induce una demenza a esordio precoce (tra i 45 e 55 anni) con andamento molto rapido e morte in pochi anni. Le mutazioni (più di 50) sono state identificate in numerose famiglie.
53
gene della Presenilina 2 (PSEN2), cr. 1 Il gene PS­2 codifica per una proteina chiamata STM2.
→ AD4: le mutazioni sono ad esordio precoce, ma meno di PS­1 (l'età media è 53 anni), e l'andamento è un po' meno grave (la sopravvivenza media è 11 anni). Le mutazioni furono identificate per la prima volta in una famiglia americana, di origini germaniche del Volga, e successivamente in una famiglia di Firenze.
Nell'AD1 le mutazioni di β­APP aumentano il catabolismo ad opera delle β­
secretasi. Nell'AD3 e AD4 le mutazioni di PSEN­1 e PSEN­2 aumentano l'attività delle γ­secretasi.
•
•
gene della Apolipoproteina E (APO­E), cr. 19
APO­E è una proteina plasmatica di 299AA coinvolta nel trasporto del colesterolo; lega la proteina βA4. Esistono 3 isoforme: ε3 è la forma wild­
type, ε2 ha una sostituzione Arg158Cys, ε4 una sostituzione Cys112Arg.
→ AD2: associato anche a forme sporadiche; esordio tardivo (> 65 aa)
L'allele ε4 aumenta il rischio di Alzheimer, mentre l'allele ε2 diminuisce il rischio: l’acquisizione con il patrimonio genetico dell’allele ε4 del gene APO­E aumenta il rischio di sviluppare Alzheimer, nelle sue forme sia precoce che tardiva, e diminuisce l’età di insorgenza della forma tardiva della malattia. Non è ancora chiaro se l’allele ε4 sia sufficiente di per sé a determinare la malattia e, a differenza delle mutazioni missense nei geni APP, PS1 e PS2, la sua scoperta in soggetti presintomatici è di significato incerto: la scoperta dell’allele ε4 del gene APOE in un soggetto con un quadro clinico compatibile con Alzheimer può avere utilità clinica ancillare.
I differenti alleli di APO­E potrebbero esercitare i loro diversi effetti influenzando in modi diversi il metabolismo lipidico e/o la riparazione neuronale: ε4 ha forte avidità di legame per Aβ diminuendone la clearance, e la proteina τ in presenza di ε4 agisce più liberamente alla formazione dei neurofilamenti.
Le proteine τ sono MAPs (Microtubule­Associate Proteins): proteine altamente solubili (6 isoforme, da 352 a 441 AA) associate ai microtubuli neuronali (soprattutto del SNC, nelle porzioni distali degli assoni, per il 54
☤ Alessandro G. - 2011/2012
trasporto intracellulare di messaggeri molecolari). Esse interagiscono con la tubulina per la sintesi e stabilizzazione dei microtubuli. La loro fosforilazione su Ser262 o Ser214 (Tau iperfosforilata) produce precipitati insolubili portando alla separazione dai microtubuli, accumulo sotto forma di microfilamenti e perdita di funzione. La localizzazione coincide con le zone di danno neuronale ed è associata con la presenza dell’allele 4 di APO­E.
FORMA SPORADICA
Fattori ambientali
Predisposizione genetica
↑ produzione o ↓ clearance
di Aβ
FORMA FAMILIARE
Oligomerizzazione ed iniziali
depositi di Aβ 42
Alterate funzioni sinaptiche
Mutazione di β-APP
↑ produzione di Aβ
↑ rapporto Aβ 42/40
Risposta infiammatoria e formazione delle placche amilodosiche
Progressivo danno di sinapsi e assoni
Alterazione dell’omeostasi neuronale e stress ossidativo
Alterata oligomerizzazione ed iperfosforilazione di tau
Diffusione del danno neuronale, morte cellulare + deficit di neurotrasmettitori
Demenza e neurodegenerazione
55