IBRIDAZIONE MOLECOLARE E MAPPATURA DEI GENI
La possibilità di visualizzare la posizione dei geni o di altre regioni non codificanti del genoma su
un cromosoma (mappatura) consente anche l’individuazione di anomalie submicroscopiche di
struttura dei cromosomi, in quanto è possibile visualizzare lo spostamento (traslocazioni), la
perdita (delezioni), o l’aggiunta (duplicazioni) di tratti cromosomici anche molto piccoli, non
rilevabili con la citogenetica convenzionale.
Ibridazione molecolare
La procedura su cui si basa la mappatura di un gene è l’ibridazione molecolare.
Quando una soluzione acquosa di DNA è riscaldata a 100 °C o esposta ad un pH molto alto, i
legami idrogeno tra le coppie di basi complementari che normalmente tengono insieme i due
filamenti della doppia elica vengono rotti e la doppia elica si dissocia in due filamenti singoli.
Questo processo viene chiamato denaturazione del DNA.
I singoli filamenti di DNA in particolari condizioni (abbassamento della temperatura a 65 °C,
variazione di pH ) possono riassociarsi a riformare doppie eliche secondo un processo chiamato
rinaturazione del DNA. Reazioni simili possono avvenire fra due catene di DNA a singolo
filamento di diversa origine, purché abbiano sequenze nucleotidiche complementari, e il risultato è
definito ibrido molecolare.
Una sonda molecolare o probe è un frammento noto di DNA a singolo filamento usato in un
esperimento di ibridazione molecolare per identificare molecole specifiche di DNA nelle quali è
presente una sequenza complementare alla sonda stessa.
Una sonda molecolare viene marcata con isotopi radioattivi o sostanze fluorescenti per consentirne
la rivelazione. Le reazioni di ibridazione con sonde di DNA possono essere effettuate in provetta (in
vitro), o con campioni di DNA posizionati su filtri di nylon o di nitrocellulosa (Southern Blotting)
per rivelare l’eventuale presenza in un particolare campione di DNA di sequenze complementari
alla sonda.
Questa tecnica, come la maggior parte delle tecniche di biologia molecolare prevede l’utilizzo di
acidi nucleici purificati, il che implica la distruzione delle strutture cellulari e cromosomiche del
campione e l’impossibilità di localizzare direttamente sui cromosomi geni o qualsiasi altra sequenza
di DNA rappresentata dalla sonda molecolare.
Per superare questo problema sono state sviluppate tecniche di ibridazione molecolare in cui sonde
marcate di acidi nucleici sono usate per localizzare sequenze specifiche di acidi nucleici in una
determinata struttura biologica, cioè in situ, attraverso un procedimento chiamato ibridazione in
situ. L’ibridazione viene fatta avvenire non in provetta, ma mettendo a contatto la sonda con un
1
substrato solido su cui, nel caso in cui si voglia localizzare un gene su un cromosoma, è stato
allestito un preparato cromosomico. L'ibridazione in situ permette quindi di visualizzare l'acido
nucleico d'interesse nella sua sede naturale, preservando perfettamente la struttura tessutale,
cellulare o cromosomica del preparato.
Ibridazione in situ fluorescente
L'ibridazione in situ fluorescente (FISH : Fluorescence In Situ Hybridization) è una tecnica di
ibridazione in situ che permette di visualizzare a livello cromosomico la localizzazione di un gene o
di una qualsiasi sequenza genomica d'interesse, usando sonde di DNA marcate con composti
fluorescenti. Il vantaggio, rispetto alla marcatura con isotopi radioattivi, è rappresentato
dall’eliminazione del radioattivo, dall’ottenimento di un “segnale” più nitido e dalla possibilità di
usare fluorocromi a diversa emissione cromatica, consentendo di usare visualizzare
contemporaneamente e più sonde.
Attraverso questa tecnica è possibile pertanto localizzare su cromosomi metafasici sequenze
genomiche di limitata estensione e di identificare una loro eventuale localizzazione alterata
(traslocazione e inversione) o una loro assenza (delezione), o una loro aggiunta (duplicazione).
Tale metodica, applicata ai cromosomi metafasici, ha incrementato il livello di risoluzione della
citogenetica convenzionale che permette di identificare le anomalie cromosomiche superiori a 4 Mb
(1 Mb = 106 paia di basi). La FISH invece consente di visualizzare anomalie che coinvolgono poche
migliaia di paia di basi.
Inoltre, la possibilità di utilizzare per questo tipo di studi anche nuclei interfasici permette di
analizzare anche tipi cellulari che difficilmente vanno incontro a divisione cellulare, oppure inclusi
in paraffina e come tali non più coltivabili in vitro.
La procedura è abbastanza semplice e prevede:
1. La scelta di una sonda, ossia di una sequenza conosciuta di DNA
2. La marcatura della sonda con composti fluorescenti che vengono legati ad essa
3. L’allestimento dei preparati da studiare
4. La denaturazione del DNA, sia dei preparati sia della sonda, per consentire successivamente la
formazione di legami idrogeno tra il filamento del DNA cromosomico e i filamento del DNA
della sonda
5. L’ibridazione: appaiamento del DNA dei preparati con il DNA sonda, introdotto in eccesso
6. Il lavaggio dalla sonda in eccesso non appaiata
7. L’osservazione dei preparati con microscopio a fluorescenza
2
Classificazione delle sonde
Fig. 1
Sonde cromosoma-specifiche
La sonda cromosoma-specifica è costituita da un insieme di sonde di DNA ognuna delle quali è omologa all’intero cromosoma considerato.
L’insieme delle sonde copre l’intero cromosoma. L’ibridazione con una sonda cromosoma-specifica marcata con un fluorocromo permetterà di
visualizzare segnali fluorescenti su una determinata coppia di cromosomi omologhi lungo tutto il cromosoma.
Sonde centromero-specifiche
La sonda centromero-specifica è costituita da un tipo di sequenze ripetute, dette alfoidi, presenti nei centromeri. Sono state isolate sequenze alfoidi
specifiche per il centromero di ogni cromosoma. L’ibridazione con una sonda centromero-specifica marcata con un fluorocromo permetterà di
visualizzare segnali fluorescenti a livello dei centromeri di una coppia di cromosomi omologhi.
Sonde telomero-specifiche
La sonda telomero-specifica è costituita da sequenze localizzate nella parte terminale dei cromosomi. Sono state isolate vicino alla parte terminale
della molecola di DNA, che ha una sequenza comune a tutti i cromosomi, sequenze specifiche per il telomero di ogni cromosoma. E’ più corretto
definire le sequenze telomeriche specifiche per uno dei due bracci di un determinato cromosoma (braccio corto “p”, braccio lungo “q”) “sequenze sub
telomeriche”. L’ibridazione con una sonda telomerica specifica per uno dei bracci di un determinato cromosoma, marcata con un fluorocromo,
permetterà di visualizzare segnali fluorescenti a livello dei telomeri di uno dei due bracci di una coppia di cromosomi omologhi.
Se la sonda telomero-specifica è costituita dalle sequenze che costituiscono la parte terminale della molecola di DNA, comuni a tutti i cromosomi, la
sonda viene detta specifica per i “telomeri comuni”. In questo caso in seguito all’ibridazione si visualizzeranno segnali fluorescenti sui telomeri di
tutti i cromosomi.
Sonde locus-specifiche
La sonda locus-specifica è costituita dalla sequenza di una regione delimitata lungo uno specifico cromosoma. L’ibridazione con una sonda locusspecifica marcata con un fluorocromo permetterà di visualizzare segnali fluorescenti a livello di una regione sul braccio corto o sul braccio lungo di
una coppia di cromosomi omologhi.
3
Fig. 2. Ibridazione in situ con diverse tipologie di sonde
a) Ibridazione in situ con una sonda specifica per il cromosoma 8: i segnali fluorescenti sono presenti lungo tutta l’estensione dei due cromosomi
omologhi. b) Ibridazione in situ con una sonda specifica per le sequenze centromeriche del cromosoma X: i segnali fluorescenti identificano due
cromosomi X. c) Ibridazione in situ con una sonda specifica per i telomeri del braccio lungo del cromosoma 4: i segnali fluorescenti sono presenti sui
telomeri del braccio lungo dei due cromosomi 4 omologhi. d) Ibridazione in situ con una sonda specifica per una regione del braccio lungo del
cromosoma 17: i segnali fluorescenti sono presenti in una regione intracromosomica del braccio lungo di entrambi gli omologhi 17.
4
Fig. 3 Livello di risoluzione della FISH
FISH su cromosomi metafasici
Il livello di risoluzione della FISH viene definito in base alla distanza minima alla
quale si vedono due segnali distinti prodotti dall’ibridazione di due sonde, questo
dipende dal grado di allungamento e/o di despiralizzazione della cromatina. Il
cromosoma metafasico rappresenta lo stadio in cui la cromatina raggiunge il
massimo grado di compattamento.
Il livello di risoluzione della FISH su cromosomi metafasici (vedi allestimento
preparati cromosomici convenzionalli.) va da 2 a 3 megabasi (Mb).
FISH su cromosomi metafasici allungati meccanicamente
I cromosomi metafasici possono essere allungati partendo da preparazioni di
cromosomi metafasici convenzionali sottoposti a stiramento meccanico.
Il livello di risoluzione della FISH su cromosomi metafasici stirati è al massimo di
200 kilobasi (Kb).
Fig. 4. FISH con due sonde marcate con due fluorocromi
diversi su cromosomi a diverso grado di allungamento
a) FISH a doppio colore su cromosomi metafasici con due
sonde localizzate a 2,5 Mb di distanza: si può notare la quasi
totale sovrapposizione dei segnali fluorescenti. b) FISH a
doppio colore su cromosomi metafasici stirati
meccanicamente con le stesse sonde utilizzate in a:
l’allungamento del cromosoma permette di visualizzare i due
segnali fluorescenti rosso e verde separati da un segmento
cromatinico. c) FISH a doppio colore su fibre di DNA con le
stesse sonde utilizzate in a e in b: l’ulteriore despiralizzazione
della cromatina è evidenziato dall’aumento della distanza tra i
segnali fluorescenti rosso e verde.
FISH su fibre di DNA
Le fibre di DNA possono essere ottenute incubando nuclei interfasici strisciati su un
vetrino portaoggetto in una soluzione di lisi che rompe la membrana nucleare. Il
vetrino viene quindi estratto verticalmente dalla soluzione di lisi. Questo movimento
permette alle fibre di DNA, fuori uscite dal nucleo, di srotolarsi e di disporsi
longitudinalmente lungo il vetrino.
Il livello di risoluzione della FISH su fibre di DNA può arrivare a 1 Kb.
5
MODELLO DIDATTICO DELLA IBRIDAZIONE IN SITU
Per rendere maggiormente comprensibile questa tecnica si propone l’utilizzo di un modello che
permetterà agli studenti di acquisirne i principi di base, apprezzandone le diverse applicazioni.
Simulazione ibridazione in situ (Brevetto n°: MI2005A000710, Milano 20/05/05)
Il modello è costituito da una tavola di legno, che simula il vetrino porta oggetto su cui vengono
strisciati i cromosomi. Sopra il piano di legno vengono collocate 3 coppie di cromosomi. Ogni
cromosoma è suddiviso in regioni contraddistinte da numeri sulla superficie superiore, mentre sulla
superficie inferiore, appoggiata al piano di legno, esiste una guida entro cui far scorrere una o più
calamite. Un ulteriore componente dello strumento è costituito da sfere di ferro che rappresentano le
molecole della sonda da utilizzare per l’ibridazione.
La specificità di appaiamento mediante legame idrogeno tra il DNA della sonda e quello del gene
localizzato sul cromosoma viene simulato dal legame tra il metallo (sonda) e la calamita (DNA
complementare cromosomico) dovuto alla forza
magnetica.
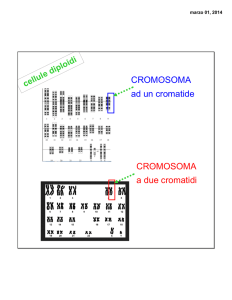
Localizzazione di un gene su un cromosoma
Uno studente posiziona 4 calamite sotto i cromatidi
fratelli di cromosomi omologhi
Un secondo studente mette le biglie magnetiche che
rappresentano diverse molecole della sonda A, a
localizzazione ignota, in contatto con il piano su cui sono
stati posizionati i cromosomi per stabilire la
localizzazione della sonda stessa e della sequenza di DNA che essa rappresenta.
Si simula l’incubazione con una quantità di sonda in eccesso, inclinando dolcemente più volte il
piano in modo tale che tutti i cromosomi possano venire coperti dalla sonda
Si elimina la sonda che è stata messa in eccesso per favorire l’ibridazione aprendo lo sportello ad un
lato del piano, inclinando il piano stesso dolcemente: le sfere non trattenute dalla forza magnetica
usciranno similmente le molecole presenti sui cromosomi e non legate mediante legame idrogeno
potranno essere selettivamente allontanate. Se non viene effettuato questo passaggio, che simula il
lavaggio nell’ibridazione in situ, la sonda depositata casualmente sui cromosomi non permette di
individuare il segnale specifico.
Si visualizza la localizzazione della sonda A sui cromatidi dei cromosomi omologhi (ibridazione
specifica) localizzandola poi più precisamente nell’ambito di regioni cromosomiche specifiche
Identificazione di anomalie cromosomiche sub-microscopiche
Traslocazione: Uno studente posiziona due calamite sotto i cromatidi fratelli di un cromosoma e
due sotto i cromatidi fratelli di un cromosoma non omologo. Un secondo studente mediante
l’ibridazione in situ (vedi sopra dal punto 2) utilizzando una sonda (sfere di metallo) a
localizzazione nota, da dichiarare, osserverà che la sequenza specifica è traslocata su un altro
cromosoma, diverso dall’omologo atteso
Delezione: Uno studente posiziona due calamite sotto i cromatidi fratelli di un cromosoma senza
posizionare la calamita nella stessa regione dei cromatidi del cromosoma omologo. Un secondo
studente mediante l’ibridazione in situ (vedi sopra dal punto 2) utilizzando una sonda a
localizzazione nota (sfere di metallo), da dichiarare, osserverà che la sequenza specifica è deleta su
uno dei due cromosomi omologhi
6
Duplicazione: Uno studente posiziona due calamite sotto i cromatidi fratelli di un cromosoma e 4
calamite sotto i cromatidi fratelli del cromosoma omologo. Un secondo studente mediante
l’ibridazione in situ (vedi sopra dal punto 2) utilizzando una sonda (sfere di metallo) a
localizzazione nota, da dichiarare, osserverà che la sequenza specifica è duplicata su uno dei due
cromosomi omologhi.
Studio della cinetica di ibridazione
Lo studente posiziona 4 calamite sotto i cromatidi fratelli di 2 cromosomi omologhi.
Lo studente mette a contatto con il piano numerose sfere di metallo e, inclinando più volte
dolcemente il piano, verifica quanto tempo occorre dall’inizio dell’oscillazione perchè le sfere si
leghino ai due cromatidi fratelli di entrambi i cromosomi omologhi
Lo studente toglie le sfere lasciandone solo quattro, cioè in rapporto molare 1:1 sufficiente per
saturare le possibilità di legame e, inclinando più volte dolcemente il piano, verifica quanto tempo
occorre dall’inizio dell’oscillazione perchè le sfere si leghino ai due cromatidi fratelli di entrambi i
cromosomi omologhi.
Potrà facilmente essere dedotto che la cinetica di ibridazione dipende dalla quantità di sonda posta a
contatto con i cromosomi poichè, in caso di eccesso di sonda, l’incontro tra molecole a sequenza
specifica, è statisticamente favorito.
7