![(Microsoft PowerPoint - Trascrizione [modalit\340 compatibilit\340])](//s1.studylibit.com/store/data/001111652_1-cde5f663b6961bc18d2a13ecc3b5ac78-768x994.png)
Trascrizione e maturazione degli RNA
L’RNA è sisntetizzato a partire dal DNA nel processo della trascrizione
L’RNA veicola l’informazione genica contenuta nel DNA (nucleo) in modo
che possa esprimersi :
•per dare proteine (mRNA)
•funzionare direttamente in attività correlate all’espressione genica/sintesi
proteica (tRNA e rRNA)
•svolgere funzioni varie (ribozimi, small RNA, micro RNA, …)
Enzima coinvolto: RNA polimerasi DNA-dipendente;
un tratto di filamento di DNA (“gene”) funziona da stampo
Vengono incorporati nella catena di RNA nascente dei ribonucleosidi
attivati in forma trifosfato (ATP, CTP, GTP, UTP) direzione 5’
3’
prima di niziare la trascrizione l’RNA polimerasi riconosce e lega un sito del
DNA: promotore
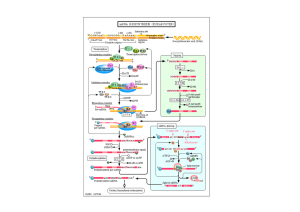
Trascrizione nei procarioti
•5’P
3’OH
•RNA polimerasi
Ponti fosfodiesterici
•Ribonucleosidi 5’-trifosfati
•Unità di trascriz.
L’una o l’altra delle 2 semieliche
•stampo
E. senso
2α
RNA polimerasi
core
1β
1 β’
PPi
Si aggiunge σ che riconosce un sito
a monte dell’inizio della trascr.
2 sequenze
Riconosce
promotore
(core
+
σ)
oloenzima
consenso
1 localizz. 35 nucleot. prima
Sequenze comuni a più geni
del sito di inizio (-35)
1 a (- 10)
Sito di apertura
Ricca di A T
Pribnow box
doppia elica
La polimerasi avanzando sul DNA
inserisce nucleotidi complementari
allo stampo (DNA ss) sulla catena
nascente di RNA (direzione 5’
3’)
La doppia elica DNA si riforma a
valle della RNA polimerasi (regione
ibrida RNA-DNA limitata a pochi
nucleotidi)
L’RNA resta associato all’enzima
durante tutta la trascrizione
Nei batteri la RNA polimerasi è di un solo
tipo ed è formata da varie subunità
proteiche
Il riconoscimento del promotore è mediato
dalla subunità σ
Il promotore presenta sequenze conservate
(consenso) a – 35 e -10 bp dal sito di inizio
di trascrizione
Fasi della trascrizione
procariotica
•Riconoscimento del promotore
•Inizio trascrizione ed
allungamento
•Terminazione determinata da
fattore ρ
dissociazione
dell’enzima dallo stampo e
dall’mRNA
•Nei batteri gli mRNA possono
essere policistronici: portano
informazione per più polipetidi
(o rRNA o tRNA9) sullo stesso
trascritto
…..trascrizione nei procarioti
Trascriz veloce nei tratti: (DNA) A
U (RNA)
(DNA) T
A (RNA)
Termine trascrizione
ATCG
Sequenze palindrome compl.
spontanea
CGAT
2 tipi di terminazione
TAGC
GCTA
Simmetria bipartita
Dipendente da proteina ρ
La terminazione può essere indotta da strutture stem
and loop che rallentano la RNA pol
In alcuni casi terminazione indipendente da fattore rho
A volte l’RNA pol ha bisogno di un attivatore per potersi legare al promotore
Maturazione dei trascritti procariotici
mRNA
maturazione quasi assente
traduzione contemporanea a trascrizione (solo
in Archea fenomeni di splicing)
tRNA e rRNA
maturazione mediante taglio di precursori
Segmento 5’ non tradotto
(da ~10 a più di 200 nucleotidi)
Segmento 3’ non tradotto
(da ~50 a più di 200 nucleotidi)
Regola la stabilità dei messaggeri.
Sequenza codificante tradotta
5’
3’
5’ cap
…….
AUG
Codon di inizio
T
...
AAUAAA
...
Coda di poli(A)
OH
Codon di terminazione
(UGA, UAA o UAG
(da ~30 a più di 200 nucleotidi)
Complessata con una proteina
che è vitale per la cellula.
Il CAP 5’:
1.
Facilità la fase di inizio
2.
E’ un segnale riconosciuto
dai complessi del poro
3.
Protegge gli mRNA dalle
ribonucleasi
Caratteristiche strutturali di un mRNA eucariotico. La coda di poli(A) non è presente in alcuni
mRNA eucariotici; la struttura cap 5’ non è presente in alcuni mRNA di virus che infettano cellule
eucariotiche.
NUCLEO
introni
Gene (DNA :doppio filamento)
AAUAAA
Sequenza di
poliadenilazione
Promotore
esoni
Inizio trascriz.
(RNA polimerasi)
Trascritto nucleare
(pre-mRNA, a singolo
filamento
5’
3’
AAAAAA…
Poliadenilazione:
aggiunta di molti
residui A
CAPPING: aggiunta di
7-metil guanosina
Splicing:
RNA messaggero
5’
3’
Leader
Trailer
5’
3’
Traduzione
CITOPLASMA
AAAAAA…..A
“Poly A”
AAAAAA…..A
Polisoma
Proteina nascente
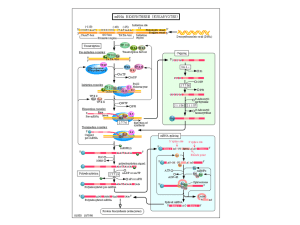
Negli eucarioti l’apparato di trascrizione è + complesso
3 enzimi che riconoscono promotori diversi e sintetizzano
classi diverse di RNA
RNA pol II è formata da un “core” (con attività polimerasica ) e da varie
subunità che agiscono di concerto per riconoscere il promotore, legare il
DNA e srotolarlo.
Sul promotore viene riconosciuta in particolare la seq TATA box
Altre sequenze a monte (anche 200 nt al 5’) possono attivare o inibire la RNA
pol mediante interazione con coattivatori o corepressori
Altre sequenze ancora più a monte possono attivare/inibire trascrizone (enhancer)
Es: il coattivatore CBP induce la
trascrizione di tutti i geni con seq
attivatrice CREB ( cAMP
responsive element)
Trascrizione di geni controllata
da livelli intracellulari di cAMP
Terminazione di trascrizione
RNA pol II ed altri fattori tagliano l’mRNA a valle del sito di
poliadenialzione
rilascio dell’mRNA, degradazione del segmento di RNA
3’ e dissociazione di RNA pol II dallo stampo
Maturazione dei trascritti eucariotici
trascritto primario o pre-mRNA (hnRNA)
•Capping
•Metilazione
•Poliadenilazione
•Splicing
•Editing
mRNA maturo
NUCLEO
La maturazione degli RNA è cotrascrizionale
Capping
L’aggiunta del CAP di metil guanosina (GMP) al 5’P dell’mRNA ne
protegge l’estremità evitando degradazione da parte di ribonucleasi e
lo posiziona correttamente sul ribosoma durante traduzione
Poliadenilazione
aggiunta di 30-200 adenosine (AMP) al 3’OH dell’mRNA
Stabilizza l’RNAm
La poliAdenilazione avviene a valle di sequenza specifica riconosciuta
da vari fattori, subito dopo il taglio che termina trascrizione
Splicing
Rimozione di sequenze prive di significato
(non codificanti) o introni e risaldatura degli
esoni
•I componenti dello splicesoma riconoscono
siti di giunzione esone/introne/esone
•Tagliano al 5’ per dare struttura a cappio
(lariat)
•Taglio al 3’ dell’introne eliminazione
e risaldatura tra i 2 esoni
Splicesoma = complesso di proteine e
snRNA
snRNP: small nuclear
ribonucleoprotein particle probabilmente
ribozimi
NB in alcuni casi si ha self-splicing introne è
ribozima
Esperimenti di associazione DNA-mRNA indicano la presenza di introni
RNA Editing
Cambiamento nella seq dell’mRNA a livello post-trascrizionale:
aggiunta-delezione di nt o cambiamento di basi azotate (U
C)
•Tipico di geni mitocondriali e di cloroplasti
•Richiede stampi di RNA guida (gRNA) originati da introni
Es editing tessuto specifico del mRNA per apoliporoteinaB (ApoB)
Solo gli mRNA maturi lasciano il nucleo
Maturazione degli rRNA eucariotici
•rRNA 28S, 18S, 5,8S derivano da singolo precursore trascritto da RNA pol I
gene ripetuto in tandem centinaia di volte nella zona NOR
zona fibrillare del nucleolo
, attivamente trascritta in alcune fasi della vita cellulare
•rRNA 5S invece trascritto da RNA pol III – cluster di geni ripetuti fuori dal nucleolo e
diverso precursore
Maturazione del precursore di rRNA 28S, 18S, 5,8S
•Modificazione di nucleotidi (metilazione e sostituzione di U con Ψ) mediata
da snoRNA
•Associazione con proteine ribosomali
•eliminazione degli spaziatori intragenici
Maturazione dei tRNA eucariotici
Trascritti da RNA pol III che riconosce promotore interno al gene (su
tratto codificante)
•geni organizzati in cluster
•Precursori con geni singoli sono accorciati al 5’ e 3’, subiscono
modificazione di basi ed eventuale splicing
•La maturazione dei pretRNA coinvolge il ribozima RNasiP
L’esportatore del t-RNA
dal nucleo verso il
citoplasma è
l’esportina-t.
Si lega pref. ai tRNA
maturi, aiutando così a
garantire che i tRNA non
maturi non siano esportati
prematuramente.
Addirittura i tRNA
vendono legati all’Aa
direttamente nel nucleo
CONCETTO DI GENE
•anni ’40: esperimenti di Beadle & Tatum
Si determina la relazione tra geni ed enzimi grazie a ricerche su alcuni ceppi della
muffa del pane (NEUROSPORA ) definiti «mutanti nutrizionali».
ALTERAZIONE DNA DIFETTO PROTEINA DIFETTO DI CRESCITA
Un gene un enzima
•Un gene
un polipeptide
Sono possibili varie definizione di gene!!
1) Il gene è una sequenza di DNA contenente l’istruzione per la produzione
di un polipeptide (fornisce cioè le istruzioni per la sintesi proteica). (questa
definizione non tiene conto dei geni per tRNA e rRNA)
2) Il gene è un unità trascrizionale (questa definizione esclude le seq regolatrici e
la possibilità che gene dia più prodotti mediante splicing e/o inizi/fine di
trascrizione alternativi)
3) Gene: segmento di DNA contenente un’informazione, non sempre univoca
4) Gene: unità ereditaria
Elementi fondamentali di un gene:
•promotore e regioni regolative,
•tratto codificante,
•terminatore di trascrizione
Negli eucarioti il tratto codificante è “interrotto” da introni!! genomi +ampi
Nei procarioti + geni sono trascritti insieme (
mRNA policistronici)
The sequence of a
prokaryotic proteincoding gene is colinear
with the translated mRNA;
that is, the transcript of
the gene is the molecule
that is translated into the
polypeptide.
The sequence of a
eukaryotic protein-coding
gene is typically not
colinear with the
translated mRNA; that is,
the transcript of the gene
is a molecule that must be
processed to remove extra
sequences (introns)
before it is translated into
the polypeptide.
Il codice genetico
Il flusso d’informazione si basa su un codice a triplette di basi: CODONI
le istruzioni che specificano la sequenza di amminoacidi di un polipeptide sono
scritte nel DNA (e sull’mRNA) come codoni
Seconda base azotata
C
A
UUU
UUC
UUG
Phe UCU
UCC
UCA
Leu
UCG
CUU
C CUC
CUA
CUG
CCU
Leu CCC
CCA
CCG
AUU
A AUC
AUA
ACU
ACC
U
Prima base azotata
Quasi tutti gli organismi (dai
batteri alle piante agli animali)
condividono lo stesso codice
genetico.
Tyr UGU
UGC
Cys U
C
UAA Stop
UGA Stop
A
UAG Stop
UGG Trp
CAU
CAC
His
CGU
CGC
G
U
CAA
CAG
CGA
Gln CGG
AAU
AAC
Asn AGU
AGC
Ser
ACA
Met o
AUG inizio ACC
AAA
AAG
Lys AGA
AGG
Arg A
G
GCU
GCC
GAU
GAC
Asp GGU
GGC
UUA
Il codice genetico è
universale!
Ser
UAU
UAC
G
GUU
GUC
G
GUA
GUG
Ile
Val
GCA
GCG
Pro
Thr
Ala
GAA
GAG
Arg
C
A
G
Glu GGA
GGG
U
C
U
Gly C
A
G
Terza base azotata
U
![(Microsoft PowerPoint - Trascrizione [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001111652_1-cde5f663b6961bc18d2a13ecc3b5ac78-768x994.png)