La mappatura dei geni umani
SCOPO
conoscere la localizzazione dei geni
per identificarne la struttura e la funzione
Un grande impulso alla costruzione di mappe genetiche
è stato dato da
• le tecniche della citogenetica
visualizzazione della morfologia dei cromosomi
• le tecniche della fusione cellulare
suddivisione del corredo cromosomico
• le tecniche molecolari
identificazione di marcatori polimorfici
Le mappe genetiche permettono di identificare
geni anche quando non si hanno informazioni
sulla loro funzione
L’approccio detto “Clonaggio posizionale” ha
contribuito a caratterizzare le basi genetiche di
molte malattie genetiche
Le mappe genetiche si basano sull’analisi di
ricombinazione tra marcatori genetici alla
MEIOSI
Che cos’è un MARCATORE
• una caratteristica fenotipica
• una particolarità biochimica
• una sequenza del DNA
Tipi di marcatori genetici
TIPO
N DI LOCI
SAGGIO
Gruppi sanguigni
Circa 20
immunologico
Proteine sieriche
Circa 30
elettroforesi
RFLP
>105
PCR
Minisatelliti
>104
PCR
Microsatelliti
>104
PCR
Un MARCATORE è
un qualsiasi carattere genetico che possa essere
distinto da individuo a individuo
deve perciò corrispondere ad un gene o ad una
sequenza di DNA che abbia almeno due
ALLELI
DUE marcatori genetici alla MEIOSI
se sono su cromosomi diversi
seguono la legge dell’assortimento indipendente
se sono sulla stessa porzione del genoma
tendono ad essere co-ereditati
in questo caso sono detti associati o linked
Analisi di linkage
L’analisi di linkage non richiede che si conosca la
funzione del gene e le mappe genetiche possono
essere costruite anche per geni conosciuti solo per i
loro effetti fenotipici
Analisi di linkage
Se vogliamo stabilire se il gene di una malattia
genetica di cui ignoriamo le basi biochimiche è
associato ad un marcatore A
dovremo esaminare molte famiglie
in cui compare la malattia
Non tutte le famiglie sono informative
Queste famiglie NON SONO informative
La figlia ha ereditato la malattia dominante dal padre
A1 A1 A2 A2
A1 A2
A1 A2 A1 A2
A1 A2
ma gli alleli del marcatore del
padre non possono essere distinti
tra di loro
ma gli alleli del marcatore del
padre non possono essere
distinti da quelli della madre
Queste famiglie SONO informative
A1 A1 A2 A2
A1 A2 A3 A4
A1 A1
A1 A4
La figlia ha ereditato l’allele A1 del marcatore
dal padre affetto INSIEME con la malattia
ma questo non basta per dire che i due loci sono
associati o linked
Marcatori polimorfici
L’esempio ci fa capire che la probabilità di avere
famiglie informative dipende
dal NUMERO e dalla FREQUENZA degli alleli
dei marcatori
cioè dal grado di eterozigosità della popolazione
per ciascun marcatore
Marcatori polimorfici
N di alleli
Frequenza
Grado di
eterozigosità
2
0,5-0,5
0,50
2
2
0,4-0,6
0,3-0,7
0,48
0,42
2
0,2-0,8
0,32
4
tutti 0,25
0,75
10
tutti 0,1
0,90
Se DUE marcatori genetici sono associati
la probabilità di essere co-ereditati
dipende dalla loro distanza
più sono vicini minore è la probabilità
che alla meiosi vengano separati dalla
RICOMBINAZIONE
Le mappe genetiche
si basano sulla misura della distanza tra i marcatori
in base alla frequenza di ricombinazione
è necessario poter distinguere i gameti
parentali dai gameti ricombinanti
In assenza di ricombinazione: gameti parentali
A
B
1
2
A
1
oppure
B
2
In caso di ricombinazione: gameti ricombinanti
A
B
1
2
A
2
oppure
B
1
La FASE
A
1
B
2
In questa meiosi si riconoscono i
ricombinanti perchè si conosce la fase
Il marcatore A è in cis rispetto al marcatore 1
e in trans rispetto al marcatore 2
APLOTIPI parentali
A
1
B
2
APLOTIPI ricombinanti
A
2
B
1
Negli incroci sperimentali si possono riconoscere i
gameti parentali dai gameti ricombinanti proprio
dalla loro frequenza
Se i marcatori sono associati i gameti parentali
saranno molto più frequenti dei gameti ricombinanti
Nell’Uomo il numero di figli del singolo incrocio
è troppo piccolo per distinguere i gameti parentali
dai ricombinanti se non si conosce la FASE
Una meiosi può essere non informativa se non
conosciamo la fase degli alleli
Aa
Bb
aa
bb
?
Aa
Bb
Aa
Bb
aa
Bb
Si può stabilire la fase
in una famiglia di tre generazioni
In
questo
modo
aa
possiamo
bb
riconoscere
Aa
Bb
Aa
Bb
aa
bb
uo
d
i
v
i
d
l’in
Aa
Bb
Aa
Bb
aa
Bb
te
n
a
in
b
m
o
c
i
r
L’unità di scala delle mappe genetiche
Il centimorgan (cM) equivale ad
una frazione di ricombinazione θ= 0.01
cioè un gamete su 100 è ricombinante
Fisicamente un cM corrisponde approssimativamente
a 0,7-1 Mb ma la relazione tra
distanze genetiche e distanze fisiche è variabile
Distanze genetiche
Negli organismi sperimentali si può esaminare la
progenie di un singolo incrocio o di molti incroci
geneticamente programmati in modo da misurare
accuratamente la frazione di ricombinazione
Nell’Uomo si fa uso dell’analisi del LOD score
un metodo introdotto da Morton nel 1955
Analisi del LOD score
Permette di utilizzare i dati ottenuti da molte
famiglie informative
Attraverso l’analisi di ciascuna famiglia si stima
la probabilità
che due loci siano associati in corrispondenza di
un dato valore della frazione di ricombinazione θ
CONTRO
l’ipotesi che non siano associati (quindi θ = 0,5)
Calcolo del LOD score
A2 A5
A1 A6
A1 A2
A3 A4
R
A1 A3
A2 A3
A1 A4
A1 A4 A2 A4
A2 A3
Tutte le meiosi sono di fase nota perciò senza
ambiguità si può desumere che
solo il sesto figlio è ricombinante
Calcolo del LOD score
se il locus di malattia è associato al marcatore
la probabilità di osservare questa distribuzione è
p = θ (1- θ)5
se il locus di malattia NON è associato al marcatore
la probabilità è
p = (0,5)6
Calcolo del LOD score
Il LOD score è un punteggio che rappresenta il
logaritmo del rapporto tra le due probabilità
Zx= log10 Probabilità che il risultato osservato se θ = x
Probabilità che il risultato osservato se θ = 0,5
nel caso della famiglia esaminata
Zx= log10 [θ (1- θ)5/(0,5)6]
Calcolo del LOD score
Si esplora l’intervallo da 0 a 0,5
FR
0
0,1
Z
-∞
0,58 0,62 0,51 0,23
0,2
0,3
0,4
0,5
0
Z è una funzione di x
che assume un massimo per un determinato valore
della frazione di ricombinazione
in questo caso 0,2
Calcolo del LOD score
A1 A2
A1 A3
A2 A3
A1 A4
A3 A4
A1 A4 A2 A4
A2 A3
La stessa famiglia è assai meno informativa quando
la fase non è nota perchè manca il genotipo dei nonni
Calcolo del LOD score
Il calcolo deve tener conto anche della possibilità
che il sesto figlio sia l’unico NON ricombinante
Zx= log10 [1/2 θ (1- θ)5 + 1/2 θ 5(1- θ) ] /(0,5)6
FR
0
0,1
0,2
0,3
0,4
0,5
Z
-∞
0,28
0,32
0,22
0,076 0
Il valore di θ per cui si ottiene Zmax è lo stesso
ma il valore di Z è circa la metà di prima
Analisi del LOD score
Il valore di θ che individua il massimo della funzione
Z sarà la distanza stimata tra i marcatori
Un valore di Z =3 è considerato statisticamente
significativo per stabilire un linkage tra due loci
Raramente dall’analisi di una sola famiglia si può
raggiungere la soglia di significatività
La probabilità complessiva di linkage in un
insieme di famiglie è il prodotto delle
probabilità in ciascuna famiglia
Le probabiltità calcolate con il LOD score sono
espresse in log
quindi i valori di Z possono essere sommati
Nella applicazione che abbiamo visto abbiamo
analizzato solo due marcatori
un locus di malattia dominante ed
un marcatore polimorfico A
ma il metodo può essere usato per analizzare
molti loci contemporaneamente
se è nota la localizzazione del marcatore in
linkage con il locus di malattia questo si troverà
nelle sue vicinanze nell’intervallo definito dalla
frazione di ricombinazione
Teoricamente bastano cinque famiglie come
quella esaminata per avere un valore
significativo di linkage
In pratica le famiglie sono molto meno
numerose e meno informative per cui è
necessario avere un insieme consistente di
famiglie da esaminare
Nell’esempio abbiamo visto un calcolo
semplificato per il caso di un locus di malattia
DOMINANTE
Il metodo si applica anche all’analisi di linkage
per le malattie RECESSIVE ma il calcolo è più
complicato perchè in molti casi si ignora il
genotipo dei non affetti
sono perciò necessarie molte più famiglie
Le famiglie CEPH
In alcuni gruppi etnici le famiglie hanno tipicamente
un gran numero di figli ed
accurati registri dei matrimoni
Sono state selezionate le famiglie con almeno tre
generazioni viventi
Sono stati stabilite linee cellulari da campioni dei
quattro nonni, dei genitori ed almeno sei figli
Le famiglie CEPH
Le mappe genetiche di un gran numero di marcatori
sono state costruite usando queste
famiglie di riferimento
La presenza di tre generazioni permette di ricostruire
la fase dei marcatori
La segregazione nei sei figli permette di analizzare le
frequenze di ricombinazione tra i marcatori
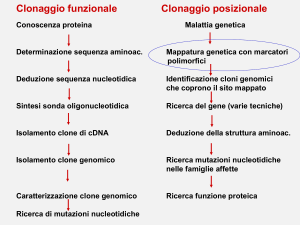
Strategie per identificare geni-malattia
Il difetto interessa una proteina a funzione nota
Determinazione della sequenza amminoacidica
Deduzione della sequenza nucleotidica
Sintesi di una sonda oligonucleotidica
Isolamento di un clone di cDNA
Selezione di un clone genomico
Caratterizzazione del clone genomico
Identificazione del gene
Caratterizzazione delle mutazioni
Strategie per identificare geni-malattia
Analisi dei sintomi della malattia
Diverse proteine note possono essere
coinvolte nel difetto genetico
Scelta del o dei geni candidati
Caratterizzazione delle mutazioni
Ricerca delle mutazioni nei pazienti
Identificazione del gene
Strategie per identificare geni-malattia
Malattia genetica
Mappatura genetica su un locus
Isolamento dei cloni di DNA genomico che ricoprono il locus
Identificazione e analisi delle ORF e degli esoni
Isolamento di un clone di cDNA
Selezione di un clone genomico
Caratterizzazione del clone genomico
Caratterizzazione delle mutazioni
Strategie per identificare geni-malattia
Malattia genetica
Mappatura genetica su un locus
Identificazione e analisi dei geni funzionali
e delle EST mappate nello stesso locus
Isolamento di un clone di cDNA
Selezione di un clone genomico
Caratterizzazione del clone genomico
Caratterizzazione delle mutazioni