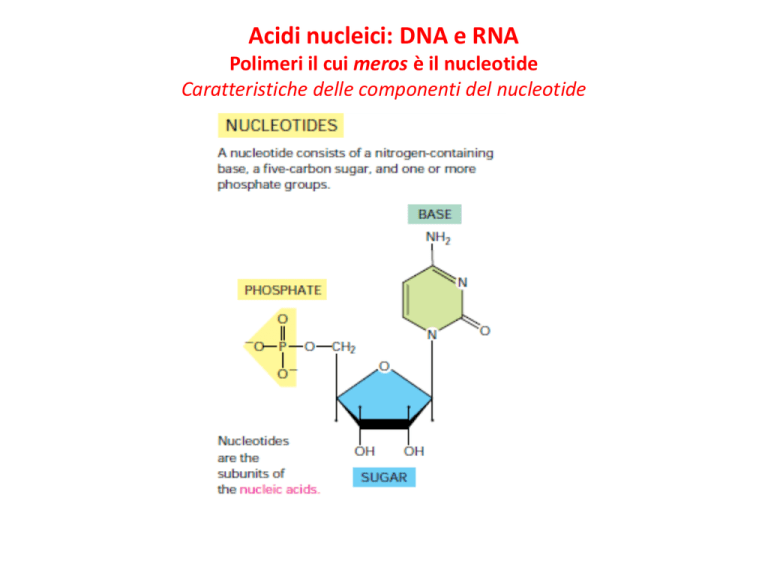
Acidi nucleici: DNA e RNA
Polimeri il cui meros è il nucleotide
Caratteristiche delle componenti del nucleotide
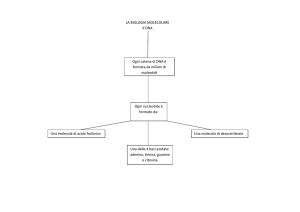
Acidi nucleici: DNA e RNA
Polimero il cui meros è il nucleotide
Caratteristiche delle componenti del nucleotide
ATP
La molecola di ATP è un esempio di nucleotide. Si può
descrivere graficamente con diverse modalità.
In acqua i fosfati
dell’ATP sono carichi
negativamente
~
Acidi nucleici: DNA
Polimero il cui meros è il nucleotide
Caratteristiche delle componenti del nucleotide
POLIMERIZZAZIONE DEI NUCLEOTIDI
Il legame fosfodiesterico è il legame
covalente che si forma in una catena
polinucleotidica (come il DNA o l’RNA) tra il
fosfato associato al carbonio in posizione 5’
dello zucchero di un nucleotide e il
carbonio in posizione 3’ dello zucchero del
nucleotide precedente. Si tratta di un
legame di condensazione.
Acidi nucleici: il DNA (acido deossiribonucleico)
Il DNA è costituito da due catene nucleotidiche complementari e
antiparallele. Ognuna ha una testa rappresentata da 5’ e una coda
rappresentata da 3’.
Ogni catena deriva dalla polimerizzazione di singoli nucleotidi legati fra loro da
legami covalenti. E’ il fosfato in posizione 5’ del secondo nucleotide che si lega
al C in posizione 3’ dello zucchero del primo nucleotide. Quando la catena è
completa il primo nucleotide avrà la posizione 5’ libera (testa 5’) dove rimane
il gruppo fosfato e l’ultimo avrà il carbonio 3’ libero da legami (coda 3’).
Caratteristiche delle componenti del nucleotide
Le due catene antiparallele del DNA sono tenute insieme da ponti
idrogeno, due fra A e T e tre fra G e C.
Acidi nucleici: DNA
Chargaff’s rules (1952). The first rule holds
that a double-stranded DNA molecule globally
has percentage base pair equality: %A = %T and
%G = %C. The rigorous validation of the rule
constitutes the basis of Watson-Crick pairs in
the DNA double helix.
The second rule holds that both %A ~ %T and
%G ~ %C are valid for each of the two DNA
strands. This describes only a global feature of
the base composition in a single DNA strand.
La percentuale dei quattro tipi di nucleotidi è
sempre la stessa nel DNA di cellule provenienti
da tessuti diversi del medesimo individuo.
I regola di Chargaff
Nel DNA la quantità totale delle purine (A+G) è pari a quella delle
pirimidine (T+C).
II regola di Chargaff
In una molecola di DNA la %A=%T e la %G=%C. Questo ha suggerito
come si realizzasse l’appaiamento tra le basi.
STRUTTURA DEL DNA
Photo 51 is the
nickname given to an
X-ray diffraction image
of DNA taken by
Rosalind Franklin in
1952 that was critical
evidence in identifying
the structure of DNA.
La cristallografia ai raggi X ha contribuito a rivelare la struttura elicoidale del
DNA. La posizione degli atomi in una sostanza chimica cristallizzata può essere
determinata in base al quadro di diffrazione dei raggi X che l’hanno attraversata. Il
quadro del DNA è estremamente regolare e ripetitivo.
Poiché la lunghezza d’onda dei raggi X (compresa
tra 0.1 e 10 angstrom) è dello stesso ordine di
grandezza delle distanze atomiche dentro la
materia, la diffrazione a raggi X permette di
definire come sono sistemati gli atomi nel
campione analizzato.
STRUTTURA DEL DNA
James Watson (L) and Francis Crick (R),
and the model they built of the structure
of DNA.
In 1953, James Watson and Francis Crick
developed the model for deoxyribonucleic
acid (DNA) a chemical that had (then)
recently been deduced to be the physical
carrier of inheritance. Crick hypothesized
the mechanism for DNA replication and
further linked DNA to proteins, an idea
since referred to as the central dogma.
Information from DNA "language" is
converted into RNA (ribonucleic acid)
"language" and then to the "language" of
proteins.
STRUTTURA DEL DNA
Il DNA è costituito da una doppia
elica di filamenti complementari e
antiparalleli.
Gli zuccheri e i fosfati dei nucleotidi
formano l’impalcatura esterna,
mentre le basi azotate si proiettano
verso l’interno, stabilendo legami
H. Le basi adiacenti di ogni
filamento stanno una sopra all’altra
su piani paralleli.
DNA Structure - YouTube
Dogma centrale della biologia molecolare, o più
semplicemente dogma centrale, è un principio secondo il quale il
flusso dell’informazione genetica è monodirezionale: parte dal
DNA, che viene convertito in RNA, per arrivare alle proteine.
Esempio di eccezione al dogma
centrale: retrovirus
FORMA PRIONICA
PROTEINA NORMALE
Esempio di eccezione al dogma centrale:
prioni (PRoteinaceus Infective ONly particle,
particella infettiva solamente proteica)
IL PRIONE CONTATTA LA
LA PROTEINA NORMALE
PROTEINA NORMALE
VIENE CONVERTITA A PRIONE
DNA (acido desossiribonucleico) e
RNA (acido ribonucleico)
differiscono in una base azotata…
... e nello zucchero che nel DNA è desossiribosio e nell’RNA è ribosio
DEFINIZIONE DI GENE
Il gene è tutta la sequenza nucleotidica necessaria per la
formazione di un RNA funzionale.
Le basi azotate dei nucleotidi che costituiscono il DNA e cioè A, T, C, G
formano le lettere di un codice, il codice genetico, che viene conservato
nel DNA e che può essere decodificato e tradotto in proteine.
Il DNA però non partecipa
direttamente alla decodificazione. In
essa è implicata una catena di acido
nucleico a lui complementare, l’RNA
messaggero (mRNA), prodotto
attraverso la TRASCRIZIONE.
TRASCRIZIONE
Processo con il quale l’RNA polimerasi, utilizzando una
catena stampo di DNA, polimerizza una catena di RNA
complementare.
mRNA
AUG
_
UAC
_
GGC
_
ACA
_
UUC
Il codice genetico è, quindi, riportato nel linguaggio dell’RNA
messaggero. Il codice si legge in triplette.
Ma da quanto tempo ci è noto che il codice
si legge in triplette, dette codoni di informazione?
George Gamow
In the years after 1953,
scientists
scrambled to be the first to
decipher the genetic code. In
an attempt to make the race
interesting, theoretical
physicist and astronomer
George Gamow came up with
a plan. He organized an
exclusive club, the “RNA Tie
Club,” in which each member
would put forward ideas as to
how the nucleotide bases
were translated into proteins
in the body's cells. His club
had twenty hand-picked
members, one for each amino
acid, and each wore a tie
marked with the symbol of that
amino acid. The group—which
did not include Marshall
Nirenberg—met several times
during the 1950s but did not
manage to be the first to break
the code.
The scientists who pondered the mystery
of the genetic code in the late 1950s
came up with many creative theories.
One main problem to work out was how
many bases would be in each code word
(later known as a codon). They knew
there was a total of four bases (guanine,
cytosine, adenine, and thymine). So if
there were two bases in each codon,
then there would only be the possibility
of (4 x 4) sixteen unique combinations of
bases, or sixteen amino acids. But there
were 20 known amino acids. So most
assumed there would be at least three
bases in each codon, providing (4 x 4 x 4)
64 possible combinations. However,
despite all the scientists working on the
problem, in 1961 the basic mystery
remained: which series of bases specified
which amino acids? What, in fact, was
the genetic code?
In a laboratory on the seventh floor of
Building 10 on the NIH campus in Bethesda,
Maryland, Marshall Nirenberg –by now an
employee of NIH–and his post doctoral fellow
Heinrich Matthaei were hard at work on the
coding problem by 1960.
Heinrich Matthaei. e
Marshal Niremberg
Their experiment required a cell-free system, created when cell walls are ruptured and release their
contents. The material inside cells is called cytoplasm, or sap, which can still synthesize protein but
only when the correct kind of RNA is added, allowing the scientists to control the experiment.
Nirenberg and Matthaei chose E. Coli bacteria cells, and ground them up using a mortar and pestle
to release the cytoplasm, or sap, they would use in their experiments. The scientists, working by
themselves for the most part, and often late into the night, would use the sap itself to force the
creation of a protein. The experiment used 20 test tubes, each filled with a different amino acid. For
each individual experiment, 19 test tubes were “cold” and one was radioactively tagged so the
scientists could watch the reaction. The “hot” amino acid would change every time they did the
experiment. Nirenberg wanted to know which amino acid would be incorporated into a protein
following the addition of a particular type of synthetic RNA.
On Saturday, May 27, 1961, at three o'clock in the morning, Matthaei combined the synthetic RNA
made only of uracil (called poly-U) with cell sap derived from E. coli bacteria and added it to each of
20 test tubes. This time the “hot” test tube was phenylalanine. The results were spectacular and
simple at the same time: after an hour, the control tubes showed a background level of 70 counts,
whereas the hot tube showed 38,000 counts per milligram of protein. The experiment showed that a
chain of the repeating bases uracil forced a protein chain made of one repeating amino acid,
phenylalanine. The code could be broken! UUU=Phenylalaline was a breakthrough experiment result
for Nirenberg and Matthaei.
The two kept their breakthrough a secret from the larger scientific community–though many NIH
colleagues knew of it –until they could complete further experiments with other strands of synthetic
RNA (Poly-A, for example) and prepare papers for publication. They had solved with an experiment
what others had been unable to solve with theoretical explanations and mathematical models.
It was only a few months before the discovery made Nirenberg a celebrity. In August, at the
International Congress of Biochemistry in Moscow, he presented his paper to a small group. One of
the listening scientists convinced the conference leaders to invite Nirenberg to repeat his
performance. Speaking before the assembled congress of more than a thousand people, Nirenberg
electrified the scientific community. Within months, his picture appeared in magazines around the
world and he was a highly sought-after lecturer.
Nirenberg's method of testing synthetic RNA in a cell-free system was a key technical innovation.
However, once this technique for decoding the relationship of mRNA to amino acids was publicly
announced in 1961, there was still much work to do before the code was deciphered. The scientists
had to determine which bases made up each codon, then determine the sequence of bases in the
codons. This represented an enormous amount of work.
At the same time, Nobel laureate Severo Ochoa was busy working on the coding problem in his own
laboratory at New York University Medical School. Ochoa had a big staff, and Nirenberg was worried
he would not be able to keep up. By the end of 1961, Matthaei had completed his fellowship and
moved back to Germany. But NIH came to Nirenberg's side to help. Faced with the possibility of
helping the first NIH scientist win a Nobel prize, many NIH scientists put aside their own work to help
Nirenberg in deciphering the mRNA codons for amino acids. Dr. DeWitt Stetten, Jr., director of the
National Institute of Arthritis and Metabolic Diseases, called this period of collaboration “NIH's finest
hour.” All in all, more than 20 people came through Nirenberg's laboratory. He needed scientists at
all levels, postdoctoral fellows, and laboratory technicians to assist him in his important work
LA DECIFRAZIONE DEL CODICE GENETICO
Nirenberg e Matthaei usarono un sistema di sintesi in vitro per
determinare gli amminoacidi specificati da mRNA sintetici di composizione
conosciuta.
CONCLUSIONI: UUU è un codone di mRNA per la fenilalanina, AAA per la lisina,
CCC per la prolina, GGG per la glicina.
This period, between 1961 and 1962, is often referred
to as the “coding race” because of the competition
between Ochoa's and Nirenberg's labs. Indeed, the
two laboratories completed the base composition part
of the code almost simultaneously. However, Ochoa’s
laboratory stopped working on the problem when they
realized how close Nirenberg and his colleagues
were to completing the sequencing.
For his landmark work on the genetic code, Nirenberg was awarded the 1968
Nobel Prize in Physiology or Medicine. He shared the award with Har Gobind
Khorana of the University of Wisconsin and Robert W. Holley of the Salk
Institute. Working independently, Khorana had mastered the synthesis of
nucleic acids, and Holley had discovered the exact chemical structure of
transfer-RNA.
NIH was particularly proud of Nirenberg's success because he was the first
NIH intramural scientist to win the Nobel Prize. Some had doubted that the
government could produce innovative and internationally recognized
scientists, but Nirenberg and his colleagues proved them wrong.
IL CODICE GENETICO SI LEGGE A TRIPLETTE
Il codone di inizio (start codon) è AUG. La metionina è l’unico amminoacido
specificato da un solo codone.
I codoni di stop (stop codons) sono UAA, UAG e UGA non specificano
nessun amminoacido. Il ribosoma si ferma e rilascia l’mRNA.
La porzione di codoni compresa tra AUG e un codone di STOP è chiamata
open reading frame (ORF).
Il codice è degenerato: gli amminoacidi sono specificati da più di un codone
che generalmente differisce solo nella terza posizione.
La "Wobble Hypothesis”, scoperta da Francis Crick, stabilisce che
l’appaiamento delle basi è rilassato e può oscillare in corrispondenza della
terza posizione, in maniera tale che una base del codone possa essere
riconosciuta da più basi dell’anticodone.
Alcuni anticodoni dei tRNA hanno l’inosina (I) in corrispondenza della terza
posizione. L’inosina può appaiarsi con U, C o A, ma non con G. Ciò significa
che non sono necessari 61 diversi tRNA (uno per ciascun codone), ma sono
sufficienti circa la metà.
RNA di trasferimento
(RNA transfer, tRNA)
La TRADUZIONE, coinvolge la conversione delle quattro basi
del codice (ATCG) in venti differenti amminoacidi. Un codone, o
tripletta di basi, specifica un dato amminoacido. La maggior parte
degli amminoacidi sono specificati da più di un codone.
La conversione dell’informazione a codoni in proteine è realizzato
da un RNA di trasferimento (tRNA), che funziona da adattatore.
Ciascun tRNA ha un anticodone con il quale può appaiarsi al
codone. Alcuni anticodoni hanno basi modificate che possono
appaiarsi con più di un codone, specificante lo stesso
amminoacido. Pertanto non occorrono 61 molecole di tRNA
diverse per il riconoscimento dei 61 codoni.
tRNA structure
Complementarietà e
antiparallelismo
nell’appaiamento tra
codoni dell’mRNA e
anticodoni dei tRNA
Poiché il codice genetico è
degenerato, la terza base è meno
discriminatoria per l’aminoacido
delle altre due basi. Questa 3°
posizione viene detta posizione di
Wobble.
Oscillazione della 3° base
(effetto Wobble)
L’accuratezza della traduzione dipende dalla fedeltà con la
quale i corretti amminoacidi sono esterificati alla loro
appropriata molecola di tRNA.
(A)
La fedeltà è dovuta all’enzima aminoaciltRNA sintetasi, uno per ciascun
amminoacido.
L’amminoacil-tRNA sintetasi catalizza la
formazione di un legame estere tra gruppo
carbossilico di un amminoacido e
l’estremità 3’ OH dell’appropriato tRNA in
due step chimici:
(1) L’amminoacido e una molecola di ATP
entrano nel sito attivo dell’enzima.
Simultaneamente l’ATP perde il
pirofosfato (P-P) e il risultante AMP si
lega covalentemente all’amminoacido.
Il pirofosfato è idrolizzato a due P.
(2) Il
tRNA
lega
covalentemente
l’amminoacido “scalzando” l’AMP.
L’amminoacil-tRNA viene poi liberato
dall’enzima.
Dal
linguaggio
amminoacidico…
nucleotidico
a
quello
La sequenza amminoacidica di una proteina corrisponde
alla sequenza delle triplette del DNA portate nel citoplasma
come codoni sull’mRNA.
mRNA
Il codice si legge a triplette (codoni): 3 nucleotidi codificano per 1 aminoacido
Met
Val
Arg
Tyr
43 possibili combinazioni:
64 codoni, di cui
61 sono codificanti e
3 sono codoni di STOP (codoni non senso)
AUG: codone di inizio (Met)
Il codice genetico è degenerato (ridondante): più triplette
codificano per uno stesso amminoacido, ma non è mai ambiguo
(una tripletta codifica per un solo amminoacido).
Il codice genetico è non sovrapposto: nessun nucleotide fa
parte contemporaneamente di più di un codone.
Il codice genetico è senza spaziature.
Il codice genetico è universale, ovvero è lo stesso in tutti gli
esseri viventi.
Esistono eccezioni: mitocondri, dove, per es., UGA è il
codone per il triptofano anziché essere una tripletta di
terminazione.
Il DNA contiene, quindi
l’informazione per la sintesi
di tutte le proteine. Il DNA,
che nella sua conformazione
a doppia catena è molto
stabile, si apre per dare
l’informazione
che
è
contenuta nella sequenza
delle sue basi in due
processi: la replicazione e la
trascrizione.
TRASCRIZIONE
Il processo con il quale utilizzando una catena stampo di DNA viene
polimerizzata con l’aiuto dell’enzima RNA polimerasi una catena di RNA
complementare.
Nei procarioti la regione di controllo è posta fra il nucleotide 10 e il nucleotide 35 a
monte del primo nucleotide da trascrivere. Questa regione (promotore) viene
riconosciuta dalla unità sigma (fattore s) della RNA polimerasi. Il riconoscimento
genera un’interazione fra enzima e DNA che favorisce l’apertura della doppia elica.
La regione posta intorno alla posizione –10 è denominata Pribnow box e presenta
frequentemente la sequenza consenso TATAAT. Intorno alla posizione –35 c’è
un’altra sequenza consenso, spesso rappresentata da TTGAC.
5’
3’
3’
5’
Negli eucarioti le regioni di controllo della trascrizione possono essere collocate
anche molto lontano dal primo nucleotide da trascrivere e possono essere sia a
monte che a valle del gene. La regione di controllo prossimale più frequente è a
circa 30 nucleotidi a monte del trascritto e contiene la sequenza TATA (TATA box)
Negli eucarioti la trascrizione inizia con il
posizionamento, nella regione di
controllo, di fattori di trascrizione. Solo
successivamente entra in gioco la RNA
polimerasi II.
TRADUZIONE
Perché l’informazione scritta nella catena di RNA complementare al DNA
possa trasformarsi in proteina è necessario che il messaggio venga tradotto.
Per effettuare la traduzione sono necessarie tre tipi di RNA: l’RNA di
trasferimento (tRNA), l’RNA messaggero (mRNA) e l’RNA ribosomiale (rRNA).
RIBOSOMI DEI PROCARIOTI E DEGLI EUCARIOTI
Il ribosoma ha tre siti che
interagiscono con il tRNA e un
sito legante l’mRNA
INIZIO della traduzione di una proteina con
sequenza: met-arg-gly-ser-pro-thr-
Traduzione: fase di ALLUNGAMENTO
Traduzione: fase di ALLUNGAMENTO
Traduzione: fase finale (TERMINE)
Codoni di stop: UAG, UAA, UGA
Riassunto delle varie tappe della traduzione
Translation
http://bcs.whfreeman.com/lodish5e/pages/bcsmain.asp?v=category&s=00010&n=04000&i=04010.03&o=|00510|00520|00530|
00540|00560|00570|00590|00600|00700|00010|00020|00030|00040|00050|0100
0|02000|03000|04000|05000|06000|07000|08000|09000|10000|11000|120
INIZIO NEI PROCARIOTI
Sequenza di Shine-Dalgarno: è ricca di purine e viene riconosciuta
dall’rRNA della subunità minore dei ribosomi.
INIZIO NEGLI EUCARIOTI
Sequenza consenso di
Kozac: una delle più
frequenti sequenze che
precedono il sito di inizio
AUG.









![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)