Orario: lunedì, martedì, mercoledì, venerdì
dalle 14:00 alle 16:00
Aula 3A
Ricevimento studenti: martedì 12.00-14.00
Docente: Prof. Silvia Morante
Dipartimento di Fisica
Corridoio D1
Interno: 4554
e-mail: [email protected]
http://people.roma2.infn.it/~morante/
http://biophys.roma2.infn.it/
Tutti gli studenti
sono pregati di inviarmi una e-mail contenente:
nome, cognome e indirizzo elettronico.
Chi non avesse un indirizzo elettronico è pregato di
fornire un recapito telefonico
Presentazione Laurea Magistrale:
Indirizzo di Fisica dei Biosistemi
Aula Grassano
Lunedì 5 Ottobre
Ore 16:20 – 17:00
Laurea Magistrale Indirizzo di
XAS
Dal genoma:
… ACU UUC CGU AAC…
Alla sequenza proteica:
… THR PHE ARG ASN…
DNA
a
iment
...sper
amino
acidi
amino
acidi
ASN
ARG
PHE
THR
stato
unfolded
folding
intermedio
stato
nativo
Struttura e funzione della proteina
...teor
ici
li
Programma svolto di Fisica Biologica 1
A.A. 2008-2009
Introduzione
•Definizione di sistema vivente. Viroidi e Plasmidi. Virus e Batteri.
Protozoi. Organismi superiori.
•RNA interference. Silencing e regolazione genica. I micro-RNA e i
viroidi.
•Fisica e Biologia. Teoria e modelli. Riduzionismo e complessità.
Definizione di complessità e sistema vivente
•Definizione di sistema complesso: numerosità e variabilità.
Classificazioni e alberi filogenetici.
•L’albero filogenetico e l’eredità “orizzontale”.
•Le macromolecole biologiche: acidi nucleici, proteine e membrane.
•Livelli di struttura: primaria, secondaria, terziaria e quaternaria.
L’evoluzione
•L’origine del sistema solare: l’ipotesi della nebulosa di Laplace e il problema
del momento angolare.
•Teoria dell’accumulazione e regolarità.
•L’evoluzione della Terra: atmosfera ossidante e atmosfera riducente.
•L’esperimento di Miller.
•La datazione con isotopi radioattivi.
•La protocellula di Oparin.
•Archeoabatteri ed estremofili.
•Il concetto di compartimentazione e la complessità biologica.
La cellula
•Cellule procariotiche e cellule eucariotiche.
•Gli organelli cellulari. Mitocondri e cloroplasti.
•La divisione cellulare: mitosi e meiosi.
•Ereditarietà e leggi di Mendel.
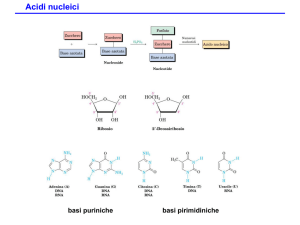
Gli acidi nucleici
•Biopolimeri. La chimica degli acidi nucleici: purine, pirimidine e legame
fosfodiesterico.
•La duplicazione del DNA. Variabilità genetica e SNP’s.
•La trascrizione: nei procarioti e negli eucarioti.
•La trascrizione: gli introni e il controllo dell’espressione genica. L’operone
LAC.
•La traduzione: l’RNA transfer e il ribosoma.
•Sistemi di controllo per la traduzione e bilancio energetico. Gli enzimi di
restrizione
•Tecniche di sequenziamento degli acidi nucleici.
•Contenuto informativo del genoma: la legge di Zipf.
•Il problema di Hamilton e il DNA computing.
•Legge di Zipf e invarianti di scala.
•Entropia relativa e similarità tra sequenze.
•Sequenze segnale e probabilità di occorrenza di oligonucleotidi,
•Processi di Markov. Teorema di Bayes e sua estensione al continuo.
•Pressione selettiva e abbondanza o rarità di oligonucleotidi: CTAG, sequenze
palindrome e siti di riconoscimento degli enzimi di restrizione.
Le proteine
•Il primo livello strutturale: sequenza aminocaidica.
•Gli amino acidi e il legame peptdico.
•La chimica degli amino acidi. Catene laterali e proprietà chimico fisiche.
•Definizione di pK. Curve di titolazione e punto isolettrico.
Programma svolto di Fisica Biologica 2
A.A. 2008-2009
• Il sequenziamento di proteine. Elettroforesi, cromatografia,
centrifugazione.
•Allineamento: Dot Plot; autoallineamento e ripetizione; programmazione
dinamica
•Matrici di sostituzione: matrici PAM e mutabilità.
•Il modello di Eigen: definizione di iperciclo e sua modellizzazione
matematica; analisi di punto fisso; soluzione numerica di esempi concreti
•Il sistema immunitario: cellule B e cellule T.
• Il sistema immunitario: anticorpi; il complesso MHC.
• Mimesi molecolare e malattie autoimmuni.
•Determinanti epitopici e individuazione di sequenze “sufficientemente”
mimetiche.
•Le funzioni termodinamiche. L’energia libera di Gibbs e la
spontaneità della reazione.
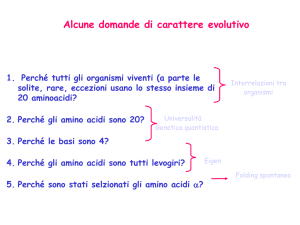
•Evoluzione e tipi di selezione: 4 basi; 20 amino acidi; amino acidi
levogiri; alpha-amino acidi.
•Il folding spontaneo e gli alpha-amino-acidi
•La struttura secondaria: alpha-elica e beta-foglietto.
•Idropaticità e DeltaG di trasferimento
•Profili di idropaticità e spettri di potenza. Anfifilicità (esempio:
alpha-tossina e miosina)
•Struttura terziaria: l’esperimento di Anfinsen. Esempi di struttura
terziaria.
•Il concetto di idrofobicità e il modello di Kauzmann.
•Potenziale chimico e funzioni di stato specifiche molari. Contributo
cratico e contributo molare.
•Il potenziale torzionale. Approssimazione di geometria rigida. I
grafici di Ramachandran.
•Interazioni di van der Waals. Il potenziale alla Lennard-Jones12-6.
•Il minimo dell’energia conformazionale: il simulated annealing.
•Le membrane biologiche: i lipidi. Micelle, liposomi e black membranes. I
Langmuir-Blodgett. I lipid rafts.
•Le proteine di membrana: caratteristiche generali e tipi di interazioni con la
membrana.
•Interazione radiazione-materia. Tecniche spettroscopiche in biologia: limiti e
potenzialità. La legge di Beer-Lambert, significato fisico del coefficiente
d’estinzione.
•Richiami di meccanica quantistica: teoria delle perturbazioni e sezioni d’urto.
Esempio: molecola neutra e momento di dipolo della transizione.
•Spettroscopia di assorbimento ai raggi X. Apparato sperimentale e rivelatori.
•Approssimazione di singolo scattering. Estrazione dei dati strutturali: chi(k)
e filtro di Fourier.
• Introduzione del multiplo scattering mediante i segnali irriducibili a n corpi.