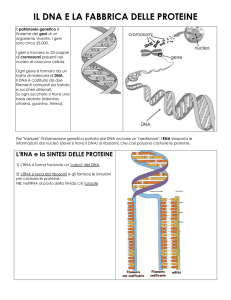
Espressione ed utilizzo
della
informazione genetica
Prof.ssa Flavia Frabetti
Aa 2010-11
L’informazione genetica, contenuta nel DNA, ha lo scopo di:
1) mantenere lo stato vitale (strutture e funzioni)
2) realizzare l’adattamento (risposta ad un cambiamento)
3) determinare il differenziamento (specializzazione di strutture e
funzioni)
L’informazione genetica ereditata determina il manifestarsi
di specifici caratteri inducendo la sintesi di certe proteine:
le proteine sono l’anello di congiunzione tra informazione
e la realizzazione/effettuazione di strutture ed attività.
1
ESPRESSIONE DELLA INFORMAZIONE BIOLOGICA
non solo le strutture, ma anche l’architettura e la distribuzione
spaziale di queste, nonché i cambiamenti temporali
DNA
dal
progetto
alla
costruzione
proteina
Cosa studiamo?
Dove è scritta l’informazione genica:
DNA
Cromatina / cromosomi
Nucleo eucariotico
Come è organizzata l’informazione genica:
Genoma
Geni - come concetto
- “struttura del gene eucariota tipo”
Come si esprime l’informazione genica:
Trascrizione e Traduzione
Codice genetico
Meccanismi di CONTROLLO della espressione genica
2
Dove è scritta la informazione?
nucleo
cromosoma
telomero
centromero
telomero
proteine acide
istoni
DNA
Come è organizzata l’informazione?
Una enorme quantità di informazione
dentro ad ogni singola cellula:
il GENOMA
Aprendo ogni libro si trovano brani così scritti:
…..ATCCGAGCTTTACGTACGGTTACCGGATCGAGCATACT……
Anche la definizione di genoma è complessa!
Il genoma è:
- il complesso dell'informazione genetica di una
cellula
- la massa totale del DNA cellulare
- il patrimonio ereditario dell'organismo a cui
appartiene
3
COMPLESSITA’ DEL GENOMA
Servono informazioni proporzionali
alla complessità dell’organismo ?
Proporzionalità da batteri a vermi
Poi non viene mantenuta
PARADOSSO DELLA NON
PROPORZIONALITA’
TRA COMPLESSITA’ GENOMA E
COMPLESSITA’ ORGANISMO
Paradosso
del valore “C”
DIMENSIONI in bp
(contenuto in DNA)
NUMERO DEI GENI
Media:
Minimo:
Massimo:
Drosophila M.
16.321
C. Elegans
21.210
61.710
27.462
153.478
Arabidobsis T.
26.000
Homo sapiens
STIMA: 22.000
(analisi computer)
4
Organizzazione generale
del genoma umano
Il genoma umano è distribuito in cromosomi:
22 tipi di AUTOSOMI
2 tipi di ETEROCROMOSOMI (X e Y)
Corredo aploide 3.200.000.000 bp ovvero 3,2 Gbp
(Cr. 1 279.000.000 bp, Cr. 21 45.000.000 bp )
Genoma mitocondriale 16.569 bp
Peso (genoma diploide) Lunghezza (genoma diploide) 7 pg
2m
ORGANIZZAZIONE DEL GENOMA UMANO
NUCLEARE
~ 40% GENI
CODIFICANTI
PROTEINE
E GENI PER ncRNA
MITOCONDRIALE
0,5 %
~ 60%
DNA EXTRAGENICO
5
ORGANIZZAZIONE DEL GENOMA UMANO
NUCLEARE
MITOCONDRIALE
0,5 %
1/3 GENI
CODIFICANTI
PROTEINE
5,3% mRNA MATURO
(ESONI)
30 Mb
1,7 % DEL GENOMA
56% CODIFICANTE (CDS)
0,96 % DEL GENOMA
replicazione o duplicazione
DNA
trascrizione
della
informazione
RNA
proteine
traduzione
della
informazione
Reintrepetazione
del dogma della biologia molecolare alla
luce delle nuove conoscenze sul genoma
6
Ai geni che codificano proteine si affiancano geni
che specificano per RNA non codificanti utili alla
regolazione della espressione genica.
Stime odierne:
Geni umani codificanti per proteine sono un numero
di 20.000-25.000
Geni per RNA non tradotti o noncoding RNA (ncRNA, RNA non
codificanti proteine), nei mammiferi
rappresentano da 1/2 a 3/4 di tutti i
trascritti
La maggior parte del genoma dei
mammiferi è trascritta
Il 98% dell’output trascrizionale nei mammiferi è
rappresentato da RNA che non verrà tradotto in aa
Known or
predicted
transcribed
7Mb
100Mb 130Mb 2.6Gb
2.9Gb
Human
Mouse
Fruitfly
Worm
Yeast
58%
55%
60%
59%
70%
Noncoding
percent of
transcription
98%
98%
71%
56%
0.6%
7
La complessità del genoma si riflette sulla espressione
dell’informazione genica e sulla definizione stessa di
gene
Geni:
definizione/concetto
struttura
regolazione
I geni sono le
unità responsabili
delle caratteristiche
ereditarie
Il gene è
una regione di DNA
trascritta che contiene
istruzioni per la sintesi
di una proteina, di un
RNA o ignota
8
Il gene è una delle tante istruzioni contenute in ogni cellula
FUNZIONI
DEI GENI
METABOLISMO
INFORMAZIONE GENETICA
STRUTTURA
SEGNALI
FUNZIONI TESSUTO-SPECIFICHE
22%
25%
21%
12%
20%
GENI PER RNA NON TRADOTTI
(rRNA 85%, tRNA 10%, RNA non-codificanti)
Ci possono essere:
1) Geni essenziali o geni housekeeping
Geni che devono essere espressi praticamente in tutti i tipi cellulari
poiché codificano prodotti utili al funzionamento generale delle cellule,
per es. alla sintesi proteica o alla produzione di energia
2) Geni con limitazioni spazio-temporali nella espressione:
SPAZIO
Espressione diversa
in diversi organi e tessuti
TEMPO
Stadio del ciclo cellulare
Specificità per tessuto,
tipo cellulare
Stadio dello sviluppo
Singole cellule (specificità di
cellula es. Ab)
Stadio del differenziamento
Distribuzione intracellulare
Espressione inducibile
9
Gene eucariota - la struttura nasconde complessità
Promotore
Gene strutturale
regione trascritta
I geni eucarioti hanno
una struttura discontinua
1977
Ibrido molecolare DNA-mRNA
10
GENE - Struttura del gene eucariota tipo
Il gene ha una natura discontinua:
esoni tratti del gene indicati con E
introni tratti del gene indicati con I
E1
E2
I1
E3
I2
+1
Tratto di DNA che viene trascritto o TRASCRITTO PRIMARIO
ESONI (in giallo), intervallate da lunghi INTRONI (in grigio)
DNA
E1
I1
E2
E3
I2
+1
Esone 1
TRASCRITTO
PRIMARIO o
pre-mRNA
Esone 3
AUG
mRNA
Proteina
Esone 2
UGA
AUG
NH2
UGA
COOH
11
Dimensione media geni
57.000 bp (57 kb)
istoni
100-400 bp (0,1-0,4 kb)
distrofina 2.220.000 bp (2,22 Mb)
ESONI
Numero medio 11
(minimo)-(massimo) 1 - 363
Dimensione media
280 bp
INTRONI
Dimensione media ca. 6.000 bp
TTAGCACTACCGTATTTGCGCATTACCAGATTAGAGAA
ATGCTAGTCGATCTATCGATCGGCTATTCGCAAAGCTG
CGCGACTGCGATGCGCTAGCATGCGATTCGCGATCGCC
GAGCGCTCGCGAGCGCGCTAGCGGAATACTATATAGC
GCGGATCAGTCTAGATCTATGAGATCGATAGCGATCTA
GAGATAGGATCGAGATCGAGGCGAGATCATATGAGCG
CGGCTATTTAGGCTTAGAGGATTCGGAGATTCGGAGCT
TAGGATTACAGAGAGCTTCTTAGGCGCTCCCGGTATCG
CTCCCATCCCATATTAAAATCTATCGATCGAGCTCTCCA
ATGCGATCGATAGGACTAGTAGCTAGCTAGCTGAGCA
TGATAGGCTCGATGAGCATGAGATGCATGTACGACTG
CATAGGCATGACTGATCGACTGCATCATGACGCATGAC
TGCATGCATGACTGCATATGACGGACTCGCATTAGCAC
TACCGTATTTGCGCATTACCAGATTAGAGAAATGCTAG
TCGATCTATCGATCGGCTATTCGCAAAGCTGCGCGACT
GCGATGCGCTAGCATGCGATTCGCGATCGCCGAGCGC
TCGCGAGCGCGCTAGCGGAATACTATATAGCGCGGAT
CAGTCTAGATCTATGAGATCGATAGCGATCTAGAGATA
GGATCGAGATCGAGGCGAGATCATATGAGCGCGGCTA
TTTAGGCTTAGAGGATTCGGAGATTCGGAGCTTAGGAT
TACAGAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCA
TCCCATATTAAAATCTATCGATCGAGCTCTCCAATGCG
ATCGATAGGACTAGTAGCTAGCTAGCTGAGCATGATA
GGCTCGATGAGCATGAGATGCATGTACGACTGCATAG
GCATGACTGATCGACTGCATCATGACGCATGACTGCAT
GCATGACTGCATATGACGGACTCGCATTAGCACTACCG
TATTTGCGCATTACCAGATTAGAGAAATGCTAGTCGAT
CTATCGATCGGCTATTCGCAAAGCTGCGCGACTGCGAT
GCGCTAGCATGCGATTCGCGATCGCCGAGCGCTCGCG
AGCGCGCTAGCGGAATACTATATAGCGCGGATCAGTC
TAGATCTATGAGATCGATAGCGATCTAGAGATAGGATC
GAGATCGAGGCGAGATCATATGAGCGCGGCTATTTAG
GCTTAGAGGATTCGGAGATTCGGAGCTTAGGATTACA
GAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCATCCC
ATATTAAAATCTATCGATCGAGCTCTCCAATGCGATCG
ATAGGACTAGTAGCTAGCTAGCTGAGCATGATAGGCT
CGATGAGCATGAGATGCATGTACGACTGCATAGGCAT
GACTGATCGACTGCATCATGACGCATGACTGCATGCAT
GACTGCATATGACGGACTCGCA
GENE
Nella realtà gli INTRONI
sono molto più lunghi
degli esoni
mRNA maturo
(in media)
Dimensione
3.000 basi
Sequenza codificante 1.600 basi (56%)
Proteina
540 AA
TTAGCACTACCGTATTTGCGCATTACCAGATTAGAGAA
ATGCTAGTCGATCTATCGATCGGCTATTCGCAAAGCTG
CGCGACTGCGATGCGCTAGCATGCGATTCGCGATCGCC
GAGCGCTCGCGAGCGCGCTAGCGGAATACTATATAGC
GCGGATCAGTCTAGATCTATGAGATCGATAGCGATCTA
GAGATAGGATCGAGATCGAGGCGAGATCATATGAGCG
CGGCTATTTAGGCTTAGAGGATTCGGAGATTCGGAGCT
TAGGATTACAGAGAGCTTCTTAGGCGCTCCCGGTATCG
CTCCCATCCCATATTAAAATCTATCGATCGAGCTCTCCA
ATGCGATCGATAGGACTAGTAGCTAGCTAGCTGAGCA
TGATAGGCTCGATGAGCATGAGATGCATGTACGACTG
CATAGGCATGACTGATCGACTGCATCATGACGCATGAC
TGCATGCATGACTGCATATGACGGACTCGCATTAGCAC
TACCGTATTTGCGCATTACCAGATTAGAGAAATGCTAG
TCGATCTATCGATCGGCTATTCGCAAAGCTGCGCGACT
GCGATGCGCTAGCATGCGATTCGCGATCGCCGAGCGC
TCGCGAGCGCGCTAGCGGAATACTATATAGCGCGGAT
CAGTCTAGATCTATGAGATCGATAGCGATCTAGAGATA
GGATCGAGATCGAGGCGAGATCATATGAGCGCGGCTA
TTTAGGCTTAGAGGATTCGGAGATTCGGAGCTTAGGAT
TACAGAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCA
TCCCATATTAAAATCTATCGATCGAGCTCTCCAATGCG
ATCGATAGGACTAGTAGCTAGCTAGCTGAGCATGATA
GGCTCGATGAGCATGAGATGCATGTACGACTGCATAG
GCATGACTGATCGACTGCATCATGACGCATGACTGCAT
GCATGACTGCATATGACGGACTCGCATTAGCACTACCG
TATTTGCGCATTACCAGATTAGAGAAATGCTAGTCGAT
CTATCGATCGGCTATTCGCAAAGCTGCGCGACTGCGAT
GCGCTAGCATGCGATTCGCGATCGCCGAGCGCTCGCG
AGCGCGCTAGCGGAATACTATATAGCGCGGATCAGTC
TAGATCTATGAGATCGATAGCGATCTAGAGATAGGAT
CGAGATCGAGGCGAGATCATATGAGCGCGGCTATTTA
GGCTTAGAGGATTCGGAGATTCGGAGCTTAGGATTAC
AGAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCATCC
CATATTAAAATCTATCGATCGAGCTCTCCAATGCGATC
GATAGGACTAGTAGCTAGCTAGCTGAGCATGATAGGC
TCGATGAGCATGAGATGCATGTACGACTGCATAGGCA
TGACTGATCGACTGCATCATGACGCATGACTGCATGCA
TGACTGCATATGACGGACTCGCA
Nel “libro”
DNA,
le pagine
sono scritte in
codice/i
12
TTAGCACTACCGTATTTGCGCATTACCAGATTAGAGAA
ATGCTAGTCGATCTATCGATCGGCTATTCGCAAAGCTG
CGCGACTGCGATGCGCTAGCATGCGATTCGCGATCGCC
GAGCGCTCGCGAGCGCGCTAGCGGAATACTATATAGC
GCGGATCAGTCTAGATCTATGAGATCGATAGCGATCTA
GAGATAGGATCGAGATCGAGGCGAGATCATATGAGCG
CGGCTATTTAGGCTTAGAGGATTCGGAGATTCGGAGCT
TAGGATTACAGAGAGCTTCTTAGGCGCTCCCGGTATCG
CTCCCATCCCATATTAAAATCTATCGATCGAGCTCTCCA
ATGCGATCGATAGGACTAGTAGCTAGCTAGCTGAGCA
TGATAGGCTCGATGAGCATGAGATGCATGTACGACTG
CATAGGCATGACTGATCGACTGCATCATGACGCATGAC
TGCATGCATGACTGCATATGACGGACTCGCATTAGCAC
TACCGTATTTGCGCATTACCAGATTAGAGAAATGCTAG
TCGATCTATCGATCGGCTATTCGCAAAGCTGCGCGACT
GCGATGCGCTAGCATGCGATTCGCGATCGCCGAGCGC
TCGCGAGCGCGCTAGCGGAATACTATATAGCGCGGAT
CAGTCTAGATCTATGAGATCGATAGCGATCTAGAGATA
GGATCGAGATCGAGGCGAGATCATATGAGCGCGGCTA
TTTAGGCTTAGAGGATTCGGAGATTCGGAGCTTAGGAT
TACAGAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCA
TCCCATATTAAAATCTATCGATCGAGCTCTCCAATGCG
ATCGATAGGACTAGTAGCTAGCTAGCTGAGCATGATA
GGCTCGATGAGCATGAGATGCATGTACGACTGCATAG
GCATGACTGATCGACTGCATCATGACGCATGACTGCAT
GCATGACTGCATATGACGGACTCGCATTAGCACTACCG
TATTTGCGCATTACCAGATTAGAGAAATGCTAGTCGAT
CTATCGATCGGCTATTCGCAAAGCTGCGCGACTGCGAT
GCGCTAGCATGCGATTCGCGATCGCCGAGCGCTCGCG
AGCGCGCTAGCGGAATACTATATAGCGCGGATCAGTC
TAGATCTATGAGATCGATAGCGATCTAGAGATAGGATC
GAGATCGAGGCGAGATCATATGAGCGCGGCTATTTAG
GCTTAGAGGATTCGGAGATTCGGAGCTTAGGATTACA
GAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCATCCC
ATATTAAAATCTATCGATCGAGCTCTCCAATGCGATCG
ATAGGACTAGTAGCTAGCTAGCTGAGCATGATAGGCT
CGATGAGCATGAGATGCATGTACGACTGCATAGGCAT
GACTGATCGACTGCATCATGACGCATGACTGCATGCAT
GACTGCATATGACGGACTCGCA
GENE
1
TTAGCACTACCGTATTTGCGCATTACCAGATTAGAGAA
ATGCTAGTCGATCTATCGATCGGCTATTCGCAAAGCTG
CGCGACTGCGATGCGCTAGCATGCGATTCGCGATCGCC
GAGCGCTCGCGAGCGCGCTAGCGGAATACTATATAGC
GCGGATCAGTCTAGATCTATGAGATCGATAGCGATCTA
GAGATAGGATCGAGATCGAGGCGAGATCATATGAGCG
CGGCTATTTAGGCTTAGAGGATTCGGAGATTCGGAGCT
TAGGATTACAGAGAGCTTCTTAGGCGCTCCCGGTATCG
CTCCCATCCCATATTAAAATCTATCGATCGAGCTCTCCA
ATGCGATCGATAGGACTAGTAGCTAGCTAGCTGAGCA
TGATAGGCTCGATGAGCATGAGATGCATGTACGACTG
CATAGGCATGACTGATCGACTGCATCATGACGCATGAC
TGCATGCATGACTGCATATGACGGACTCGCATTAGCAC
TACCGTATTTGCGCATTACCAGATTAGAGAAATGCTAG
TCGATCTATCGATCGGCTATTCGCAAAGCTGCGCGACT
GCGATGCGCTAGCATGCGATTCGCGATCGCCGAGCGC
TCGCGAGCGCGCTAGCGGAATACTATATAGCGCGGAT
CAGTCTAGATCTATGAGATCGATAGCGATCTAGAGATA
GGATCGAGATCGAGGCGAGATCATATGAGCGCGGCTA
TTTAGGCTTAGAGGATTCGGAGATTCGGAGCTTAGGAT
TACAGAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCA
TCCCATATTAAAATCTATCGATCGAGCTCTCCAATGCG
ATCGATAGGACTAGTAGCTAGCTAGCTGAGCATGATA
GGCTCGATGAGCATGAGATGCATGTACGACTGCATAG
GCATGACTGATCGACTGCATCATGACGCATGACTGCAT
GCATGACTGCATATGACGGACTCGCATTAGCACTACCG
TATTTGCGCATTACCAGATTAGAGAAATGCTAGTCGAT
CTATCGATCGGCTATTCGCAAAGCTGCGCGACTGCGAT
GCGCTAGCATGCGATTCGCGATCGCCGAGCGCTCGCG
AGCGCGCTAGCGGAATACTATATAGCGCGGATCAGTC
TAGATCTATGAGATCGATAGCGATCTAGAGATAGGAT
CGAGATCGAGGCGAGATCATATGAGCGCGGCTATTTA
GGCTTAGAGGATTCGGAGATTCGGAGCTTAGGATTAC
AGAGAGCTTCTTAGGCGCTCCCGGTATCGCTCCCATCC
CATATTAAAATCTATCGATCGAGCTCTCCAATGCGATC
GATAGGACTAGTAGCTAGCTAGCTGAGCATGATAGGC
TCGATGAGCATGAGATGCATGTACGACTGCATAGGCA
TGACTGATCGACTGCATCATGACGCATGACTGCATGCA
TGACTGCATATGACGGACTCGCA
ccctgtggag ccacacccta gggttggcca atctactccc aggagcaggg agggcaggag
61
ccagggctgg gcataaaagt cagggcagag ccatctattg cttacatttg cttctgacac
121
aactgtgttc actagcaacc tcaaacagac accATGgtgc acctgactcc tgaggagaag
181
tctgccgtta ctgccctgtg gggcaaggtg aacgtggatg aagttggtgg tgaggccctg
241
ggcaggttgg tatcaaggtt acaagacagg tttaaggaga ccaatagaaa ctgggcatgt
301
ggagacagag aagactcttg ggtttctgat aggcactgac tctctctgcc tattggtcta
361
ttttcccacc cttaggctgc tggtggtcta cccttggacc cagaggttct ttgagtcctt
421
tggggatctg tccactcctg atgctgttat gggcaaccct aaggtgaagg ctcatggcaa
481
gaaagtgctc ggtgccttta gtgatggcct ggctcacctg gacaacctca agggcacctt
541
tgccacactg agtgagctgc actgtgacaa gctgcacgtg gatcctgaga acttcagggt
601
gagtctatgg gacccttgat gttttctttc cccttctttt ctatggttaa gttcatgtca
661
taggaagggg agaagtaaca gggtacagtt tagaatggga aacagacgaa tgattgcatc
721
agtgtggaag tctcaggatc gttttagttt cttttatttg ctgttcataa caattgtttt
781
cttttgttta attcttgctt tctttttttt tcttctccgc aatttttact attatactta
841
atgccttaac attgtgtata acaaaaggaa atatctctga gatacattaa gtaacttaaa
901
aaaaaacttt acacagtctg cctagtacat tactatttgg aatatatgtg tgcttatttg
961
catattcata atctccctac tttattttct tttattttta attgatacat aatcattata
Cromosoma 12
1021 catatttatg ggttaaagtg taatgtttta atatgtgtac acatattgac caaatcaggg
1081 taattttgca tttgtaattt taaaaaatgc tttcttcttt taatatactt ttttgtttat
1141 cttatttcta atactttccc taatctcttt ctttcagggc aataatgata caatgtatca
1201 tgcctctttg caccattcta aagaataaca gtgataattt ctgggttaag gcaatagcaa
1261 tatttctgca tataaatatt tctgcatata aattgtaact gatgtaagag gtttcatatt
1321 gctaatagca gctacaatcc agctaccatt ctgcttttat tttatggttg ggataaggct
1381 ggattattct gagtccaagc taggcccttt tgctaatcat gttcatacct cttatcttcc
1441 tcccacagct cctgggcaac gtgctggtct gtgtgctggc ccatcacttt ggcaaagaat
1501 tcaccccacc agtgcaggct gcctatcaga aagtggtggc tggtgtggct aatgccctgg
1561 cccacaagta tcacTAAgct cgctttcttg ctgtccaatt tctattaaag gttcctttgt
1621 tccctaagtc caactactaa actgggggat attatgaagg gccttgagca tctggattct
1681 gcctaataaa aaacatttat tttcattgca atgatgtatt taaattattt ctgaatattt
1741 tactaaaaag ggaatgtggg aggtcagtgc atttaaaaca taaagaaatg atgagctgtt
1801 caaaccttgg gaaaatacac tatatcttaa actccatgaa agaaggtgag gctgcaacca
1861 gctaatgcac attggcaaca gcccctgatg cctatgcctt attcatccct cagaaaagga
1921 ttcttgtaga ggcttgattt gcaggttaaa gttttgctat gctgtatttt acattactta
1981 ttgttttagc tgtcctcatg aatgtctttt cactacccat ttgcttatcc tgcatctctc
2041 tcagccttga ct
Gene della beta-globina (Homo sapiens)
da 1 a 2052 nucleotidi
Sequenza GenBank n. NM_000518
DNA a filamento doppio (l’altro si ricava per complementarità)
13
acatttg cttctgacac
aactgtgttc actagcaacc tcaaacagac accATGgtgc acctgactcc tgaggagaag
tctgccgtta ctgccctgtg gggcaaggtg aacgtggatg aagttggtgg tgaggccctg
ggcaggctgc tggtggtcta cccttggacc cagaggttct ttgagtcctt tggggatctg
mRNA maturo
per la beta-globina
tccactcctg atgctgttat gggcaaccct aaggtgaagg ctcatggcaa gaaagtgctc
ggtgccttta gtgatggcct ggctcacctg gacaacctca agggcacctt tgccacactg
agtgagctgc actgtgacaa gctgcacgtg gatcctgaga ctcctgggca acgtgctggt
ctgtgtgctg gcccatcact ttggcaaaga attcacccca ccagtgcagg ctgcctatca
gaaagtggtg gctggtgtgg ctaatgccct ggcccacaag tatcacTAAg ctcgctttct
tgctgtccaa tttctattaa aggttccttt gttccctaag tccaactact aaactggggg
atattatgaa gggccttgag catctggatt ctgcctaata aaaaacattt at
proteina
beta-globina,
catena beta della
emoglobina
e mioglobina
Gene e sua funzione: Cosa fa accendere il gene?
A “sfumare l’oggetto” gene e renderlo più un “CONCETTO” sta
la parte regolatrice del gene
SEQUENZE
TRASCRIVIBILI
GENE
PROMOTORE
SEQUENZE
REGOLATRICI
INTENSIFICATORI
SILENZIATORI
UNITA’ DI TRASCRIZIONE
Tratto di DNA che codifica per una molecola
di RNA e delle sequenze necessarie per la sua
trascrizione
14
Gene networks - un gene può esprimere le proprie
informazioni solo nel contesto dell’intero genoma
della cellula
DNA
Promot 1
gene1
Promot 2
gene2
Promot 3
-
gene3
Promot 4
gene4
+
FATTORI DI
TRASCRIZIONE
RNA
proteine
Come si esprime l’informazione?
Per i geni classici vedremo:
I meccanismi di Trascrizione e Traduzione
Cosa è il Codice genetico
I principali meccanismi di CONTROLLO della espressione genica
15
"DNA makes RNA, RNA makes protein, and proteins make us."
Francis Crick
replicazione o duplicazione
DNA
trascrizione
della
informazione
RNA
proteine
traduzione
della
informazione
Criteri:
tempestività
economia
ovvero sintetizzare le proteine giuste al momento giusto
e nella giusta quantità
1. Trascrizione
tRNA
rRNA
2. Traduzione
mRNA
Sintesi delle proteine
16
TRASCRIZIONE (eucarioti)
“assemblaggio catalizzato”
Nella trascrizione l’informazione genetica contenuta
nel DNA viene trascritta in una sequenza di RNA in
base al principio della complementarietà delle basi
è un processo controllato, regolato
poiché le necessità sono:
1- riconoscimento del gene da esprimere
2- individuazione di inizio e termine del gene specifico
3- trascrizione nella giusta quantità del gene
4- alto grado di fedeltà, cioè accuratezza
17
La trascrizione genera diversi tipi di RNA:
rRNA o RNA ribosomiale
gene
tRNA o RNA transfer o RNA di
trasporto
proteina in crescita
mRNA o RNA messaggero
ribosomi
HnRNA o RNA eterogeneo nucleare o
pre-RNA
snRNA o piccoli RNA non codificanti
aa legato
al tRNA
Il processo è realizzato da enzimi chiamati
RNA polimerasi DNA-dipendenti
l’enzima si lega ad una sequenza sul DNA detta promotore
Negli eucarioti:
RNA polimerasi I
rRNA : 28 S, 18 S, 5.8 S
RNA polimerasi II
precursori degli mRNA,
ncRNA, diversi snRNA
RNA polimerasi III
tRNA e rRNA 5 S e
alcuni snRNA
18
TRASCRIZIONE
PROMOTORE
Promotore
RNA
polimerasi
Direzione della trascrizione
RNA polimerasi
TRASCRIZIONE:
fasi
iniziazione
allungamento
terminazione
RNA polimerasi
19
ORIENTAMENTO DELLE UNITA’
filamento CODING
o SENSO
3'
5'
5'
3'
3'
5'
RNA POLIMERASI
filamento STAMPO
o TEMPLATE
o ANTI SENSO
VELOCITA’
ca. 40 nucleotidi al secondo
Es. gene distrofina lungo ca. 2.200.000 bp???
Quante ore per trascriverlo?
17 ore!
Complementarietà delle basi
filamento non-senso serve da stampo
5'
basi
5'
3'
filamento senso
Entrambi i filamenti del DNA possono costituire stampo
per la sintesi dell’RNA, in funzione del gene, saranno sempre
letti in direzione 3'- 5', poiché la crescita del filamento di RNA
di neosintesi è in direzione 5'-3'
20
Meccanismo della trascrizione:
sintesi di RNA sulla base di uno stampo di DNA
5'
Enzima: RNA polimerasi
5'
Trascrizione:
lo stampo di DNA è letto da 3' a 5',
l’RNA è sintetizzato da 5' a 3'
3'
3'
2° nucleotide
Uscita del pirofosfato
Formazione legame
fosfodiesterico
1° nucleotide
1° nucleotide
5'
CODICE GENETICO o
Codice di traduzione (1964)
Definizioni
Codice = sistema di segnali, o segni, o simboli,
che, per convenzione, è destinato a rappresentare
una informazione tra la fonte dei segnali e il
punto di destinazione.
Es. comunicare in codice/ c. linguistico,
formato da suoni/ c.grafico, c.fiscale,
c. a barre, c.morse, ecc.
Codice genetico
sequenza di codoni contenenti le informazioni
genetiche del gene e determinanti la sequenza
degli aa che origina la proteina in base alla
lettura dei codoni stessi
21
Codice genetico è un codice a triplette di nucleotidi, detti codoni
codice genetico
anni ‘60
I codoni specificano per gli aa. In totale 43=64 codoni.
Caratteristiche del codice genetico
• il codice è ridondante
• il codice non è ambiguo
• il codice non ha punteggiatura ovvero interruzioni
• il codice è letto senza sovrapposizioni
• per interpretarlo è fondamentale la cornice di lettura o quadro di
lettura (reading frame)
• il codice è universale (ad eccezione di quello dei mitocondri)
Cornice di
lettura
ACCAAACCG
DNA
UGGUUUGGC
mRNA
Trp Phe Gly
proteina
22
TRADUZIONE
“assemblaggio codificato”
TRASCRIZIONE
TRADUZIONE
Nella traduzione l’informazione genetica contenuta nella
sequenza di codoni lungo l’mRNA viene decodificata o
tradotta in una sequenza di aa costituenti la proteina, uniti
in una sequenza precisa determinata dalla sequenza dei
codoni
Protagonisti principali di questo processo:
porta nel citoplasma
il messaggio genetico
sotto forma di una
specifica sequenza di
codoni
mRNA
tRNA
ribosomi
sono gli interpreti
del linguaggio
facilitano l’appaiamento
specifico tra gli anticodoni
del tRNA e i codoni
dell’mRNA
23
Ribosomi (60% rRNA con azione
catalizzante+proteine)
subunità
minore
mRNA
subunità
maggiore
1. Hanno un sito di legame per l’mRNA.
2. Un sito P (peptidil-tRNA) che ospita il tRNA che porta la catena
aminoacidica in allungamento.
3. Un sito A (aminoacil-tRNA) che ospita il tRNA a cui è legato il
successivo aa da aggiungere.
4. Un sito E di uscita del tRNA scarico di aa.
tRNA
sito di attacco
per gli aa
(sempre CCA)
legami H tra
basi complementari
anticodone
24
Attacco
dell’aa specifico
in 3'OH del tRNA
Aminoacil-tRNA
sintetasi
L’enzima riconosce
l’anticodone e lega
l’aa opportuno
L’aminoacil-tRNA
ha un aa “attivato”
Riconoscimento
codone-anticodone
aminoacido
ANTICODONE
tRNA = adattatore
U G A
CODONE
tripletta di nucleotidi
codificante per 1 aa
25
Met
Inizio sintesi proteica
Per individuare il sito di INIZIO
della TRADUZIONE: Sequenza
consenso di M.Kozak: R--AUGG,
R sta per una purina A o G
5’
Met Ser
AUG
3’
mRNA
Allungamento sintesi proteica
Met- Ser Pro
Met- Ser-Pro-Thr
5’
3’
mRNA
Terminazione sintesi proteica
NH3
COO-
Met- Ser-Pro-Thr-...-...-...-
STOP!
UGA
5’
3’
mRNA
Destino proteico:
l’mRNA può essere tradotto
da ribosomi liberi o ribosomi legati al RER
m.p.
vescicole
secretorie
lisosomi
RIBOSOMI LIBERI
nucleo
citoplasma
RIBOSOMI SUL
RETICOLO
ENDOPLASMATICO
mitocondri
cloroplasti
26
Le proteine ed i loro segnali di smistamento
Proteina non ripiegata
Proteina ripiegata
sequenza
segnale
zona
segnale
Ad ogni segnale deve corrispondere un recettore proteico
complementare
REGOLAZIONE
ESPRESSIONE GENICA
eucarioti
27
Controllo o regolazione
della espressione genica negli EUCARIOTI
Scopo:
differenziamento cellulare,
ovvero espressione coordinata nel tempo
di geni diversi in cellule diverse.
Geni costitutivi cioè trascritti e tradotti in tutte le cellule
Geni specifici attivati selettivamente da meccanismi di
regolazione genica
L’espressione genica è modulabile anche da segnali esterni
che possono “accendere” o “spegnere” geni specifici.
Livelli di controllo sulla
espressione genica
DNA
1.pre-trascrizionale o
cromatinico
2. trascrizionale
trascritto primario
di RNA (HnRNA)
3. post-trascrizionale
mRNA
4. traduzionale
proteina
5. post-traduzionale
proteina attiva
o inattiva
controllo sull’attività
della proteina
28
1
DNA
gene scelto
nel nucleo
Livello di controllo pre-trascrizionale o cromatinico (sul DNA)
- già nell’organizzazione della cromatina (eucromatina/eterocromatina)
- in alcuni casi il silenziamento può essere invertito e i geni venire
attivati attraverso processi che rendono la cromatina meno compatta
modificazioni strutturali della cromatina operate da
COMPLESSI PROTEICI di rimodellamento ed enzimi
che modificano gli istoni:
modulazione della espressione genica
DNA
2
pre-mRNA o HnRNA
nel nucleo
Livello di controllo trascrizionale (da DNA a pre m-RNA):
il controllo si esercita sulla attività della RNA polimerasi.
- sul suo riconoscimento ed aggancio al promotore
- sulla sua efficienza nel trascrivere
regolazione su quale gene trascrivere e su quanto pre-RNA produrre
29
Gene: Cosa fa accendere il gene?
PROMOTORE
SEQUENZE
REGOLATRICI
GENE
INTENSIFICATORI
SILENZIATORI
SEQUENZE
TRASCRIVIBILI
PROMOTORE
Sequenza nel DNA di almeno 40 nucleotidi,
natura modulare con affinità più o meno elevata per la RNA
polimerasi, di solito posta a monte del gene.
Hanno:
siti di riconoscimento (es. TATA box)
sito di legame stabile
sito di inizio della trascrizione
siti di regolazione (simili a intensificatori e/o silenziatori)
Risposta: NECESSITA’ DI SEQUENZE SEGNALE
Il PROMOTORE
Sequenza data dalla combinazioni di corti elementi
solitamente collocati nella regione immediatamente a
monte del gene, spesso entro 200 bp dal sito di inizio
della trascrizione.
I PROMOTORI HANNO NATURA MODULARE!
30
SITI DI REGOLAZIONE, anche a grande distanza!
Si tratta di corte SEQUENZE di DNA
Enhancer ovvero intensificatori = aumentano la capacità di iniziare
la trascrizione, intensificano la trascrizione
Silenziatori = spengono o reprimono la trascrizione
Sequenze tessuto-specifiche= in grado di attivare/inattivare trascrizione
in rapporto alla trasduzione di segnali recepiti dall’esterno
La trascrizione ovvero la attività della RNA polimerasi
sarà dovuta ad un bilancio complessivo tra fattori che
la favoriscono o la inibiscono.
SEQUENZE di DNA riconosciute da
specifiche proteine dette FATTORI DI TRASCRIZIONE
“Dialogo”
tra sequenze regolatrici di DNA
e proteine
31
Nella regolazione della trascrizione, ad attivare o reprimere
la RNA polimerasi, intervengono
proteine di 2 tipi:
FATTORI GENERALI
o BASALI
di trascrizione
PROTEINE REGOLATRICI
SPECIFICHE
Inizio della trascrizione
FATTORI GENERALI
di trascrizione
RNA polimerasi II
TFIID
TFIIB
TATA box
gene X
DNA
promotore
Questi fattori sono richiesti per l’inizio della trascrizione
e sono simili per tutti i geni trascritti dalla RNApolimerasi II
32
PROTEINE REGOLATRICI
SPECIFICHE
DNA
RNA polimerasi II
gene X
TFIID TFIIB
TATA box
promotore
trascritto di RNA
Sequenze regolatrici
Le proteine specifiche si possono
Integrazione al PROMOTORE di:
legare a sequenze specifiche, anche
- RNA pol
lontane sia a valle che a monte del gene.
- fattori generali di trascrizione
- serie multiple di proteine regolatrici Il ripiegamento del DNA le porta
poi vicine al promotore per influenzare
la polimerasi.
Come agiscono i fattori di trascrizione o proteine regolatrici?
EFFETTO COMBINATORIO
Un singolo promotore può essere regolato da
molte sequenze regolatrici sparse lungo il DNA e
riconosciute da più proteine regolatrici
proteine regolatrici
+
+
-
+
sinergia trascrizionale
33
EFFETTO DI COORDINAZIONE
Una singola proteina regolatrice/fattore di trascrizione
può regolare e quindi coordinare l’espressione genica
di parecchi geni diversi
proteina regolatrice
Gene networks - un gene può esprimere le proprie
informazioni solo nel contesto dell’intero genoma
della cellula
DNA
Promot 1
gene1
Promot 2
gene2
Promot 3
-
gene3
Promot 4
gene4
+
FATTORI DI
TRASCRIZIONE
RNA
proteine
34
pre-mRNA o HnRNA
3
nel nucleo
mRNA (maturo)
4
Livello di controllo post-trascrizionale (da pre m-RNA a mRNA):
-controllo sulla elaborazione dell’mRNA,
la maturazione comporta 3 tipi fondamentali di modificazioni
chimiche del trascritto primario a ottenere mRNA maturo
- controllo del trasporto dell’mRNA al citoplasma e sulla sua
stabilità nel citoplasma (importante il 3’UTR)
Eventi post-trascrizionali:
dal preRNA all’mRNA
1- aggiunta del CAP (m7G)
aggiunta in 5’ di una 7metil-guanosina:
preserva il trascritto dalla degradazione ed è segnale di
aggancio per il ribosoma
2- poli-adenilazione in 3’OH
aggiunta di una coda di poliA (200-250):
aiuta il passaggo al citoplasma, influenza la stabilità dell’mRNA
3- splicing
processo di taglia e cuci per eliminare gli introni
35
Da pre-mRNA a mRNA maturo!!!
PROMOTORE
DNA
E1
TATA
-25
I1
E3
I2
+1
Esone 1
Trascritto
Primario o
pre-mRNA
mRNA
E2
Esone 3
Esone 2
AUG
CAP
UGA
AUG
aaaaaaaaa
UGA
5’
3’
SPLICING= TAGLIA E CUCI degli introni/esoni
36
Spliceosoma = complesso ribonucleoproteico
150 proteine, 5 RNA snRNA (50-200 nucleotidi): U1, U2, U4, U5, U6
Dimensioni simili ad un ribosoma
Proteine
snRNA
small nuclear RNA
snRNPs
SPLICING ALTERNATIVO: lo splicing mostra una
notevole flessibilità
Aggiunta di un esone
Aggiunta di un tratto
intronico
1
2
1
2
Esclusione a vicenda
di esoni
1
2
Sito di splicing interno
1
2
37
Splicing alternativo
può produrre forme diverse di
mRNA e dunque di una proteina
dallo stesso gene
Calcitonina= ormone che riduce la
concentrazione del Ca2+nel plasma
prodotto dalla tiroide
CGRP= peptide correlato alla calicitonina,
potente vasodilatatore che può intervenire
nella trasmissione del dolore su sistema
nervoso periferico e centrale
Variazioni
al modello standard di gene:
un gene
un mRNA
una proteina
un LOCUS
più prodotti genici alternativi
Un singolo gene è un genoma in
miniatura
38
DOPO MATURAZIONE
mRNA MATURO
SEQUENZA CODIFICANTE
5’
CAP
AUG
CDS
5’UTR
STOP
UAA
UGA
UAG
3’
AAAAA
3’UTR
TRADUZIONE
C
N
PROTEINA
L’mRNA al termine della maturazione presenta:
- Regione non tradotta in 5´, 5´ UTR o leader
- Regione codificante, CDS (coding sequence)
- Regione non tradotta in 3´, 3´ UTR o trailer
Funzione delle regioni dell'mRNA
5´UTR
Efficienza della traduzione
CDS
Traduzione (1%)
3´UTR
Stabilità (velocità di degradazione;
concetto di emivita da pochi minuti
a 20-30 ore)
39
mRNA (maturo)
nel citoplasma
5
proteina
6
proteina attiva
5. Livello di controllo traduzionale (da m-RNA a proteina):
Operato da fattori proteici che possono influire sull’inizio della
traduzione e quindi modularla, legandosi alla regione 5´UTR e
magari “mascherandola” al ribosoma
6. Livello di controllo post-traduzionale (sulla proteina):
- controllo sulla modificazione delle proteine sintetizzate
-controllo del ripiegamento (aiutato da proteine chaperon)
e trasporto nella cellula al sito di funzione
Livello di controllo post-traduzionale (sulla proteina):
modificazione delle proteine sintetizzate che risultano essenziali
perché la proteina possa funzionare
Fosforilazione
in TIROSINA
TREONINA
SERINA
40
A modulare la espressione genica delle proteine cellulari
intervengono anche i geni non codificanti proteine o
ncRNA (non coding RNA)
Il nuovo “dogma”
microRNA
ncRNA
Ipotesi
ncRNA non sono semplice “rumore trascrizionale”, ma
più probabilmente svolgono un ruolo regolatorio e sono
coinvolti in numerosi processi cellulari e biologici, in
particolare durante lo sviluppo e il differenziamento
41
ncRNA - generalità
Sempre di più nei database internazionali
Gruppo eteogeneo per dimensioni e funzione:
difficile classificazione
Caratterizzati spesso da una alta densità di codoni
di STOP e quindi dalla assenza di ORF (cornici di
lettura aperte) estese
La loro trascrizione ed elaborazione è sottoposta a
controllo
Possono formarsi dalle zone introniche di geni coding
o da vere e proprie unità geniche
Grande importanza per il ruolo da protagonisti
nella REGOLAZIONE GENICA
ncRNA - meccanismi
Dinamica della struttura della cromatina (regolatori epigenetici)
Modulazione della trascrizione (interazioni a triplice elica)
Silenziamento genico post-trascrizionale: stabilità mRNA
Attraverso meccanismi di “Interferenza da RNA”
Regolazione espressione genica
Cambiamenti nei livelli di espressione degli ncRNA sono associati
con forme diverse di cancro e patologie neurodegenerative
42
~20-300 nt
2- small RNA
snoRNA
3-Medium e large
RNAs
~300->10.000 nt
1- microRNA
siRNA
ncRNAs
~18-25 nt
alcuni
non noti
F.F. Costa
“Non-coding RNAs: lost in
translation?”
Gene 386 (2007) 1-10
Studi funzionali
Nuovi ncRNAs
funzionali
~20- 300 nt
- modificazioni in RNA
- regolazione trascrizionale
- ruolo strutturale
2- small RNA
snoRNA,
3-Medium e large
RNAs
~300 - >10000 nt
1- microRNA
siRNA
~18-25 nt
- silenziamento
genico posttrascrizionale
(interferenza da
RNA)
ncRNAs
- inattivazione X
- regolazione
trascrizionale
alcuni
non noti
43
miRNA micro-RNA
Regolazione post-trascrizionale sequenza-specifica
dell’espressione genica; associata a differenziamento e
sviluppo embrionale
Come si formano?
Da precursori più lunghi (70nt)
ripiegati a forcina
Questa sorta di trascritti primari
sono tagliati da un complesso
enzimatico “Dicer”
I miRNA (20-22nt) sono così
trasportati al proprio bersaglio
molecolare dal complesso
proteico RISC
miRNA+
RISC
miRNA micro-RNA: meccanismi di azione
Regolazione post-trascrizionale:
1. Degradazione mRNA
1
2. Blocco della traduzione
dsRNA
Quasi perfetta
complementarietà
al bersaglio (20-22 basi)
2
Brevi tratti di
complementarietà
al bersaglio (6-8 basi)
44
miRNA
Curiosità e spunti:
• la maggioranza di quelli specificamente espressi in
cellule staminali embrionali umane sono down-regolati
durante il proseguo dello sviluppo embrionale
• molti specificamente implicati nello sviluppo del
cervello come dimostrato in diversi lavori sul modello
murino
• altri legati a sviluppo di cancro: agirebbero come
oncogeni od onco-soppressori (es. miR-21, let-7, miR122a)
Nature, feb. 1998
“Potent and specific genetic interference by doublestranded RNA (dsRNA)
in Caenorhabditis elegans”
Andrew Fire, 47 anni
Stanford California
Craig Mello, 46 anni
Massachusetts Medical School
Premio Nobel per la medicina 2006
45
Cosa si intende per RNA interference
(RNAi)
Sistema di silenziamento genico basato su RNA
RNAi è un processo mediante il quale RNA a doppio filamento (double
strand RNA o dsRNA) silenzia, in modo sequenza-specifico,
l’espressione di geni omologhi per sequenza attraverso l’appaiamento
con l’mRNA bersaglio, seguito dalla sua degradazione; quindi:
silenziamento post-trascrizionale.
E’ un meccanismo conservato a livello evolutivo
di degradazione specifica di mRNA bersaglio che
avviene in 2 stadi:
1°stadio-iniziale. I dsRNAs sono
processati da un enzima dimerico,
DICER (RNasiIII). Si generano i
siRNAs.
2°stadio-effettore. siRNAs o small
interfering RNA (< 22nt), vengono
incorporati in un secondo complesso
enzimatico (endonucleasico), RISC
(complesso di silenziamento indotto da
RNA) che degrada uno dei due
filamenti e usa l’altro per riconoscere,
in base all’appaiamento delle basi,
il substrato da degradare attraverso
l’azione in base alla complementarietà
delle basi.
dsRNA
siRNA
46
Modello degli stadi dell’RNAi: i siRNAs
DICER
siRNA
RNAsi ATP-dip.
+ RISC (elicasi ATP-dip.)
Riconoscimento dell’mRNA bersaglio
DNA
+/AAAA
HnRNA
_
ncRNA
AAAA mRNA
proteina X
Fattore di
trascrizione
proteina
nucleare
47
INTERATTOMA COMPLESSO
fondamentale nella regolazione
della espressione genica
Proteoma
Trascrittoma
Tutte le molecole di RNA
trascritte a partire da un
genoma ANCHE GLI ncRNAs
Tutte le proteine codificate
dal genoma ANCHE QUELLE
A LOCALIZZAZIONE NUCLEARE
Immagine mentale della attivazione dei geni lungo il DNAi geni lungo il DNA sono come “luci di Natale” lungo la
matassa dei fili: si accendono e si spengono ad intermittenza e
con una intensità che va da MASSIMA luminosità a MINIMA
luminosità e può essere modulata cioè regolata sia nella qualità
che nelle quantità.
48