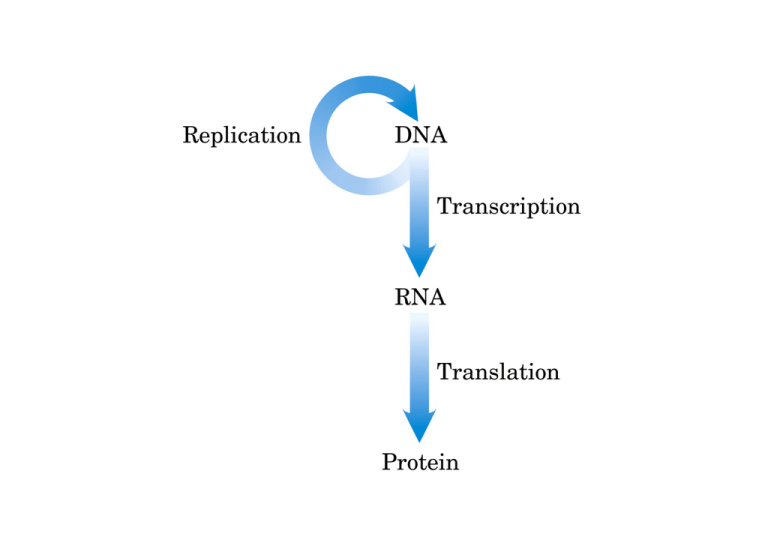
La replicazione del DNA
Mappa del cromosoma di E. coli in cui sono indicate le posizioni dei geni che
codificano proteine importanti per il metabolismo del DNA
L’esperimento di Meselson e Stahl (1957):
la replicazione del DNA è semiconservativa.
E‘ possibile visualizzare il DNA marcato con 14N e 15N separato con
centrifugazione in un gradiente di densità di cloruro di cesio.
Fotografia dopo irradiazione con luce ultravioletta.
……e quantizzarlo (analisi densitometrica)
John Cairns, mediante marcatura con trizio (3H) dimostrò che entrambe le catene
vengono replicate contemporaneamente (la catena nuova è in rosso).
La replicazione dei cromosomi batterici è bidirezionale
Replicazione bidirezionale
Forcelle
di replicazione
Origine
Replicazione unidirezionale
Origine
Entrambe le estremità delle
anse hanno forcelle di
replicazione
E’ possibile visualizzare il DNA parzialmente denaturato
Le anse di replicazione iniziano sempre in un punto preciso, chiamato origine.
La sintesi del DNA avviene in direzione 5’Â3’ ed è discontinua.
Catena
veloce
Catena
lenta
Le catene di DNA a livello della forcella di replicazione
Enzimi che degradano il DNA
Gli enzimi che degradano specificamente il DNA sono chiamati nucleasi o
Dnasi.
Le esonucleasi degradano il DNA a partire da un’estremità della molecola.
Alcuni enzimi possono operare solo in direzione 5’Æ3’, altri in direzione
3’ Æ 5’, rimuovendo rispettivamente i nucleotidi a partire dall’estremità 5’ o
dall’estremità 3’ di una catena di acido nucleico a doppia elica.
Le endonucleasi agiscono sulla porzione interna degli acidi nucleici,
degradandoli in frammenti sempre più piccoli.
Alcune importanti endonucleasi (es.: endonucleasi di restrizione) effettuano
tagli solo in corrispondenza di specifiche sequenze nucleotidiche.
Il DNA viene sintetizzato dalle DNA polimerasi
Arthur Kornberg nel 1955 purificò e caratterizzò la prima DNA
polimerasi di E. coli.
Questo enzima a singola catena polipeptidica è ora chiamato DNA
polimerasi I.
In seguito si è scoperto che E. coli contiene almeno altre quattro DNA
polimerasi
La DNA polimerasi catalizza la formazione dei ponti fosfodiestere
La reazione di allungamento della catena, catalizzata dalla DNA polimerasi, procede
attraverso un attacco nucleofilico del gruppo ossidrilico terminale 3’-OH del primer
sull’atomo di fosforo più interno (gruppo fosforico α) di un deossiribonucleoside trifosfato.
Nuovo deossinucleoside
5’-trifosfato
da aggiungere
Catena nascente
di DNA (primer)
Catena stampo
di DNA
Deossiribosio
(dNMP)n + dNTP Æ (dNMP)n+1+ PPi
DNA
DNA allungato
di un nucleotide
Per vedere questa immagine
occorre QuickTime™ e un
decompressore Animation.
Il DNA viene sintetizzato dalle DNA polimerasi
Tutte le DNA polimerasi necessitano di uno stampo.
Tutte le DNA polimerasi necessitano di una molecola di innesco o primer.
Il numero medio di nucleotidi aggiunti prima che la polimerasi si dissoci
determina la sua processività. Le diverse DNA polimerasi sono caratterizzate da
processività molto variabili.
Il frammento di Klenow della DNA polimerasi I
Sono
note
tridimensionali
polimerasi.
le
di
strutture
molte DNA
La prima struttura determinata è
stata quella del cosidetto frammento
di Klenow della DNA polimerasi I
di E. coli.
Questo frammento comprende le due
parti principali dell’enzima, l’unità
con attività polimerasica e l’unità con
attività esonucleasica 3’Æ5’ .
Tutte le DNA polimerasi hanno proprietà strutturali simili
Le DNA polimerasi sono notevolmente simili
nella loro forma generale.
I domini “dita” e “pollice” si avvolgono
intorno al DNA e lo tengono in posizione in
modo che possa attraversare il sito attivo
dell’enzima, costituito principalmente da
residui del dominio “palmo”.
Tutte le polimerasi catalizzano lo stesso tipo di
reazione, che dipende dalla presenza di ioni
metallici.
Oltre al dominio con attività polimerasica,
il frammento di Klenow comprende un
dominio con attività esonucleasica in
direzione 3’Æ5’
Alla reazione di polimerizzazione partecipano due ioni metallici
Come tutti gli enzimi i cui substrati sono nucleosidi
trifosfato, le DNA polimerasi richiedono ioni metallici
(generalmente Mg2+) per il loro funzionamento.
Uno ione metallico lega i deossinucleosidi trifosfato
(dNTP) e il gruppo ossidrilico in posizione 3’ del
primer.
Entrambi gli ioni metallici interagiscono con il gruppo
carbossilico di due residui di aspartato del dominio
“palmo” della polimerasi, che contribuiscono a
mantenere gli ioni nella giusta posizione.
Lo ione metallico legato al primer attiva il gruppo
ossidrilico 3’, facilitando l’attacco nucleofilo al gruppo
fosforico α del dNTP.
I due ioni metallici stabilizzano la carica negativa che
si accumula nello stato di transizione pentacoordinato.
Lo ione metallico inizialmente legato al dNTP
stabilizza la carica negativa sul pirofosfato che viene
prodotto.
La replicazione del DNA deve procedere con un grado elevato di precisione.
In E. coli viene commesso un errore ogni 109-1010 nucleotidi aggiunti.
Nel caso del cromosoma di E. coli, lungo circa 4,6 x 106 coppie di basi, questo
significa un errore ogni 1000 - 10.000 replicazioni.
Durante la polimerizzazione, la distinzione tra nucleotidi corretti e sbagliati
consiste non solo nei legami idrogeno che conducono ad un corretto
appaiamento tra basi complementari, ma anche nel riconoscimento della
geometria delle coppie di basi A=T e G ≡C.
Contributo della geometria delle coppie di basi alla replicazione fedele del DNA
Le coppie standard A=T e G≡C hanno una geometria molto simile
La geometria di coppie di basi sbagliate
le esclude dal sito attivo perché la DNA
polimerasi I ha un sito attivo in cui tali
coppie non possono entrare.
Selettività di forma
Il legame di un nucleoside trifosfato (NTP) alla DNA polimerasi, induce un
cambio conformazionale che genera una tasca dove possono alloggiare il
nucleotide e il suo complementare sul filamento stampo.
Questo cambiamento conformazionale può avvenire solo se il nucleotide da
aggiungere corrisponde a quello complementare sul filamento stampo secondo
l’appaiamento di Watson e Crick.
Le DNA polimerasi formano due legami idrogeno con le coppie di basi presenti
nel solco minore
Tra i meccanismi che contribuiscono ad aumentare la fedeltà della replicazione del DNA
da parte delle DNA polimerasi vi sono i legami idrogeno formati da alcuni residui
dell’enzima con il lato della scanalatura minore che si trova in contatto con il sito attivo.
Nella scanalatura minore gli accettori di legami idrogeno si trovano sempre nelle stesse
posizioni in tutti gli appaiamenti di basi del tipo Watson e Crick.
Questi legami idrogeno che si stabiliscono tra l’enzima ed il substrato agiscono come
“righello” graduato che misura se nel sito attivo si viene a trovare un appaiamento di basi
con una corretta disposizione spaziale.
Accurati studi in vitro hanno dimostrato che le DNA polimerasi inseriscono
un nucleotide sbagliato ogni 104-105.
Spesso tali errori accadono perché i nucleotidi si presentano per breve tempo
in una forma tautomerica insolita che permette la formazione di legami
idrogeno con una base normalmente non complementare.
L’attività esonucleasica 3’Æ5’ (proofreading o di correzione delle bozze) delle
DNA polimerasi serve a ridurre questa frequenza di errori.
Attività esonucleasica 3’Æ5’ (proofreading) della DNA polimerasi I
Attività esonucleasica 3’Æ5’ (proofreading) della DNA polimerasi I
Correzione di bozze
La catena polinucleotidica in formazione abbandona occasionalmente il sito
polimerasico della DNA polimerasi I e si sposta verso il sito esonucleasico.
Il meccanismo di correzione di bozze si basa sul fatto che l’estremità di un filamento in
corso di sintesi contenente un nucleotide erroneamente appaiato ha una maggiore
probabilità di abbandonare il sito attivo della polimerasi e di avvicinarsi al sito
esonucleasico.
Combinando l’attività proofreading e la selezione delle basi, una DNA
polimerasi commette circa 1 errore ogni 106-108 coppie di basi.
La maggiore precisione riscontrata nella replicazione del DNA di E.coli è
dovuta all’azione degli enzimi del riparo.
Nel 1969 John Cairns isolò un ceppo batterico in cui il gene della DNA
polimerasi I era mutato e l’enzima prodotto era inattivo.
Il ceppo, sebbene più sensibile all’azione di agenti che danneggiano il DNA,
era vitale!
Escherichia coli possiede almeno cinque DNA polimerasi.
La DNA polimerasi II ha una funzione altamente specializzata nel riparo del
DNA.
La DNA polimerasi III è il più importante enzima della replicazione di E. coli.
Le DNA polimerasi IV e V, isolate nel 1999, sono coinvolte in un particolare
processo di riparazione, la riparazione soggetta ad errori o risposta SOS.
DNA polimerasi I
La DNA polimerasi I ha una serie di funzioni di “pulizia” durante la
replicazione, la ricombinazione e il riparo.
La DNA polimerasi I è l’unica DNA polimerasi di E. coli provvista di attività
esonucleasica 5’ Æ3’.
Quando la regione contenente l’attività esonucleasica 5’Æ3’ della DNA
polimerasi I viene rimossa mediante un blando trattamento con proteasi, il
frammento rimanente (Mr = 68.000) mantiene l’attività polimerasica e di
proofreading e viene chiamato frammento di Klenow.
Il frammento di Klenow della DNA polimerasi I
“Nick translation”, una attività di cui è dotata solo la DNA polimerasi I
L’attività 5’ Æ3’ esonucleasica della
DNA polimerasi I è in grado di
degradare una catena di RNA o di
DNA legata allo stampo e di sostituire
simultaneamente la catena degradata
con l’attività polimerasica.
DNA polimerasi III
La DNA polimerasi III è molto più complessa della DNA polimerasi I. La sua
struttura multimerica comprende 10 diverse subunità ed ha una massa di
circa 900 kd.
Le attività di polimerizzazione e di proofreading risiedono in due subunità
diverse, le subunità α e ε rispettivamente.
La subunità θ associata con α e ε forma il nucleo della polimerasi, in grado di
sintetizzare il DNA anche se con bassa processività (numero di nucleotidi
aggiunti prima che l’enzima si dissoci).
Due nuclei αεθ si organizzano a costituire un dimero asimmetrico.
Struttura proposta per l’oloenzima della DNA polimerasi III
L’oloenzima è organizzato come un dimero per permettere di replicare
entrambi i filamenti del DNA parentale.
L’oloenzima è asimmetrico perché la catena veloce e quella lenta sono
sintetizzati con modalità differenti.
Una subunità τ2 è associata con una parte dell’enzima; una subunità γ2 e
le subunità ( δδ’χΨ)2 sono dall’altra parte.
Un complesso αεθ è associato con ciascuna delle due regioni.
La processività è conferita dalle
subunità β2 e τ2.
Le due subunità β della DNA
polimerasi III di E. coli formano
una struttura a forma di ciambella
che avvolge il DNA e agisce come
una pinza.
Ciascuno dei dimeri di
si
associa
con
cataliticamente attivo
lungo il DNA con il
della replicazione.
subunità β
un
core
e scivola
procedere
La pinza formata dalle subunità β
aumenta la processività della
polimerasi fino a 500.000 volte
perché evita la dissociazione
dell’enzima.
La replicazione del DNA richiede numerosi enzimi e fattori proteici.
L’intero complesso è detto replisoma.
Le elicasi si muovono lungo il DNA e separano le catene usando
l’energia dell’ATP.
Le topoisomerasi risolovono la tensione topologica nella struttura ad
elica del DNA che si genera con la separazione delle catene.
Le proteine che legano il DNA a singolo filamento stabilizzano le catene
separate.
Le primasi sintetizzano i primer (generalmente brevi frammenti di
RNA).
La DNA polimerasi I rimuove i primer e li sostituisce con DNA.
Le DNA ligasi riparano le interruzioni dei legami fosfodiesterici che
rimangono dopo l’azione della DNA polimerasi I.
La replicazione del DNA
Inizio
Allungamento
Termine
oriC, origine della replicazione di E.coli (245 bp)
Modello dell’inizio della replicazione all’origine di E. coli, oriC
DnaA si lega alle 4 sequenze ripetute
di 9 coppie di basi
Attraverso una reazione che richiede
ATP ed è facilitata dalla proteina HU,
la proteina DnaA riconosce e denatura
le 3 sequenze ripetute di 13 bp, ricche
in A=T.
DNA girasi
(TopoII)
SSB
La proteina DnaB si lega a questa
regione denaturata con una reazione
che richiede DnaC.
Facilitano
il disavvolgimento
da parte di DnaB
Due esameri di DnaB, che funzionano
come elicasi, disavvolgono il DNA in
entrambe le direzioni, creando due
potenziali forcelle di replicazione
Modello dell’inizio della replicazione all’origine di E. coli, oriC
L’origine
della
replicazione
è
influenzata dalla metilazione del DNA
da parte della metilasi Dam
(5’-GAmTC-3’)
La
regione
oriC
dell’elica
neosintetizzata è emimetilata ed
interagisce
con
la
membrana
plasmatica.
DNA girasi
(TopoII)
SSB
Facilitano
il disavvolgimento
da parte di DnaB
La regione oriC deve essere
nuovamente metilata dalla Dam
metilasi prima che possa legare
nuovamente DnaA e iniziare la
replicazione.
Allungamento (catena veloce)
La sintesi della catena veloce incomincia con la sintesi da parte di una primasi
(DnaG) di un breve primer di RNA (da 10 a 60 nucleotidi) all’origine della
replicazione.
I deossiribonucleotidi vengono poi aggiunti al primer da parte della DNA
polimerasi III.
Una volta iniziata, la sintesi della catena veloce procede in modo continuo,
avanzando di pari passo con la forcella di replicazione.
Allungamento (catena lenta)
La sintesi della catena lenta viene compiuta tramite corti frammenti di
Okazaki.
Inizialmente il primer è sintetizzato dalla primasi.
Come nella sintesi della catena veloce, la DNA polimerasi III si lega al primer e
aggiunge deossiribonucleotidi.
La complessità della replicazione sta nel coordinamento della sintesi delle due
catene: entrambe le catene sono prodotte contemporaneamente da un singolo
dimero asimmetrico della DNA polimerasi III.
Il DNA della catena lenta si piega ad anello, di modo che i due percorsi di
polimerizzazione possano procedere insieme.
Sintesi dei frammenti di Okazaki
(DnaG)
Sia la catena veloce che la catena lenta sono prodotte da un singolo dimero
asimmetrico di DNA polimerasi III
(γ2δδ’χψ)
Sia la catena veloce che la catena lenta sono prodotte da un singolo dimero
asimmetrico di DNA polimerasi III
Per vedere questa immagine
occorre QuickTime™ e un
decompressore Animation.
Quando il frammento di Okazaki neosintetizzato è completo, il primer viene rimosso dalla
DNA polimerasi I e rimpiazzato da DNA.
L’interruzione che rimane viene ricucita dalla DNA ligasi.
Catena
lenta
La DNA ligasi catalizza la formazione di un legame fosfodiesterico tra l’ossidrile
all’estremità 3’ di una catena di DNA e il fosfato all’estremità 5’di un’altra catena.
Il fosfato deve essere attivato tramite adenilazione.
lisina
Meccanismo di azione della DNA ligasi
Le due forcelle di replicazione del cromosoma circolare di E. coli si incontrano
in una regione terminale contenente copie multiple di una sequenza di 20 coppie
di basi detta Ter (“termine”)
Origine
Forcella in senso
antiorario
Trappola per
la forcella in
senso
orario
Forcella in
senso
orario
Trappola per
la forcella in
senso
antiorario
La proteina Tus (terminus utilization
substance) lega la sequenza Ter
Quando una delle due forcelle di replicazione incontra un complesso funzionale Tus-Ter,
si ferma; l’altra forcella si ferma quando incontra la prima forcella già ferma.
Termine della replicazione del cromosoma di E. coli.
A replicazione avvenuta, i due cromosomi
figli rimangono intersecati come catenanes,
e non possono separarsi perché entrambi
sono chiusi covalentemente.
Forcella in senso
antiorario
Forcella in senso
orario
Fine della replicazione
Cromosomi
Intersecati (catenanes)
DNA topoisomerasi IV
Cromosomi
separati
In E. coli, la separazione dei catenanes
richiede l’azione dell’enzima topoisomerasi
IV, che separa i due cromosomi intersecati
rompendo temporaneamente entrambe le
catene di uno dei cromosomi e permettendo
all’altro
di
passare
attraverso
l’interruzione.
La replicazione del DNA negli eucarioti.
Nel lievito le origini della replicazione sono sequenze di circa 150 coppie di
basi dette replicatori (o ARS, autonomously replicating sequences)
L’inizio della replicazione in tutti gli eucarioti richiede una proteina
multimerica, ORC (origin recognition complex), che si lega a numerose
sequenze contenute nel replicatore.
ORC interagisce ed è regolata da varie altre proteine coinvolte nel controllo
del ciclo cellulare eucariotico.
Come i batteri anche le cellule eucariotiche possiedono vari tipi di DNA
polimerasi.
La replicazione dei cromosomi nucleari è effettuata dalla DNA polimerasi α,
dalla DNA polimerasi δ e dalla DNA polimerasi ε.
La replicazioneDNA polimerasi α
La DNA polimerasi α è un enzima multimerico.
Una delle subunità ha attività primasica; la subunità maggiore contiene
l’attività polimerasica.
La DNA polimerasi α non è dotata di attività 3’Æ5’ esonucleasica di
proofreading e probabilmente funziona per la sintesi dei primer per i
frammenti di Okazaki.
DNA polimerasi δ
La DNA polimerasi δ è costituita da due subunità.
La DNA polimerasi δ è dotata di attività 3’Æ5’ esonucleasica di proofreading
e probabilmente è responsabile della sintesi della catena veloce e della catena
lenta agendo come un complesso analogo alla DNA polimerasi III batterica.
La DNA polimerasi δ è associata a una proteina chiamata antigene nucleare
della cellula proliferante (PCNA), che sembra avere una funzione analoga a
quella della subunità β della DNA polimerasi III di E. coli formando una
sorta di pinza circolare che aumenta la processività dell’enzima.
La DNA polimerasi ε è probabilmente coinvolta nella rimozione dei primer dei
frammenti di Okazaki (come DNA pol I di E. coli) e nel riparo del DNA.
La proteina di replicazione A (RPA) è una proteina che lega il DNA a singola
elica e svolge una funzione analoga a quella della proteina SSB di E. coli.
Il fattore di replicazione C (RFC) interagisce con PCNA e facilita
l’assemblaggio dei complessi replicativi attivi.
I telomeri sono strutture caratteristiche alle estremità dei cromosomi lineari
Mentre i genomi di quasi tutti i procarioti sono circolari, i cromosomi degli esseri
umani e di altri eucarioti sono lineari.
E’ difficile replicare interamente le estremità di un DNA lineare, perché le polimerasi
agiscono solo in direzione 5’Æ3’.
Dopo la rimozione del primer di RNA la catena ritardata avrà quindi una estremità 5’
incompleta.
Ogni ciclo di replicazione accorcerà ulteriormente il cromosoma.
Soluzione del problema:
Le sequenze che si trovano all’estremità dei cromosomi, dette telomeri, hanno
caratteristiche di sequenza e strutturali particolari.
Il DNA telomerico contiene centinaia di ripetizioni in fila di una sequenza di sei
nucleotidi ricca in G su uno dei due filamenti (nell’uomo AGGGTT). Il filamento ricco
in G è leggermente più lungo dell’altro.
Speciali enzimi, le telomerasi, sono necessari per la sintesi dei telomeri.
Formazione dei telomeri
I telomeri sono replicati dalla telomerasi.
La telomerasi è una trascrittasi inversa specializzata che
contiene il proprio stampo di RNA.
Per vedere questa immagine
occorre QuickTime™ e un
decompressore Animation.

![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)






![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)



