caricato da
sabadiego96
Statistica multivariata: analisi delle componenti principali
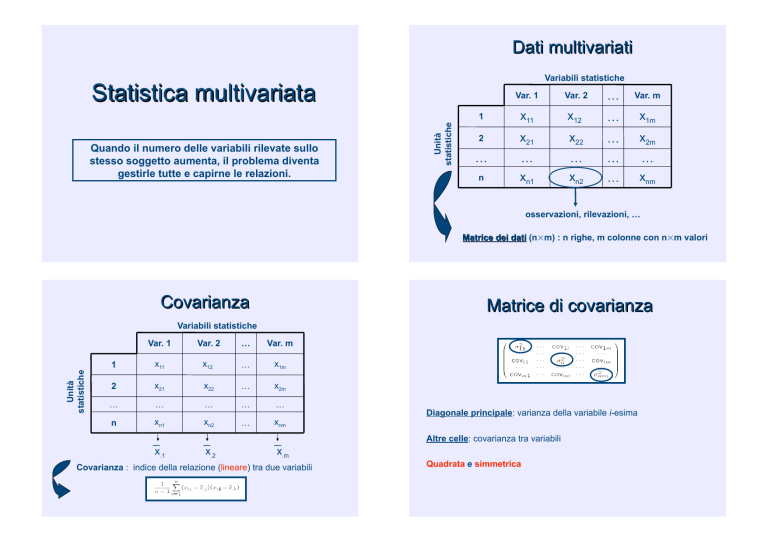
Dati multivariati Variabili statistiche Quando il numero delle variabili rilevate sullo stesso soggetto aumenta, il problema diventa gestirle tutte e capirne le relazioni. Unità statistiche Statistica multivariata Var. 1 Var. 2 … Var. m 1 x11 x12 … x1m 2 x21 x22 … x2m … … xn1 … xn2 … … xnm n … osservazioni, rilevazioni, … Matrice dei dati (n!m) : n righe, m colonne con n!m valori Covarianza Matrice di covarianza Unità statistiche Variabili statistiche Var. 1 Var. 2 … Var. m 1 x11 x12 … x1m 2 x21 x22 … x2m … … … … … n xn1 xn2 … xnm x.1 x.2 Diagonale principale: varianza della variabile i-esima Altre celle: covarianza tra variabili x.m Covarianza : indice della relazione (lineare) tra due variabili k Quadrata e simmetrica Analisi multivariata Statistica multivariata Cercare di capire le relazioni che intercorrono tra le variabili ! Analisi fattoriale Introdurre tante variabili in un’analisi non ha molto senso, né al livello biologico, né al livello statistico. ! ! ! ! analisi (prevalentemente) simmetrica rappresentare un numero elevato di variabili per mezzo di un numero inferiore di variabili ipotetiche (o latenti), i cosiddetti fattori Il modello diventa troppo complesso. Diventa difficile interpretare i risultati. Le stime dei parametri diventano molto instabili. Più parametri inseriamo, più osservazioni ci vogliono per stimarli. ! Regressione multipla analisi asimmetrica formulare opportuni modelli descrittivi/interpretativi Eliminiamo le variabili che sono molto correlate tra di loro. Se due variabili sono molto correlate, allora l’informazione di una è contenuta quasi completamente nell’altra. Metodi multivariati - 1 Analisi fattoriale Definizioni Riduzione di dimensione ! componenti principali (PCA) ! multidimensional scaling (MDS) ! Insieme di diverse tecniche per l’analisi della struttura dei dati, legate da un obiettivo comune, quello di rappresentare un numero elevato di variabili per mezzo di un numero inferiore di variabili ipotetiche (o latenti), i cosiddetti fattori. ! Processo di “riduzione della complessità” della realtà con il duplice obiettivo di una semplificazione dei modelli interpretativi e di un chiarimento concettuale, arrivando anche al risultato di una riduzione dei dati. Analisi fattoriale Analisi di raggruppamento tecnica descrittiva Analisi discriminante - analisi di segmentazione/cluster - analisi interna tecnica predittiva (unsupervised learning) - classificazione (machine/supervised learning) Componenti principali Riduzione della dimensione m dimensioni Perché ? " " Informazione ridondante Algoritmi lenti Come ? " " " " Analisi delle componenti principali Analisi delle corrispondenze Multidimensional scaling ... 2. Non potendo le distanze essere uguali, cerco di minimizzare le distorsioni. Var. m 1 x11 x12 … x1m 2 x21 x22 … x2m … … xn1 … xn2 … … xnm … Per visualizzare i dati ho bisogno di un metodo che riduca la dimensione del mio spazio ad al più 3 dimensioni. Componenti principali Si costruiscono m nuovi assi cartesiani: # ortogonali; # combinazione lineare delle variabili originarie; # catturano man mano porzioni decrescenti di variabilità. PC2 Y " La proiezione riduce le distanze; quindi seguiremo il criterio di rendere max la media dei quadrati delle distanze tra le proiezioni. … n punti 3. Geometricamente si ottiene proiettando i punti su sottospazi dello spazio originario. " Scegliamo il piano di proiezione in cui le distanze saranno conservate al meglio. Var. 2 n Componenti principali 1. Voglio costruire un grafico in modo che le distanze originali tra i punti siano mantenute al meglio. Var. 1 PC1 … combinazione lineare delle variabili originarie X Analisi delle componenti principali # PC loadings : Misura l’importanza di ogni variabile nel catturare la variabilità di una PC. Si può dare una interpretazione alle prime PCs in base alle variabili originali che hanno loadings maggiori. # Scores : nuove coordinate cartesiane dei punti nel nuovo sistema di assi. Analisi delle componenti principali Analisi componenti principali Calcolando, quindi, la matrice di covarianze delle nuove variabili ottengo: Per costruzione queste nuove variabili sono: # ortogonali o indipendenti; (Se ricalcolo la matrice di covarianza sulle Zi, i valori al di fuori della diagonale principale saranno tutti nulli.) # “catturano” quantità decrescente di varianza. Var(Z1) = !1 La varianza totale dei dati è data dalla somma delle varie componenti di varianza: Var(Z2) = !2 … Var(Zn) = !n con !1" !2 " … " !n traccia della matrice di covarianza delle Zi Analisi delle componenti principali Scree test La porzione spiegata da ogni singola componente è data da: Quante componenti principali usare senza perdere troppa informazione? Esistono degli indici per definire il numero ottimo di PC da prendere. Generalmente si fa in modo di catturare almeno l’80% della variabilità totale. Correlazione o covarianza ? Quando la curva diventa piatta (visivamente), quello è il numero di PC più adatto da prendere. Correlazione o covarianza ? Le variabili statistiche possono avere delle unità di misura e dei range di variazione molto diversi. Omogeneizzare i dati: cioè devo standardizzare. standardizzare Fare l’analisi con la matrice di covarianza o con la matrice di correlazione cambia i risultati. Facendo l’analisi PC su queste nuove variabili standardizzate otteniamo che la matrice di covarianza in realtà è una matrice di correlazione. Standardizzare solo quando è necessario ! Esempio Difetti delle PC Classe ED #Sensibilità ai valori anomali (outliers): Poche osservazioni con valori particolarmente “strani” possono far variare sostanzialmente la direzione delle componenti principali. #Si deve usare la totalità dei dati: Non è possibile in una successiva raccolta dei dati ridurre il numero di variabili da raccogliere limitandosi solo ai valori delle variabili latenti che non sono in generale misurabili direttamente. PC è un buon metodo a posteriori. # oggettivo # L’interpretazione dei fattori può essere complessa. # Cosa vuol dire che una PC ha varianza nulla? 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 KRH 15,8 11,29 13,28 13,45 13,15 11,51 13,96 14,16 13,82 11,66 13,9 11,5 13,35 14,05 11,88 11,78 10,31 11,87 13,31 12,05 12,17 10,3 10,29 10,26 11,31 12,32 11,05 11,13 11,1 12,32 13,67 13,06 12,37 10,69 V ar ia nza tota le s pie ga ta 3 4 5 To tale % di vari anza 5 2.42 6 2 8.55 0 1 2.64 3 6. 38 1 2 .775 E- 05 2 .621 1 .427 .632 .319 1 .388 E- 06 STQN 12,54 14,03 14,58 14,18 14,72 14,76 14,69 15,3 14,66 18,42 15,81 18,49 16,54 16,32 19,11 18,72 19,37 17,87 19,36 20,77 19,22 16,18 16,14 16,32 17,02 19,94 21,18 20,93 21,15 20,67 19,19 19,49 21,19 19,96 AGP LVIFMWYC 21,86 22,55 18 18,19 17,85 23,99 16,67 15,13 18,95 15,77 16,75 15,85 16,92 19,61 17,2 18,17 22,68 21,35 18,11 17,4 23,92 28,06 27,88 27,46 24,72 15,1 17,77 18,25 17,65 14,72 14,83 14,36 17,52 20,97 P es i d e i fatto ri non ruo tati % cu mul at a 52 .4 26 80 .9 75 93 .6 19 1 00.00 0 1 00.00 0 T otale % di var ianza 52 .4 26 28 .5 50 2 .621 1 .427 % cumul ata 5 2.42 6 8 0.97 5 • ED : acidi • KRH : basici • STQN : non carichi • AGP : non idrofobici • LVIFMWYC : idrofobici a Componente ED K RH S TQN A GP LVI FMWYC M etod o di e str az io ne: An alisi co mpo ne nti pr incipali. Classificazione dei proteomi batterici attraverso la composizione aminoacidica 35,5 38,39 39,19 39,13 39,22 36,38 38,77 39,73 38,4 40,69 38,8 40,42 38,53 36,79 38,73 38,55 35,69 35,45 36,51 36,73 32,69 33,19 33,34 33,17 34 38,52 36,98 36,79 36,96 38,21 38,38 38,47 36,47 35,97 Mat rice di comp onenti A uto valor i iniziali C omp one nte 1 2 14,3 13,74 14,94 15,04 15,06 13,36 15,91 15,67 14,17 13,46 14,74 13,75 14,66 13,23 13,08 12,78 11,95 13,46 12,71 13,05 12 12,27 12,35 12,78 12,95 14,14 13 12,89 13,12 14,08 13,93 14,62 12,45 12,4 1 .347 .761 .429 -.972 .890 2 .742 .409 -.820 .189 -3.76E-02 Met odo estra zione: ana lisi com ponenti principali. a. 2 componenti estrat ti Grafico decrescente degli autovalori 3.0 4 2.5 2 2.0 1.5 0 1.0 CLASSE .5 -2 Autovalore 3.00 0.0 1 2 Numero componente 3 4 5 REGR factor score 2 for analysis -4 -3 -2 -1 0 REGR factor score 1 for analysis 2.00 1 1.00 1 2 1 3 Esempio RI 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 Na 3,01 -0,39 -1,82 -0,34 -0,58 -2,04 -0,57 -0,44 1,18 -0,45 -2,29 -0,37 -2,11 -0,52 -0,37 -0,39 -0,16 3,96 1,11 -0,65 -0,5 1,66 -0,64 -0,49 -0,8 -0,36 -0,07 -0,79 -0,32 -0,16 -0,32 Mg 13,64 13,89 13,53 13,21 13,27 12,79 13,3 13,15 14,04 13 12,72 12,8 12,88 12,86 12,61 12,81 12,68 14,36 13,9 13,02 12,82 14,77 12,78 12,81 13,38 12,98 13,21 12,87 12,56 13,08 12,65 Al 4,49 3,6 3,55 3,69 3,62 3,61 3,6 3,61 3,58 3,6 3,46 3,66 3,43 3,56 3,59 3,54 3,67 3,85 3,73 3,54 3,55 3,75 3,62 3,57 3,5 3,54 3,48 3,48 3,52 3,49 3,56 Si 1,1 1,36 1,54 1,29 1,24 1,62 1,14 1,05 1,37 1,36 1,56 1,27 1,4 1,27 1,31 1,23 1,16 0,89 1,18 1,69 1,49 0,29 1,29 1,35 1,15 1,21 1,41 1,33 1,43 1,28 1,3 K 71,78 72,73 72,99 72,61 73,08 72,97 73,09 73,24 72,08 72,99 73,2 73,01 73,28 73,21 73,29 73,24 73,11 71,36 72,12 72,73 72,75 72,02 72,79 73,02 72,85 73 72,64 73,04 73,15 72,86 73,08 Ca 0,06 0,48 0,39 0,57 0,55 0,64 0,58 0,57 0,56 0,57 0,67 0,6 0,69 0,54 0,58 0,58 0,61 0,15 0,06 0,54 0,54 0,03 0,59 0,62 0,5 0,65 0,59 0,56 0,57 0,6 0,61 Ba 8,75 7,83 7,78 8,22 8,07 8,07 8,17 8,24 8,3 8,4 8,09 8,56 8,05 8,38 8,5 8,39 8,7 9,15 8,89 8,44 8,52 9 8,7 8,59 8,43 8,53 8,43 8,43 8,54 8,49 8,69 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 V ar ia nza tota le s pie ga ta A population of women at least 21 years old, of Pima Indian heritage and living near Phoenix, Arizona, was tested for diabetes according to World Health Organization criteria. C omp one nte 1 2 3 4 A uto valor i iniziali % di vari anza 3 4.41 8 2 1.37 8 1 3.02 7 1 1.42 8 9. 85 9 5. 56 9 4. 32 1 To tale 2 .409 1 .496 .912 .800 .690 .390 .302 5 6 7 % cu mul at a 34 .4 18 55 .7 96 68 .8 23 80 .2 51 90 .1 10 95 .6 79 1 00.00 0 2 .409 1 .496 .912 P es i d e i fatto ri non ruo tati % di var ianza 34 .4 18 21 .3 78 13 .0 27 6 7 T otale % cumul ata 3 4.41 8 5 5.79 6 6 8.82 3 M etod o di e str az io ne: An alisi co mpo ne nti pr incipali. Grafico decrescente degli autovalori 3.0 2.5 2.0 Npreg : number of pregnancies Glu : plasma glucose concentration in an oral glucose tolerance test Bp : diastolic blood pressure (mm Hg) Skin : triceps skin fold thickness (mm) Bmi : body mass index (weight in kg/(height in m)^2) Ped : diabetes pedigree function Age : age in years Type : Yes or No, for diabetic according to WHO criteria 1.5 1.0 .5 Autovalore 0.0 1 2 3 4 5 Numero componente REGR factor score 1 REGR factor score 2 Mat rice d icom po nenti a .567 .568 Component e 2 -.566 -1.56E-02 3 8.174E-02 .399 bp .640 -.132 -8.59E-02 sk in bmi .670 .607 .507 .627 -.322 -.230 ped age 4.522E-02 .732 .550 -.453 .750 .141 preg glu 1 Metodo es traz ione: analisi componenti princi pal i. a. 3 componenti estratt i REGR factor score 3 type 2.00 1.00 5 4 3 2 1 0 -1 VAR00009 -2 REGR factor score 3 for analysis -3 -3 -2 -1 0 1 REGR factor score 1 for analysis 2.00 1 1.00 2 1 3 4 Analisi delle corrispondenze Analisi delle corrispondenze È un metodo simile all’analisi delle componenti principali, ma per dati categoriali. Il punto di partenza è quindi una tabella di contingenza a doppia, tripla, … entrata. Il primo passo è standardizzare la tabella facendo in modo di avere le frequenze relative per ogni cella. (La somma delle frequenze sarà 1.) Analisi delle corrispondenze A NA tot B pB,A pB,NA pB. NB pNB,A pNB,NA pNB. tot p.A p.NA 1 • Massa : 1 unità di massa è distribuita all’interno della tabella. Analisi delle corrispondenze A NA tot B pB,A pB,NA pB. NB pNB,A pNB,NA pNB. tot p.A p.NA 1 Considerando le righe (colonne) come osservazioni statistiche si può procedere come per l’analisi delle componenti principali. • Massa di righe : totali di riga • Massa di colonne : totali di colonna • Inerzia : X2/n (corrisponde al concetto di varianza per variabili numeriche) Calcolo gli autovalori (loadings) e gli autovettori (scores) e disegno i punti nel nuovo sistema cartesiano. Esempio fair blue light medium dark tot red 326 688 343 98 1455 38 116 84 48 286 medium dark black tot 241 110 3 718 584 188 4 1580 909 412 26 1774 403 681 85 1315 2137 1391 118 5387 Esempio Biplot : Usualmente si disegnano i punti di riga e colonna nello stesso grafico. Attenzione: Si possono valutare solo distanze tra punti di righe e tra punti di colonne separatamente, ma non tra punti di riga e colonne. Si possono solo fare delle considerazioni qualitative sui possibili significati dei fattori. Esempio fair blue light medium dark tot 326 688 343 98 1455 red 38 116 84 48 286 medium dark black tot 241 110 3 718 584 188 4 1580 909 412 26 1774 403 681 85 1315 2137 1391 118 5387 Analisi delle corrispondenze multiple … e quando ci sono più di due variabili categoriali da analizzare? Costruisco una matrice disgiuntiva di tante righe quante sono le osservazioni e tante colonne quante sono le modalità di tutte le variabili considerate. Da una trasformazione di questa matrice (matrice di Burt) procediamo esattamente come abbiamo fatto per l’analisi delle componenti principali. Esempio Analisi dei gruppi Nodal Involvement in Prostate Cancer # Unsupervised methods (analisi interna) ANALISI CLUSTER (di raggruppamento, di segmentazione) tecnica descrittiva 1 : grave – 0 : non grave # Supervised methods (“machine learning”) ANALISI DISCRIMINANTE tecnica predittiva Grade : biopsia Xray : raggi X Acid : fosfatasi Stage : visita R : indice post operatorio Analisi cluster Var. 1 Var. 2 … Var. m 1 x11 x12 … x1m Identificazione di gruppi di soggetti con caratteristiche … … … … … simili n xn1 xn2 … xnm Simili rispetto a cosa ? Definizione di distanza m=3 n punti in uno spazio di m dimensioni Var 2 Var 3 Le osservazioni diventano punti nello spazio: punti vicini nello spazio sono raggruppati insieme. Ogni riga è un punto in uno spazio di m dimensioni Var 1 Misure di similarità o dissimilarità • Matrice dei dati • Matrice di similarità • Matrice di dissimilarità # # x 11 ... x i1 ... x n1 ... ... ... ... ... x 1f .. . x if .. . x nf . .. . .. . .. . .. . .. x 1p . .. x ip . .. x np $ 0 d % 2,1 & 0 d % 3,1 & d % 3,2 & 0 : : : d % n ,1 & d % n ,2 & . .. . . . 0 Alcune distanze Dati i vettori x = (x1, …, xn) e y = (y1, …, yn), definiamo: () n • distanza euclidea $ d E % x,y &' i=1 % x i* y i & 2 n • distanza di Manhattan d M % x,y &' ) +x i * y i+. i=1 • distanza di correlazione ) % x i* ,x &% y i * ,y & d C % x,y &'1* i=1 () % xi*,x &2 ) % y i* ,y &2 i=1 Alcune distanze . i=1 Multidimensional Scaling (MDS) • MDS algorithms work by finding coordinates in 2-D or 3-D space that preserve the distance ranking between the points in the high dimensional space. • The starting point of MDS algorithms is the distance or similarity matrix between the data points, which is followed by an optimisation algorithm. • MDS preserves the notion of nearness, and therefore clusters in the high dimensional space still look like cluster on an MDS plot. # 0 d % 2,1 & 0 d % 3,1 & d % 3,2 & 0 : : : d % n ,1 & d % n ,2 & . .. . . . 0 $ Esempio Multidimensional Scaling (MDS) 1. Calcola la matrice di dissimilarità. • Decidi il numero di dimensioni (k) in cui vuoi vedere i tuoi oggetti. 2. # Classe ED 0 d % 2,1 & 0 d % 3,1 & d % 3,2 & 0 : : : d % n ,1 & d % n ,2 & . .. . . . 0 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 $ Metti i punti in una configurazione iniziale magari inizializzata da una PCA. • Sposta iterativamente i punti nello spazio a k dimensioni in modo tale che le distanze tra gli oggetti e gli indici di dissimilarità iniziali siano più correlati possibili (funzione di stress). 3. KRH 15,8 11,29 13,28 13,45 13,15 11,51 13,96 14,16 13,82 11,66 13,9 11,5 13,35 14,05 11,88 11,78 10,31 11,87 13,31 12,05 12,17 10,3 10,29 10,26 11,31 12,32 11,05 11,13 11,1 12,32 13,67 13,06 12,37 14,3 13,74 14,94 15,04 15,06 13,36 15,91 15,67 14,17 13,46 14,74 13,75 14,66 13,23 13,08 12,78 11,95 13,46 12,71 13,05 12 12,27 12,35 12,78 12,95 14,14 13 12,89 13,12 14,08 13,93 14,62 12,45 STQN 12,54 14,03 14,58 14,18 14,72 14,76 14,69 15,3 14,66 18,42 15,81 18,49 16,54 16,32 19,11 18,72 19,37 17,87 19,36 20,77 19,22 16,18 16,14 16,32 17,02 19,94 21,18 20,93 21,15 20,67 19,19 19,49 21,19 AGP 21,86 22,55 18 18,19 17,85 23,99 16,67 15,13 18,95 15,77 16,75 15,85 16,92 19,61 17,2 18,17 22,68 21,35 18,11 17,4 23,92 28,06 27,88 27,46 24,72 15,1 17,77 18,25 17,65 14,72 14,83 14,36 17,52 Classificazione dei proteomi batterici attraverso la composizione aminoacidica LVIFMWYC 35,5 38,39 39,19 39,13 39,22 36,38 38,77 39,73 38,4 40,69 38,8 40,42 38,53 36,79 38,73 38,55 35,69 35,45 36,51 36,73 32,69 33,19 33,34 33,17 34 38,52 36,98 36,79 36,96 38,21 38,38 38,47 36,47 2.0 1.5 1.0 .5 0.0 • ED: acidi • KRH: basici • STQN: non carichi • AGP: non idrofobici • LVIFMWYC: idrofobici Continua fino a convergenza. 2.0 CLASS -.5 3.00 -1.0 2.00 DIM_2 -1.5 -2.0 1.00 -1.5 -1.0 -.5 0.0 .5 1.0 1.5 2.0 DIM_1 Analisi cluster 1.5 1.0 # .5 0.0 CLASS -.5 Risultato: dendrogramma 3.00 -1.0 2.00 DIM_2 -1.5 -2.0 1.00 2.0 -1.5 DIM_1 -1.0 -.5 0.0 .5 1.0 1.5 Metodi gerarchici: Il numero di cluster non è definito a priori. 2.0 # 4 1.5 1.0 2 Metodi non gerarchici: Il numero di cluster è definito a priori. Risultato: gruppi separati .5 0 0.0 # Metodi divisivi: Parto dalla radice per arrivare alle foglie. # Metodi agglomerativi: Parto dalle foglie per arrivare alle radici. CLASSE CLASS -.5 -2 3.00 -1.0 REGR factor score 2 for analysis -4 DIM_2 -1.5 -3 -2.0 -2 -1.5 -1 -1.0 0 -.5 0.0 REGR factor score 1 for analysis DIM_1 2.00 1 1.00 1 .5 2 1 1.0 3 1.5 3.00 2.00 1.00 2.0 ANALISI CLUSTER Dendrogramma non gerarchica radice gerarchica agglomerativi divisivi complete single foglie dell’albero average correlazione euclidea … Esempio Analisi CLUSTER gerarchica 4 3 1 – procedimento agglomerativo – 6 5 Punto di partenza : matrice di distanza 2 1 Prendo la distanza minima e collego i due punti corrispondenti. radice 2 3 1 2 3 4 foglie dell’albero 5 Ricalcolo la matrice di distanza. Prendo la distanza minima della nuova matrice di distanza e collego i due punti corrispondenti. 6 4 Ricalcolo la matrice di distanza. … … Esempio Matrice di distanza iniziale 1 2 3 4 5 1 0 1 2 1.2 1.3 2 1 0 7 2.01 4.5 3 2 7 0 3.2 4 4 1.2 2.01 3.2 0 1.1 5 1.3 4.5 4 1.1 0 A 3 4 5 A 0 4.5 1.6 2.9 3 4.5 0 3.2 4 4 1.6 3.2 0 1.1 5 2.9 4 1.1 0 A 3 B A 0 4.5 2.2 3 4.5 0 5.2 B 2.2 5.2 0 A 1 2 1 C 2 4 5 B A B A 1 2 4 C 3 C 0 4.8 3 4.8 0 1 2 4 5 3 5 Metodi NON gerarchici 1 (posiziono a caso 3 centri) Analisi CLUSTER non gerarchica – K-Means – Inizializzazione random dei centri dei clusters # Iterazione: " riassegna le osservazioni al centro più vicino " ricalcola le medie dei cluster # Termina a convergenza 2 (assegno i punti al centro più vicino) # 3 (ricalcolo i centri) 4 (riassegno i punti) Note: $ veloce $ Come selezionare k ? $ Ho gruppi e non relazioni tra gruppi. … fino a convergenza … ancora sulle distanze … Single Come definiamo la distanza tra gruppi? " single linkage " complete linkage " average linkage Complete dati originali Average … ancora sulle distanze … Complete " " Distanze diverse danno risultati diversi ! Definizione degli obiettivi dello studio Definizione della distanza Euclidea Correlazione di Pearson Single Livelli vs. Pattern … ancora sulle distanze … Var. 1 Var. 2 … Var. m 1 x11 x12 … x1m … … … … … n xn1 xn2 … xnm # Distanza euclidea : Raggruppa soggetti con livelli simili di valori delle variabili. Ogni profilo può essere inserito in un grafico … 1 X # Indice di correlazione di Pearson : Raggruppa soggetti con 2 andamenti simili dei valori delle variabili. 1 2 3 4 m Esempio Distanza euclidea Correlazione di Pearson Variabili Ross et al. (2000) Nature Gen. 24: 227-235 Esempio La classificazione molecolare dei tumori – I linfomi • Lymphochip -18,000 trascritti • 46 DLBCL, 9 FL, 11 CLL • 96 esperimenti in tutto • esperimenti di controllo Alizadeh et al. (2000) Nature 403: 503-511 • Le tre patologie DLBCL, FL e CLL sono separate chiaramente. • CLL e FL sono raggruppate insieme a “resting B cell”. . tumori relativamente lenti • La maggiore differenza tra CLL e FL si ha nel profilo “germinal centre B”. Curve di Kaplan-Meier (curve di sopravvivenza) • Focalizzandosi sui geni del profilo “germinal centre B” riescono ad evidenziare 2 grandi gruppi tra i DLBCL. • Questi due gruppi sono evidenziati dall’espressione differenziale di centinaia di geni. • I due gruppi sembrano essere correlati a diversi stadi di differenziazione e attivazione delle cellule tipo B. Alizadeh et al. (2000) Nature 403: 503-511 Analisi discriminante (Machine Learning) Alizadeh et al. (2000) Nature 403: 503-511 • I due gruppi di DLBCL devono essere considerati come due malattie distinte, senza però eliminare l’ipotesi di ulteriori divisioni al loro interno. • I profili evidenziati potrebbero essere usati come indicatori prognostici della patologia. • Non sono ancora stati identificati geni particolarmente correlati ad una migliore risposta al trattamento chemioterapico. Analisi discriminante " SUPPORT VECTOR MACHINE (SVM) Perché ? Fase di calibrazione Perché imparano Training set " LINEAR DISCRIMINANT ANALYSIS (LDA) " QUADRATIC DISCRIMINANT ANALYSIS " NEURAL NETWORKS (NNET) NON parametrici Parametrici " RECURSIVE PARTITIONING (RPART) Fase di predizione Test set " k-NEAREST NEIGHBOUR Problema: $ Parametrici: Fanno forti assunzioni sui dati (normalità, varianza Se il training set è troppo diverso dal test set, l’analisi non è robusta. robusta $ Non parametrici: Non fanno assunzioni sui dati, ma hanno spesso uguale, …). bisogno di un un training set molto grande. Analisi discriminante lineare Analisi discriminante lineare vs. Analisi delle componenti principali Funzione discriminante In base al valore della funzione discriminante si classifica una nuova osservazione in una delle due classi iniziali. Analisi discriminante quadratica Funzione discriminante In base al valore della funzione discriminante si classifica una nuova osservazione in una delle due classi iniziali. Il metodo stima la varianza all’interno di ogni classe, quindi bisogna avere un numero elevato di untià statistiche per classe. % LDA utilizza una combinazione lineare delle variabili iniziali in modo da separare i due gruppi. % PCA utilizza combinazioni lineari delle variabili originali per catturare porzioni diverse di variabilità dei dati. Confusione Matrice di confusione Matrice dei dati Il risultato di un’analisi discriminante è generalmente riportato sotto forma di matrice di confusione e di errore di classificazione. classificazione Matrice iniziale x1,2 x1,3 object 2 object 3 ... object i object i+1 object i+2 ... object n-1 object n Predictor variables Criterion variable variable 2 ... variable p class x2,1 xp,1 ... A x2,2 xp,2 ... A x2,3 x ... A p,3 ... x1,i ... x2,i ... ... ... xp,i ... B x1,i+1 x1,i+2 ... x1,n-1 x1,n x2,i+1 x2,i+2 ... x2,n-1 x2,n ... xp,i+1 xp,i+2 B xp,n-1 xp,n K ... ... ... predetti originali variable 1 x1,1 object 1 B K variable 1 x1,1 x1,2 x1,3 object 2 object 3 ... object i object i+1 object i+2 ... object n-1 object n 1 2 … n 3 2 … 0 2 1 5 … 1 … … … … … n 1 0 … 5 Classificazione errata Matrice finale object 1 1 Predictor variables variable 2 ... variablep x2,1 xp,1 ... x2,2 xp,2 ... x2,3 xp,3 ... A B A ... K 1 2 3 xp,i+1 xp,i+2 B B B B 1 3 2 0 xp,n-1 xp,n K K K K 2 1 5 1 3 1 0 5 ... x2,i ... ... ... xp,i x1,i+1 x1,i+2 ... x1,n-1 x1,n x2,i+1 x2,i+2 ... x2,n-1 x2,n ... ... ... Somma delle classificazioni errate Totale unità statistiche Error rate = A ... B ... x1,i ... C riterionvariable know n predicted A A Classificazione corretta Error rate = 5/18 = 0.27 = 27% Applicazione Matrice di confusione I metodi discriminanti sono altamente dipendenti dalle classi definite dall’utente. L’errata classificazione potrebbe essere indice di: % errata divisione in classi; % training set errato; % effettiva omogeneità dei dati. In alcuni casi la matrice di confusione può dare degli indizi sulle similarità delle osservazioni statistiche. Alu originali predetti 1 2 … n 1 3 2 … 0 2 1 5 … 1 … … … … … n 1 0 … 5 A volte la classe predetta potrebbe avere un significato biologico. Y Matrici di confusione Applicazione Applicazion van’t Veer et al. (2002) Nature 415: 530-536 • Chemioterapia e terapia ormonale riduce di un terzo il rischio di recidive. • 70%-80% delle pazienti che ricevono questi trattamenti avrebbero comunque avuto un esito positivo. • 98 primary breast cancer Gli errori di classificazione sembrano ricalcare molto bene le linee migratorie dell’Homo Sapiens ipotizzate attraverso studi antropologici. • 34 distant metastasis, 44 disease-free, 18 con mutazioni in BRCA1, 2 con mutazioni su BRCA2 • Distant metastasis: comparsa di metastasi nei cinque anni successivi alla mastectomia Prima analisi esplorativa su tutti i campioni disponibili • La presenza di cellule tumorali nei linfonodi al momento della diagnosi è considerato un elemento aggravante per la prognosi. • 78 casi lymph-node negative, 44 disease-free, 34 distant metastasis van’t Veer et al. (2002) Nature 415: 530-536 • Con 70 geni ottengono un errore di class. del 10%.









