MINISTERO DELL'UNIVERSITÀ E DELLA RICERCA
DIREZIONE GENERALE PER IL COORDINAMENTO E LO SVILUPPO DELLA RICERCA
PROGRAMMI DI RICERCA SCIENTIFICA DI RILEVANTE INTERESSE NAZIONALE
RICHIESTA DI COFINANZIAMENTO (DM n. 1175 del 18 settembre 2007)
PROGETTO DI UNITÀ DI RICERCA - MODELLO B
Anno 2007 - prot. 20079JLCRZ_003
1 - Area Scientifico-disciplinare
06: Scienze mediche 70% *
05: Scienze biologiche 30%
* Area prescelta ai fini della valutazione
2 - Durata del Progetto di Ricerca
24 Mesi
3 - Coordinatore Scientifico
MARTELLI
ALBERTO MARIA
Professore Ordinario
Università degli Studi di BOLOGNA
Facoltà di FARMACIA
Dipartimento di SCIENZE ANATOMICHE UMANE E FISIOPATOLOGIA DELL'APPARATO LOCOMOTORE
4 - Responsabile dell'Unità di Ricerca
MARTINELLI
GIOVANNI
Professore Associato non confermato
30/05/1960
MRTGNN60E30M052J
051/6364037
(Numero fax)
[email protected]
Università degli Studi di BOLOGNA
Istituto di UCI - CARDIOLOGIA ED EMATOLOGIA
051/6363829
(Prefisso e telefono)
5 - Curriculum scientifico
Testo italiano
Giovanni Martinelli, nato a Vimercate (MI) il 30/05/1960.
Laureato in Medicina e Chirurgia, presso l'Università degli Studi di Verona nel 1985. Specializzazione in Ematologia Generale (Clinica e Laboratorio), Università
degli Studi di Verona nel 1988. Specializzazione in Genetica Medica (Indirizzo Medico), Università degli Studi di Verona nel 1992.
Idoneità al ruolo di Professore Associato per il settore scientifico disciplinare F07G - Malattie del Sangue, presso la Facoltà di Medicina e Chirurgia dell'Università
degli Studi di Milano il 20 luglio del 2001.
Dal 1994 ad oggi, Medico di I Livello dirigenziale fascia B di Ruolo a Tempo Pieno di Ematologia, presso l'Azienda Ospedaliera di Bologna Policlinico S.
Orsola-Malpighi - Istituto di Ematologia e Oncologia Medica "L. & A. Seràgnoli".
Responsabile del Laboratorio di Biologia Molecolare presso l'Istituto Seràgnoli.
Dal 01/03/2005, Professore di II fascia, MED/15 - Malattie del Sangue, presso l'Università degli Studi di Bologna.
E' stato Professore a contratto presso la Scuola di Specializzazione in Ematologia (Prof. M. Cavo) e la Scuola di Specializzazione in Genetica Applicata (Prof. G.
Della Valle).
E' stato cooperatore nei seguenti progetti di ricerca MURST/COFIN del Prof. Baccarani:
Anno 2001 - Modello A: "terapia molecolare delle leucemie Philadelphia positive"
Anno 2001 - Modello B: "Gli inibitori della trasduzione del segnale (tirosina-chinasi e farnesil transferasi) e gli interferoni nella terapia delle leucemie Ph positive:
basi molecolari e studio clinico"
Anno 2002 - Modello B: "Leucemie acute secondarie alle sindromi mieloproliferative croniche: un modello di leucemogenesi"
Anno 2003 - Modello A: "Le tirosino-chinasi come bersaglio molecolare della terapia delle leucemie mieloidi croniche e acute"
Anno 2003 - Modello B: "Studi preclinici e clinici delle alterazioni delle tirosin-chinasi (TK) e degli effetti terapeutici degli inibitori delle TK nella leucemia mieloide
cronica (LMC) e nelle sindromi correlate".
Nei progetti di Ricerca fondamentale orientata (ex 60%) del Prof. Baccarani:
Anni 2002 - 2003 - 2004: "STI571 nella Leucemia Mieloide Cronica: identificazione dei fattori prognostici clinici e biomolecolari"
MUR - BANDO 2007 - * MODELLO B *
-1-
Ministero dell ,Università e della Ricerca
Anno 2002 - 2003: "Terapia molecolare delle leucemie mieloblastiche acute"
nei progetti FIRB del Prof. Baccarani:
Anno 2001: "Gli inibitori del segnale nella terapia delle leucemie PH1+"
Anno 2003: "Piattaforme diagnostico-molecolari per la caratterizzazione delle alterazioni genetiche e dell'espressione genica nelle neoplasie umane: impatto su
prognosi e terapia"
Principal Investigator per l'AIRC - Grant Proposal 2004:
"Tyrosine-kinases as molecular target of the treatment of Ph+ chronic and acute myeloid leukemias"
Titolare del progetto di ricerca European LeukemiaNet WP 4 CML.
Ha pubblicato n° 150 lavori scientifici su riviste nazionali ed internazionali.
Coordinatore del progetto FIRB 2006: "Studio e trattamento dei tumori e delle malattie degenerative: sviluppo e produzione di una nuova piattaforma analitica in
DHPLC (Denaturing High Pressure Liquid Chromatography) completa di test diagnostici dedicati ai differenti settori applicativi in oncologia e nelle malattie
degenerative".
Ha partecipato a congressi scientifici nazionali ed internazionali delle Società quali SIE, SIES, EBMT, EHA, AACR, ASH, con la sottomissione di numerosi abstracts
come primo autore e co-autore presentati come poster e come relazioni orali.
E' socio della Società Italiana di Ematologia (SIE) e della Società Italiana di Ematologia Sperimentale SIES), dell'European Haematology Association (EHA),
dell'American Society of Hematology (ASH), dell'American Association for Cancer Research (AACR), dell'American Society for Microbiology (ASM), dell'American
Association for the Advancement of Science (AAAS) e dell'International Society for Cellular Therapy (ISCT).
E' membro del Gruppo Italiano Malattie Ematologiche dell'Adulto (GIMEMA).
Revisore per le seguenti riviste internazionali: Leukemia, Blood, Haematologica.
Collaborazioni internazionali con la Dr.ssa Giuseppina Nucifora e la Dr.ssa Silvia Buonamici del Department of Pathology and Cancer Center, University of Illinois,
Chicago, USA, sul gene EVI1;
con il Dr. Bruno Calabretta del Department of Microbiology and Immunology, Kimmel Cancer Center, Thomas Jefferson Medical College, Philadelphia, USA su
CML.
Sono previsti i seguenti progetti di studio:
"An open-label Phase II trial of CEP-701 in older patients with Acute Myeloid Leukaemia not considered fit for intensive chemotherapy"
"A Phase II Study of SKI-606 in Subjects with Chronic Myeloid Leukemia or Acute Lymphoblastic Leukemia Ph+ positive resistant, refractory or intolerant to
Imatinib Mesylate"
"A phase II study of ZARNESTRA R-115777 plus Velcade in the treatment of patients with refractory or resistant acute myeloid leukemia (AML), or not amenable to
conventional therapy for the disease"
"A phase II study of AZD0530 in the treatment of patients with Chronic Myeloid Leukemia (CML) Ph+ or ALL Ph+, resistant, relapsed or refractory to imatinib
therapy"
"A pilot open label phase IIa study of SU011248 plus conventional intensive chemotherapy including Aracytin and Daunorubicin ("3+7") in the treatment of patients
over 60 years with de novo acute myeloid leukemia (AML), with FLT3 receptor length and/or point mutation"
"A phase II study of SU011248 in the treatment of patients over 65 years with de novo acute myeloid leukemia (AML), with refractory or resistant AML, or not
amenable to conventional therapy for the disease, with FLT3 receptor length and/or point mutation"
"A phase I-II study of Bortezomib (PS341-Velcade) plus Imatinib in the treatment of patients over 18 years with Chronic Myeloid Leukemia (CML) Ph+, or Acute
Lymphoblastic Leukemia (ALL) Ph+ in progression, resistant or refractory to imatinib therapy alone"
"A randomized Phase II Study of BMS-354825 plus steroid alone versus BMS-354825 plus steroid followed by intensive chemotherapy in Subjects with De novo
Philadelphia Chromosome Positive Acute Lymphoblastic Leukemia"
"In vitro growth inhibition and induction of apoptosis of acute myelogenous leukaemia (AML) and acute lymphoblastic leukaemia (ALL) Ph+ positive cell by
AZD1152"
"An open-Label, Single-Arm, Multi-Center, Phase II study of Troxatyl (troxacitabine) administered by continuous infusion in patients with Acute Myeloid Leukemia
(AML) in second salvage"
Sono attivati i seguenti protocolli di studio:
Open label, pilot phase II study of STI571 in the treatment of patients with idiopathic hypereosinophilic syndrome (HES) and eosinophilic leukemias (Protocol No.
HES0203)
A phase II study of velcade (Bortezomib -PS341) in the treatment of patients over 18 years with ph+ chronic myeloid leukemia (CML). (Study Number: CML 033).
Intensificazione della terapia post-remissionale nella leucemia acuta linfoide dell'adulto ad alto rischio di recidiva e monitoraggio della malattia minima residua.
(Prot. GIMEMA LAL 0904)
Terapia con Alemtuzumab (MabCampath) per pazienti adulti affetti da leucemia linfoblastica acuta (LAL) in recidiva o refrattaria. Studio di fase II prospettico,
multicentrico, non randomizzato. (Prot. GIMEMA LAL 1004)
An open label, phase II, non randomized, clinical trial of chemotherapy treatment with 5-Azacytidine plus atra plus valproic acid for patients diagnosed with
intermediate II and high risk Myelodisplastic syndrome (MDS) or resistant, refractory, relapsed, or adverse cytogenetic acute myeloid leukemia (AML). (Protocol
AML-MDS 1305)
THESIS: (Treatment of HES: International Study). Hypereosinophilic syndrome (HES): a multicentre, international, retrospective and observational study of Institute
of Hematology and Medical Oncology "L. e A. Seragnoli" Bologna, Italy for European LeukemiaNet, Heosinophilia Study Group
Aree principali di ricerca: leucemie acute, leucemia mieloide cronica: ricerca di terapie mirate nelle principali malattie ematologiche: terapia delle leucemie acute,
terapia delle leucemie croniche Ph+ e non e Sindrome Ipereosinofila (HES).
Testo inglese
Giovanni Martinelli, born in Vimercate (MI) on 30/05/1960.
Degree University of Verona Medical School, 1985. Postgraduate Specialization School in Hematology, Università of Verona, 1988. Postgraduate Specialization
School in Medical Genetics, University of Verona, 1992.
Qualification to the role of Associate University Professor, scientific field disciplinary F07G - Malattie del Sangue, on the University of Milan, July 20, 2001.
From 1994 to present, Medical Doctor full-time on the Institute of Hematology and Medical Oncology "L. & A. Seràgnoli", Azienda Ospedaliera S.Orsola-Malpighi.
Responsible for the Laboratory of Molecular Biology, Seràgnoli Institute.
From 01/03/2005, Associate Professor, MED/15 - Malattie del Sangue, University of Bologna.
Professor in the Post-graduate School of Hematology (Prof. M. Cavo) and Post-graduate School of Hematological Genetic (Prof. G. Della Valle).
Co-operator in the following Research projects MURST/COFIN of Prof. Baccarani:
Year 2001 - Modello A: "Positive molecular treatment of Philadelphia positive leukemias"
Year 2001 - Modello B: "The molecular bases and clinical applications of signal transduction inhibitors (tyrosine-kinase and farnesyltransferase) and interferons in
the treatment of Ph-positive leukemias"
Year 2002 - Modello B: "Acute leukemia secondary to chronic myeloproliferative disorders: a model of leukemogenesis"
Year 2003 - Modello A: "Tyrosine-kinases as molecular target of the treatment of chronic and acute myeloid leukemias"
Year 2003 - Modello B: "preclinical and clinical studies on tyrosine-kinases (TK) abnormalities and on the therapeutic effects of TK inhibitors in chronic myeloid
leukemia (CML) and related disorders"
In the projects "Ricerca fondamentale orientata (ex 60%)" of Prof. Baccarani:
Years 2002, 2003, 2004: "STI571 nella Leucemia Mieloide Cronica:identificazione dei fattori prognostici clinici e biomolecolari"
Years 2002, 2003: "Terapia molecolare delle leucemie mieloblastiche acute"
FIRB Projects:
Year 2001: "Signal transduction inhibitors in Ph1+ leukemias"
Year 2003: "Molecular and diagnostic platforms for the characterization of the tumor associated genetic aberations and gene expression: their impact on the
prognosis and therapy"
Coordinator of FIRB 2006 project: "Analysis and treatment of tumours and degenerative diseases: development and production of a new analytic platform in DHPLC
(Denaturing High Pressure Liquid Chromatography) with diagnostic kits dedicated to different fields in oncology and in degenerative diseases".
Principal Investigator AIRC Grant Proposal 2004:
"TYROSINE-kinases as molecular target of the treatment of Ph+ chronic and acute myeloid leukemias".
Responsible for the European LeukemiaNet WP 4 CML project.
Participation at national and international Scientific Congress as SIE, SIES, EBMT, EHA, AACR, ASH. He has presented several abstracts either as first author or
co-author, both as oral presentations and poster.
He has published n. 150 scientific papers on national and international journals.
MUR - BANDO 2007 - * MODELLO B *
-2-
Ministero dell ,Università e della Ricerca
Member of Società Italiana di Ematologia (SIE) and Società Italiana di Ematologia Sperimentale SIES), of European Haematology Association (EHA), of the
American Society of Hematology (ASH), of the American Association for Cancer Research (AACR), of the American Society of for Microbiology (ASM), of the
American Association for the Advancement of Science (AAAS) and of the International Society for Cellular Therapy (ISCT). Member of the Gruppo Italiano Malattie
Ematologiche dell'Adulto (GIMEMA).
Reviewer for international journals: Leukemia, Blood, Haematologica.
International Co-operation with Dr.ssa Giuseppina Nucifora and Dr.ssa Silvia Buonamici of Department of Pathology and Cancer Center, University of Illinois,
Chicago, USA, on gene EVI1;
with Dr. Bruno Calabretta of the Department of Microbiology and Immunology, Kimmel Cancer Center, Thomas Jefferson Medical College, Philadelphia, USA on
CML.
These study projects are in process:
"An open-label Phase II trial of CEP-701 in older patients with Acute Myeloid Leukaemia not considered fit for intensive chemotherapy"
"A Phase II Study of SKI-606 in Subjects with Chronic Myeloid Leukemia or Acute Lymphoblastic Leukemia Ph+ positive resistant, refractory or intolerant to
Imatinib Mesylate"
"A phase II study of ZARNESTRA R-115777 plus Velcade in the treatment of patients with refractory or resistant acute myeloid leukemia (AML), or not amenable to
conventional therapy for the disease"
"A phase II study of AZD0530 in the treatment of patients with Chronic Myeloid Leukemia (CML) Ph+ or ALL Ph+, resistant, relapsed or refractory to imatinib
therapy"
"A pilot open label phase IIa study of SU011248 plus conventional intensive chemotherapy including Aracytin and Daunorubicin ("3+7") in the treatment of patients
over 60 years with de novo acute myeloid leukemia (AML), with FLT3 receptor length and/or point mutation"
"A phase II study of SU011248 in the treatment of patients over 65 years with de novo acute myeloid leukemia (AML), with refractory or resistant AML, or not
amenable to conventional therapy for the disease, with FLT3 receptor length and/or point mutation"
"A phase I-II study of Bortezomib (PS341-Velcade) plus Imatinib in the treatment of patients over 18 years with Chronic Myeloid Leukemia (CML) Ph+, or Acute
Lymphoblastic Leukemia (ALL) Ph+ in progression, resistant or refractory to imatinib therapy alone"
"A randomized Phase II Study of BMS-354825 plus steroid alone versus BMS-354825 plus steroid followed by intensive chemotherapy in Subjects with De novo
Philadelphia Chromosome Positive Acute Lymphoblastic Leukemia"
"In vitro growth inhibition and induction of apoptosis of acute myelogenous leukaemia (AML) and acute lymphoblastic leukaemia (ALL) Ph+ positive cell by
AZD1152"
"An open-Label, Single-Arm, Multi-Center, Phase II study of Troxatyl (troxacitabine) administered by continuous infusion in patients with Acute Myeloid Leukemia
(AML) in seconde salvage"
Ongoing protocols:
Open label, pilot phase II study of STI571 in the treatment of patients with idiopathic hypereosinophilic syndrome (HES) and eosinophilic leukemias (Protocol No.
HES0203)
A phase II study of velcade (Bortezomib -PS341) in the treatment of patients over 18 years with ph+ chronic myeloid leukemia (CML). (Study Number: CML 033)
Intensificazione della terapia post-remissionale nella leucemia acuta linfoide dell'adulto ad alto rischio di recidiva e monitoraggio della malattia minima residua.
(Prot. GIMEMA LAL 0904)
Terapia con Alemtuzumab (MabCampath) per pazienti adulti affetti da leucemia linfoblastica acuta (LAL) in recidiva o refrattaria. Studio di fase II prospettico,
multicentrico, non randomizzato. (Prot. GIMEMA LAL 1004)
An open label, phase II, non randomized, clinical trial of chemotherapy treatment with 5-Azacytidine plus atra plus valproic acid for patients diagnosed with
intermediate II and high risk Myelodisplastic syndrome (MDS) or resistant, refractory, relapsed, or adverse cytogenetic acute myeloid leukemia (AML). (Protocol
AML-MDS 1305)
THESIS: (Treatment of HES: International Study). Hypereosinophilic syndrome (HES): a multicentre, international, retrospective and observational study of Institute
of Hematology and Medical Oncology "L. e A. Seragnoli" Bologna, Italy for European LeukemiaNet, Heosinophilia Study Group
Main fields of research: acute leukemia, chronic myeloid leukemia: research of target therapies in the major haematological syndromes: therapy of acute leukemias,
therapy of chronic myeloid leukemias Ph+ and non Ph+, and therapy of Hypereosinophilic syndrome (HES).
6 - Pubblicazioni scientifiche più significative del Responsabile dell'Unità di Ricerca
1. BACCARANI M, CILLONI D, RONDONI M, OTTAVIANI E, MESSA F, MERANTE S, TIRIBELLI M, BUCCISANO F, TESTONI N, GOTTARDI
E, DE VIVO A, GIUGLIANO E, IACOBUCCI I, PAOLINI S, SOVERINI S, ROSTI G, RANCATI F, ASTOLFI C, PANE F, SAGLIO G,
MARTINELLI G. (2007). The efficacy of imatinib mesylate in patients with FIP1L1-PDGFRalpha-positive hypereosinophilic syndrome. Results
of a multicenter prospective study. HAEMATOLOGICA. vol. 92(9), pp. 1173 - 1179 ISSN: 0390-6078. [ARTICOLO].
2. CILLONI D, MARTINELLI G., MESSA F, BACCARANI M, SAGLIO G. (2007). Nuclear factor kB as a target for new drug development in
myeloid malignancies. HAEMATOLOGICA. vol. 92(9), pp. 1224 - 1229 ISSN: 0390-6078. [ARTICOLO].
3. CILLONI D, MESSA F, MARTINELLI G., GOTTARDI E, ARRUGA F, DEFILIPPI I, CARTURAN S, MESSA E, FAVA M, GIUGLIANO E,
ROSSO V, CATALANO R, MERANTE S, NICOLI P, RONDONI M, OTTAVIANI E, SOVERINI S, TIRIBELLI M, PANE F, BACCARANI M,
SAGLIO G. (2007). WT1 transcript amount discriminates secondary or reactive eosinophilia from idiopathic hypereosinophilic syndrome or
chronic eosinophilic leukemia. LEUKEMIA. vol. 21(7), pp. 1442 - 1450 ISSN: 0887-6924. [ARTICOLO].
4. FOLLO MY, MONGIORGI S, BOSI C, CAPPELLINI A, FINELLI C, CHIARINI F, PAPA V, LIBRA M, MARTINELLI G., COCCO L, MARTELLI
AM. (2007). The Akt/mammalian target of rapamycin signal transduction pathway is activated in high-risk myelodysplastic syndromes and
influences cell survival and proliferation. CANCER RESEARCH. vol. 67(9), pp. 4287 - 4294 ISSN: 0008-5472. [ARTICOLO].
5. MALAGOLA M, DAMIANI D, MARTINELLI G., MICHELUTTI A, CESANA B, VIVO AD, PICCALUGA PP, OTTAVIANI E, CANDONI A,
GEROMIN A, TIRIBELLI M, FANIN R, TESTONI N, LAURIA F, BOCCHIA M, GOBBI M, PIERRI I, ZACCARIA A, ZUFFA E, MAZZA P,
PRICCOLO G, GUGLIOTTA L, BONINI A, VISANI G, SKERT C, BERGONZI C, ROCCARO AM, FILI C, BACCARANI M, RUSSO D. (2007).
Case-control study of multidrug resistance phenotype and response to induction treatment including or not fludarabine in newly diagnosed acute
myeloid leukaemia patients. BRITISH JOURNAL OF HAEMATOLOGY. vol. 136, pp. 87 - 95 ISSN: 0007-1048. [ARTICOLO].
6. MARTINELLI G. (2007). Atypical chronic myeloproliferative disorders: genes and imatinib-sensitive targets. SEMINARS IN HEMATOLOGY.
vol. 44 (Suppl 2), pp. S1 - S3 ISSN: 0037-1963. [ARTICOLO].
7. MARTINELLI G., RONDONI M, OTTAVIANI E, PAOLINI S, BACCARANI M. (2007). Hypereosinophilic syndrome and molecularly targeted
therapy. SEMINARS IN HEMATOLOGY. vol. 44 (Suppl 2), pp. S4 - S16 ISSN: 0037-1963. [ARTICOLO].
8. SOVERINI S, COLAROSSI S, GNANI A, CASTAGNETTI F, ROSTI G, BOSI C, PAOLINI S, RONDONI M, PICCALUGA PP, PALANDRI F,
GIANNOULIA P, MARZOCCHI G, LUATTI S, TESTONI N, IACOBUCCI I, CILLONI D, SAGLIO G, BACCARANI M, MARTINELLI G. (2007).
Resistance to dasatinib in Philadelphia-positive leukemia patients and the presence or the selection of mutations at residues 315 and 317 in the
BCR-ABL kinase domain. HAEMATOLOGICA. vol. 92(3), pp. 401 - 404 ISSN: 0390-6078. [ARTICOLO].
9. SOVERINI S, IACOBUCCI I, BACCARANI M, MARTINELLI G. (2007). Targeted therapy and the T315I mutation in Philadelphia-positive
leukemias. HAEMATOLOGICA. vol. 92(4), pp. 437 - 439 ISSN: 0390-6078. [ARTICOLO].
10. SOVERINI S, MARTINELLI G., COLAROSSI S, GNANI A, RONDONI M, CASTAGNETTI F, PAOLINI S, ROSTI G, BACCARANI M. (2007).
Second-line treatment with dasatinib in patients resistant to imatinib can select novel inhibitor-specific BCR-ABL mutants in Ph+ ALL.
LANCET ONCOLOGY. vol. 8(3), pp. 273 - 274 ISSN: 1470-2045. [ARTICOLO].
11. TAZZARI P.L., CAPPELLINI A., RICCI F., EVANGELISTI C., PAPA V., GRAFONE T., MARTINELLI G., CONTE R., COCCO L., MCCUBREY
J.A., MARTELLI A.M. (2007). Multidrug resistance-associated protein 1 expression is under the control of the phosphoinositide 3 kinase/Akt
signal transduction network in human acute myelogenous leukemia blasts. LEUKEMIA. vol. 21, pp. 427 - 438 ISSN: 0887-6924. [ARTICOLO].
12. TAZZARI PL, TABELLINI G, BORTUL R, PAPA V, EVANGELISTI C, GRAFONE T, MARTINELLI G., MCCUBREY JA, MARTELLI AM.
(2007). The insulin-like growth factor-I receptor kinase inhibitor NVP-AEW541 induces apoptosis in acute myeloid leukemia cells exhibiting
MUR - BANDO 2007 - * MODELLO B *
-3-
Ministero dell ,Università e della Ricerca
autocrine insulin-like growth factor-I secretion. LEUKEMIA. vol. 21, pp. 886 - 896 ISSN: 0887-6924. [ARTICOLO].
13. CILLONI D, MESSA F, ARRUGA F, DEFILIPPI I, MOROTTI A, MESSA E, CARTURAN S, GIUGLIANO E, PAUTASSO M, BRACCO E,
ROSSO V, SEN A, MARTINELLI G., BACCARANI M, SAGLIO G. (2006). The NF-kappaB pathway blockade by the IKK inhibitor PS1145 can
overcome Imatinib resistance. LEUKEMIA. vol. 20, pp. 61 - 67 ISSN: 0887-6924. [ARTICOLO].
14. IACOBUCCI I, ROSTI G, AMABILE M, POERIO A, SOVERINI S, CILLONI D, TESTONI N, ABRUZZESE E, MONTEFUSCO E, OTTAVIANI
E, IULIANO F, RUSSO D, GOBBI M, ALIMENA G, MARTINO B, TERRAGNA C, PANE F, SAGLIO G, BACCARANI M, MARTINELLI G.
(2006). Comparison between patients with Philadelphia-positive chronic phase chronic myeloid leukemia who obtained a complete cytogenetic
response within 1 year of imatinib therapy and those who achieved such a response after 12 months of treatment. JOURNAL OF CLINICAL
ONCOLOGY. vol. 24(3), pp. 454 - 459 ISSN: 0732-183X. [ARTICOLO].
15. NYÅKERN M, TAZZARI PL, FINELLI C, BOSI C, FOLLO MY, GRAFONE T, PICCALUGA PP, MARTINELLI G., COCCO L, MARTELLI AM.
(2006). Frequent Elevation of Akt Kinase Phosphorylation in Blood Marrow and Peripheral Blood Mononuclear Cells from High Risk
myelodysplastic Syndrome Patients. LEUKEMIA. vol. 20, pp. 230 - 238 ISSN: 0887-6924. [ARTICOLO].
16. SOVERINI S., TASCO G., GRAFONE T., COLAROSSI S., GNANI A., ROSTI G., CASTAGNETTI F., PALANDRI F., RONDONI M., POERIO A.,
IACOBUCCI I., AMABILE M., BACCARANI M., CASADIO R., MARTINELLI G. (2006). Binding Mode Of The Novel Dual SRC and ABL
Inhibitor SKI-606 To The BCR-ABL Kinase As Predicted By Molecular Docking Studies. HAEMATOLOGICA. vol. 91(s3), pp. 31 ISSN:
0390-6078. [ATTO DI CONVEGNO].
17. TAGLIAFICO E, TENEDINI E, MANFREDINI R, GRANDE A, FERRARI F, RONCAGLIA E, BICCIATO S, ZINI R, SALATI S, BIANCHI E,
GEMELLI C, MONTANARI M, VIGNUDELLI T, ZANOCCO-MARANI T, PARENTI S, PAOLUCCI P, MARTINELLI G., PICCALUGA PP,
BACCARANI M, SPECCHIA G, TORELLI U, FERRARI S. (2006). Identification of a molecular signature predictive of sensitivity to
differentiation induction in acute myeloid leukemia. LEUKEMIA. vol. 20, pp. 1751 - 1758 ISSN: 0887-6924. [ARTICOLO].
18. VISANI G, OLIVIERI A, MALAGOLA M, BRUNORI M, PICCALUGA PP, CAPELLI D, POMPONIO G, MARTINELLI G., ISIDORI A,
SPARAVENTI G, LEONI P. (2006). Consolidation therapy for adult acute myeloid leukemia: A systematic analysis according to evidence based
medicine. LEUKEMIA & LYMPHOMA. vol. 47, pp. 1091 - 1102 ISSN: 1042-8194. [ARTICOLO].
19. CAPPELLINI A, MANTOVANI I, TAZZARI PL, GRAFONE T, MARTINELLI G., COCCO L, MARTELLI AM. (2005). Application of flow
cytometry to molecular medicine: Detection of tumor necrosis factor-related apoptosis-inducing ligand receptors in acute myeloid leukaemia
blasts. INTERNATIONAL JOURNAL OF MOLECULAR MEDICINE. vol. 16, pp. 1041 - 1048 ISSN: 1107-3756. [ARTICOLO].
20. G. TABELLINI, P.L. TAZZARI, R. BORTUL, C. EVANGELISTI, A.M. BILLI, T. GRAFONE, MARTINELLI G., M. BACCARANI, A.M.
MARTELLI. (2005). Phosphoinositide 3-kinase/Akt inhibition increases arsenic trioxide-induced apoptosis of acute promyelocytic and T-cell
leukaemias. BRITISH JOURNAL OF HAEMATOLOGY. vol. 130, pp. 716 - 725 ISSN: 0007-1048. [ARTICOLO].
21. MALAGOLA M, MARTINELLI G., RONDONI M, PAOLINI S, GAITANI S, ARPINATI M, PICCALUGA PP, AMABILE M, BASI C, OTTAVIANI
E, CANDONI A, GOTTARDI E, CILLONI D, BOCCHIA M, SAGLIO G, LAURIA F, FANIN R, VISANI G, MARRE MC, MADERNA M,
RANCATI F, VINACCIA V, RUSSO D, BACCARANI M. (2005). Imatinib mesylate in the treatment of c-kit-positive acute myeloid leukemia: is
this the real target?. BLOOD. vol. 105(2), pp. 904 ISSN: 0006-4971. [ARTICOLO].
22. R. BORTUL, P.L. TAZZARI, A.M. BILLI, G. TABELLINI, I. MANTOVANI, A. CAPPELLINI, T. GRAFONE, MARTINELLI G., R. CONTE, A.M.
MARTELLI. (2005). Deguelin, A PI3K/AKT inhibitor, enhances chemosensitivity of leukaemia cells with an active PI3K/AKT pathway.
BRITISH JOURNAL OF HAEMATOLOGY. vol. 129, pp. 677 - 686 ISSN: 0007-1048. [ARTICOLO].
23. RUSSO D, MALAGOLA M, VIVO A, FIACCHINI M, MARTINELLI G., PICCALUGA PP, DAMIANI D, CANDONI A, MICHIELUTTI A,
CASTELLI M, TESTONI N, OTTAVIANI E, RONDONI M, PRICOLO G, MAZZA P, ZUFFA E, ZACCARIA A, RASPADORI D, BOCCHIA M,
LAURIA F, BONINI A, AVANZINI P, GUGLIOTTA L, VISANI G, FANIN R, BACCARANI M. (2005). Multicentre phase III trial on fludarabine,
cytarabine (Ara-C), and idarubicin versus idarubicin, Ara-C and etoposide for induction treatment of younger, newly diagnosed acute myeloid
leukaemia patients. BRITISH JOURNAL OF HAEMATOLOGY. vol. 131(2), pp. 172 - 179 ISSN: 0007-1048. [ARTICOLO].
24. SOVERINI S, MARTINELLI G., ROSTI G, BASSI S, AMABILE M, POERIO A, GIANNINI B, TRABACCHI E, CASTAGNETTI F, TESTONI N,
LUATTI S, DE VIVO A, CILLONI D, IZZO B, FAVA M, ABRUZZESE E, ALBERTI D, PANE F, SAGLIO G, BACCARANI M. (2005). ABL
mutations in late chronic phase chronic myeloid leukemia patients with up-front cytogenetic resistance to imatinib are associated with a greater
likelihood of progression to blast crisis and shorter survival: a study by the GIMEMA Working Party on Chronic Myeloid Leukemia. JOURNAL
OF CLINICAL ONCOLOGY. vol. 23(18), pp. 4100 - 4109 ISSN: 0732-183X. [ARTICOLO].
25. BUONAMICI S, OTTAVIANI E, VISANI G, BONIFAZI F, FIACCHINI M, BACCARANI M., MARTINELLI G. (2004). Patterns of AML1-ETO
transcript expression in patients with acute myeloid leukemia and t(8;21) in complete hematologic remission. HAEMATOLOGICA. vol. 89, pp.
103 - 105 ISSN: 0390-6078. [ARTICOLO].
26. MARTINELLI G., MALAGOLA M, OTTAVIANI E, ROSTI G, TRABACCHI E, BACCARANI M. (2004). Imatinib mesylate can induce complete
molecular remission in FIP1L1-PDGFR-a positive idiopathic hypereosinophilic syndrome. HAEMATOLOGICA. vol. 89, pp. 236 - 237 ISSN:
0390-6078. [ARTICOLO].
27. MARTINELLI G., RONDONI M, BUONAMICI S, OTTAVIANI E, PICCALUGA PP, MALAGOLA M, BACCARANI M. (2004). Molecular
monitoring to identify a threshold of CBFbeta/MYH11 transcript below which continuous complete remission of acute myeloid leukemia inv16 is
likely. HAEMATOLOGICA. vol. 89, pp. 495 - 497 ISSN: 0390-6078. [ARTICOLO].
28. PICCALUGA PP, MARTINELLI G., RONDONI M, MALAGOLA M, GAITANI S, ISIDORI A, BONINI A, GUGLIOTTA L, LUPPI M, MORSELLI
M, SPARAVENTI G, VISANI G, BACCARANI M. (2004). Gemtuzumab ozogamicin for relapsed and refractory acute myeloid leukemia and
myeloid sarcomas. LEUKEMIA & LYMPHOMA. vol. 45(9), pp. 1791 - 1795 ISSN: 1042-8194. [ARTICOLO].
29. TAZZARI PL, CAPPELLINI A, GRAFONE T, MANTOVANI I, RICCI F, BILLI AM, OTTAVIANI E, CONTE R, MARTINELLI G., MARTELLI
AM. (2004). Detection of serine 473 phosphorylated Akt in acute myeloid leukaemia blasts by flow cytometry. BRITISH JOURNAL OF
HAEMATOLOGY. vol. 126(5), pp. 675 - 681 ISSN: 0007-1048. [ARTICOLO].
30. VISANI G, ISIDORI A, GRAFONE T, TOSI P, SANTINI V, MALAGOLA M, MARTINELLI G., PICCALUGA PP, GRAZIEV D, OTTAVIANI E,
SPARAVENTI G, TURA S. (2004). No preferential sensitivity of t(8;21) acute myeloid leukemias to cytosine arabinoside in vitro: is intensity of
therapy or high dose Ara-C crucial for response?. LEUKEMIA & LYMPHOMA. vol. 45(7), pp. 1361 - 1364 ISSN: 1042-8194. [ARTICOLO].
7 - Elenco dei partecipanti all'Unità di Ricerca
7.1 - Componenti
Componenti della sede dell'Unità di Ricerca
nº Cognome
Nome
Università/Ente
Qualifica
Impegno
1°
anno
1.
MARTINELLI
Giovanni
Università degli Studi di BOLOGNA
MUR - BANDO 2007 - * MODELLO B *
Professore Associato non confermato
2°
anno
6
6
-4-
Ministero dell ,Università e della Ricerca
TOTALE
6
6
Componenti di altre Università / Enti vigilati
Nessuno
Titolari di assegni di ricerca
Nessuno
Titolari di borse
nº Cognome
Nome
Università/Ente
Qualifica
Impegno
1°
anno
2°
anno
1.
GIULIANI
Silvia
Università degli Studi di FERRARA
Dottorando
4
4
2.
IACOBUCCI
Ilaria
Università degli Studi di BOLOGNA
Dottorando
4
4
3.
PAOLINI
Stefania
Università degli Studi di BOLOGNA
Specializzando
4
4
4.
PAPAYANNIDIS
Cristina
Università degli Studi di BOLOGNA
Specializzando
4
4
16
16
TOTALE
7.2 - Altro personale
nº Cognome Nome
Dipartimento
Qualifica
AZIENDA OSPEDALIERA
S.ORSOLA-MALPIGHI
2. DE ROSA FRANCESCO AZIENDA OSPEDALIERA
S.ORSOLA-MALPIGHI
3. FALCIANO FRANCESCO CINECA - CONSORZIO
INTERUNIVERSITARIO
UNIVERSITA' DI BOLOGNA
4. FORMICA SERENA
EMATOLOGIA
5. OTTAVIANI
EMATOLOGIA
DIRIGENTE MEDICO I
LIVELLO
LAUREATO
FREQUENTATORE
COLLABORATORE A
PROGETTO
COLLABORATORE A
PROGETTO
DIRIGENTE MEDICO I
LIVELLO
1. ARPINATI
Università/Ente
MARIO
EMANUELA AZIENDA OSPEDALIERA
S.ORSOLA-MALPIGHI
EMATOLOGIA
High Performance
Systems
PEDIATRIA
TOTALE
Impegno Impegno
1° anno 2° anno
4
4
4
4
3
3
4
4
4
4
19
19
7.3 - Personale a contratto da destinare a questo specifico Progetto
Nessuno
7.4 - Dottorati a carico del PRIN da destinare a questo specifico Progetto
Nessuno
8 - Titolo specifico del Progetto svolto dall'Unità di Ricerca
Testo italiano
ATTIVAZIONE COSTITUTIVA DI FOSFATIDILINOSITOLO 3-CHINASI/AKT/MAMMALIAN TARGET OF RAPAMYCIN NELLA LEUCEMIA MIELOIDE ACUTA:
ANALISI GENOMICA E PROFILI DI ESPRESSIONE GENICA IN RISPOSTA ALLA SUA INIBIZIONE IN VIVO ED IN VITRO
Testo inglese
CONSTITUTIVE UPREGULATION OF PHOSPHATIDYLINOSITOL 3-KINASE/AKT/MAMMALIAN TARGET OF RAPAMYCIN SIGNALING IN ACUTE
MYELOGENOUS LEUKEMIA: GENOME ANALYSIS AND GENE EXPRESSION PROFILING IN RESPONSE TO ITS INHIBITION IN VIVO AND IN VITRO
MUR - BANDO 2007 - * MODELLO B *
-5-
Ministero dell ,Università e della Ricerca
9 - Abstract del Progetto svolto dall'Unità di Ricerca
Testo italiano
La leucemia acuta mieloide (LAM) è una malattia emopoietica caratterizzata dall'accumulo di mieloblasti immaturi che non sono capaci di differenziare e sono
resistenti alla morte cellulare. E' principalmente una malattia dell'anziano, con un'età mediana di presentazione di circa 65 anni. Il trattamento della LAM ha subito
notevoli miglioramenti negli ultimi decenni, soprattutto nei pazienti giovani che possono tollerare strategie intensive ed il trapianto di midollo osseo. Invece sono stati
fatti pochi progressi nel trattamento dei pazienti anziani. Questo è dovuto sicuramente alla più alta co-morbidità nella popolazione anziana, ma riflette anche il
differente aspetto biologico della leucemia, manifestandosi questa spesso con caratteristiche ad alto rischio, come la mielodisplasia, presenza di anomalie
citogenetiche e l'espressione dei geni della resistenza ai farmaci. Nonostante siano stati condotti numerosi studi, la patogenesi della LAM ancora non è
completamente compresa e definita. Inoltre, recenti evidenze mostrano e confermano la costitutiva attivazione della chinasi lipidica fosfatidilinositolo 3-chinasi
(PI3K) nella LAM e suggeriscono che essa abbia un ruolo critico nella sopravvivenza dei blasti leucemici. La maggior parte delle funzioni che PI3K svolge, richiede
l'attivazione di Akt (protein kinase B [PKB], successivamente definita come Akt). L'attivazione di Akt potrebbe portare all'attivazione di NF-kappaB (nuclear factorB), CREB, mTOR (mammalian target of rapamycin), e all'inattivazione di GSK-3beta (glycogen synthase kinase-3beta), p53, e fattori di trascrizione FOXO
(Forkhead box class O). Le conseguenze dell'attivazione di questa via di segnalazione sono differenti a seconda del tipo cellulare; comunque numerosi studi
dimostrano che la via di segnalazione mediata da PI3K/Akt regola la sopravvivenza, la proliferazione cellulare, la crescita, la farmacoresistenza ed altri effetti. Tra
gli effettori di Akt, NF B sembra sia costitutivamente attivato nei blasti di LAM e richiesto dalle cellule leucemiche per la sopravvivenza allo stress genotossico. In
generale, rimane ancora poco chiaro il ruolo di tale via di segnalazione nella LAM e se essa possa costituire un effettivo bersaglio terapeutico.
Al fine di identificare bersagli a valle di PI3K/Akt/mTOR nella LAM e di comprendere i meccanismi responsabili della resistenza e sensibilità, studieremo la
potenziale efficacia della combinazione di un anologo nucleosidico, Clofarabina, e di un inibitore di mTOR, Temsirolimus, nella LAM mediante differenti approcci
genetici. La recente tecnologia basata sull'analisi genomica dei polimorfismi a singolo nucleotide (SNP) mediante GeneChip array consente di rilevare con elevata
risoluzione la variazione del contenuto genico a livelli microscopici. Questo approccio ci consentirà di identificare la perdita di eterozigosi (LOH) e di caratterizzare
cambiamenti del contenuto genico nei campioni con LAM prima e dopo il trattamento farmacologico in vivo con Clofarabina e Temsirolimus. Inoltre, condurremo
uno studio dei profili di espressione genica mediante DNA microarrays sia su linee cellulari di LAM geneticamente modificate per ottenere un'attivazione del
signaling di PI3K/Akt/mTOR, che su blasti da pazienti con LAM che presentino un'attivazione costitutiva di PI3K/Akt/mTOR. I risultati di questo studio potrebbero
pertanto portare all'identificazione di nuovi bersagli di questa via di segnalazione che potrebbero poi essere usati per migliorare i trattamenti terapeutici mirati.
Testo inglese
Acute myeloid leukaemia (AML) is a malignant hematopoietic disease characterized by the accumulation of immature myeloblasts which are blocked at various stages
of their differentiation process and resistant to cell death. It is primarily a disease of the elderly, with a median age at presentation of approximately 65 years.
Treatment outcome for AML has somehow improved over the last decade, especially in younger patients who can tolerate intensified treatment strategies including
hematopoietic stem cell transplantation. On the other hand, there has been little progress in the treatment of older patients This is undoubtedly related to increased
co-morbidity in the ageing population, but it also reflects the differing biology of the disease, often presenting in the elderly with a constellation of poor risk features
including associated myelodysplasia, adverse cytogenetic abnormalities and expression of the multidrug resistance phenotype. Despite extensive study, the molecular
pathogenesis of AML is still only partially understood. Several genetic mutations in growth regulatory genes, including Flt3, Ras, and c-kit, have been described.
Several lines of evidence confirm constitutive activation of PI3-kinase (PI3K) in AML and suggest that it plays a critical role in the survival and drug-resistance of
AML blasts. PI3K activates multiple downstream effector pathways. Most of the described actions of PI3K require activation of Akt (protein kinase B [PKB],
subsequently referred to as Akt). Activated Akt may itself lead to activation of NF-kappaB (nuclear factor-kappaB), CREB, mTOR (mammalian target of rapamycin),
and inactivation of GSK-3beta (glycogen synthase kinase-3beta), p53, and FOXO family of transcription factors (Forkhead box class O). The effects of these
pathways are varied in different cell types; however, multiple studies demonstrate that the PI3K/Akt pathway regulates cell survival, proliferation, growth,
metabolism, and other effects, although the exact effects are cell type specific. Of these effectors, NF B is known to be constitutively activated in AML blasts and
required for leukemic cells to survive genotoxic stress. Nevertheless, our knowledge of PI3K/Akt/mTOR downstream targets is very limited. It also remains to be
established whether this pathway could be effectively targeted in vivo. To address these issues, we have chosen to study the potential of the combination of a novel
nucleotide analog, Clofarabine, and a mTOR inhibitor, Temsirolimus, in AML. Recently, the introduction of array-based analysis of single nucleotide polymorphism
(SNP) has allowed for the rapid determination of genome-wide allelic information at high density for a DNA sample. This approach will be used to search for loss of
heterozygosity (LOH) and to characterize DNA copy number change in samples with AML, before and after treatment with Clofarabine/Temsirolimus in vivo.
Moreover, we will perform DNA microarray analysis to obtain gene expression profiles in AML cell lines genetically modified to result in activation of
PI3K/Akt/mTOR signalling, and in AML patient blasts with constitutive activation of PI3K/Akt/mTOR signal transduction pathway. This project could lead to the
identification of novel targets of this signal transduction pathway which could then be used to improve therapeutic strategies targeting this signaling network.
10 - Parole chiave
nº Parola chiave (in italiano) Parola chiave (in inglese)
1. LEUCEMIA ACUTA MIELOIDE ACUTE MYELOID LEUKEMIA
GENE EXPRESSION
2. ESPRESSIONE GENICA
ANALISI
GENOMICA
GENE WIDE ANALYSIS
3.
PI3K/AKT
4. PI3K/AKT
MTOR
MTOR
5.
11 - Stato dell'arte
Testo italiano
La leucemia acuta mieloide (LAM) è un disordine clonale caratterizzato dall'accumulo di progenitori emopoietici tumorali aventi un'alterata capacità di
differenziare. Nonostante siano stati compiuti notevoli progressi nel trattamento dei pazienti con LAM ottenendo alte percentuali di remissioni complete, la maggior
parte dei pazienti perde la risposta e progredisce. La prevenzione della resistenza si basa su programmi di intensificazione che comprendono chemioterapia ad alte
dosi, trapianto autologo e allogenico che possono beneficiare i giovani. Inoltre, le terapie per i pazienti con età superiore ai 60 anni non sono state migliorate da
anni, suggerendo il bisogno di sperimentare farmaci che siano più mirati e meno tossici per il trattamento di questa forma di leucemia(1). Un modello di patogenesi
della LAM sostiene che mutazioni in recettori tirosino-chinasici ed in fattori di trascrizione sono necessarie per causare la leucemia poichè conferiscono un
vantaggio in termini di sopravvivenza e proliferazione (mutazioni di classe I) e alterano la differenziazione cellulare (mutazione di classe II) (2). Mutazioni in FLT3,
c-KIT, e RAS si verificano nel 50%-60% dei casi di LAM (3-7) portando all'attivazione aberrante delle vie di segnalazione che regolano la sopravvivenza e la
proliferazione cellulare come mitogen-activated protein kinase (MAPK), fosfatidilinositolo 3-chinasi (PI3K)/Akt/mTOR, attivatore trascrizionale STAT, o il fattore
nucleare-kappaB (NF-kappaB) (8-10). L'attivazione di queste vie di segnalazione anti-apoptotiche può contribuire alla resistenza agli agenti citotossici che
attualmente si utilizzano nella LAM (11,12). Recentemente, differenti studi hanno mostrato che la via di segnalazione PI3K/Akt/mTOR è attiva nella LAM, sia nelle
cellule blastiche, che nelle popolazioni più immature CD34+, CD38, e CD123 + (13-16). Questa rete di segnalazione è fondamentale per differenti processi
fisiologici, come la progressione del ciclo cellulare, la differenziazione, trascrizione, traduzione, apoptosi e metabolismo. L'attivazione della via PI3K/Akt/mTOR
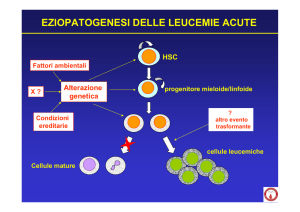
determina la perdita del controllo della proliferazione e dell'apoptosi, provocando la crescita delle cellule tumorali (Fig.1).
Le molecole coinvolte in questa via di trasduzione del segnale possono rappresentare dei potenziali ed importanti bersagli terapeutici nella LAM. mTOR (mammalian
target of rapamycin) è una serina-treonina chinasi coinvolta nella regolazione della crescita e proliferazione cellulare mediante il controllo translazionale della
MUR - BANDO 2007 - * MODELLO B *
-6-
Ministero dell ,Università e della Ricerca
funzione di alcune proteine quali p27kip1 (inibitore delle chinasi ciclina-dipendenti), la proteina del retinoblastoma, la ciclina D1, c-myc, o STAT3. mTOR è attivato
da differenti stimoli come i nutrienti ed i fattori di crescita (17,18). Una volta attivato, mTOR, può fosforilare i suoi effettori a valle, la chinasi ribosomale p70S6
(p70S6K) e la 4E-BP1 (4E-binding protein 1) (19,20). p70S6K attivata, fosforila la proteina ribosomale S6 che inizia la traduzione di un tratto oligopirimidinico
contenente l'mRNA codificante per i componenti del macchinario della sintesi proteica. 4E-BP1 è fosforilata e inattivata da mTOR in risposta ai segnali di crescita.
4E-BP1, dopo che è stata fosforilata, si dissocia dal fattore 4E (eIF-4E), che successivamente si lega al 5' dell'mRNA ed inizia la traduzione dei trascritti che
codificano per proteine coinvolte nel controllo del ciclo cellulare. Questa via di trasduzione del segnale può rappresentare un bersaglio per trattamenti terapeutici
innovativi nella LAM. Questo studio in vitro and in vivo ha l'obiettivo di valutare l'efficacia della combinazione di un analogo nucleosidico, la Clofarabina, e un
inibitore di mTOR, il Temsirolimus, nella LAM. Sono stati descritti differenti meccanismi di resistenza agli inibitori di mTOR, comprese mutazioni in FKBP-12, alto
rapporto eIF-4/4E-BP, alterata regolazione di p27kip1, o l'amplificazione di c-myc (21,22). Tuttavia, è da chiarire se questi meccanismi siano importanti anche nella
LAM. Al fine di identificare possibili cause di resistenza al trattamento con Clofarabina+Temserilimus, eseguiremo un'analisa genomica e di espressione genica su
pazienti prima e dopo il trattamento con questa combinazione di farmaci.
Inoltre, ci proponiamo di identificare geni il cui controllo è, in cellule di LAM, sotto il controllo dell'asse PI3K/Akt/mTOR, mediante tecniche di DNA microarrays.
Studi di questo genere sono stati eseguiti in altri tipi di tumore, dove hanno portato all'identificazione di nuovi bersagli di questa via di segnalazione, ma non sono
mai stati effettuati in cellule di LAM.
Figura 1. Via di segnalazione Akt/mTOR.
Testo inglese
Acute myeloid leukemia (AML) is a clonal disorder characterized by accumulation of malignant hematopoietic progenitor cells with impaired differentiation program.
Despite important progress in the therapy of AML and high rates of complete remission after induction chemotherapy, most patients will relapse and die from the
disease. Prevention of relapse is based on intensified programs, including high-dose chemotherapy and autologous or allogenic transplantion that can benefit young
patients. Thus, outcome of patients older than 60 years has not been improved for decades, underlying the need for targeted and less toxic drugs for the treatment of
this disease(1). A pathogenesis model for AML suggests that mutations of both tyrosine kinase receptors and transcription factors, by conferring survival and/or
proliferative advantage (class I mutation) and by impairing cell differentiation (class II mutation), are needed to cause leukemia (2). Fms-like tyrosine kinase 3
(FLT3), c-KIT, and RAS mutations occur in 50% to 60% of AML cases (3-7) leading to aberrant activation of major cell survival or proliferation pathways such as
mitogen-activated protein kinase (MAPK), phosphatidylinositol 3-kinase (PI3K)/Akt, signal transducer and activator of transcription (STAT), or nuclear
factor-kappaB (NF-kappaB) (8-10). These antiapoptotic signaling pathways also contribute to AML resistance to the cytotoxic agents currently used in this disease
(11,12). Several recent articles have highlighted that the phosphoinositide 3-kinase (PI3K)/Akt/mTOR pathway is activated in AML, both in blast cells and in the
immature CD34 + , CD38Low, and CD123 + population (13-16). This signaling network is crucial to widely divergent physiological processes which include cell
cycle progression, differentiation, transcription, translation, apoptosis, and metabolism. Activation of PI3K/Akt/mTOR signaling results in disrupted proliferation
control and apoptosis, resulting in competitive growth advantage for tumor cells (Fig.1).Molecules integrating multiple signal transduction pathways may represent
relevant therapeutic targets in AML. Mammalian target of rapamycin (mTOR) is a serine/threonine kinase involved in the regulation of cell growth and proliferation
by translational control of key proteins such as the cyclin-dependent kinase (CDK) inhibitor p27kip1, retinoblastoma protein, cyclin D1, c-myc, or STAT 3. mTOR is
activated by different stimuli including nutrients or growth factors (17,18). Once activated, mTOR can phosphorylate its downstream targets, the ribosomal p70S6
kinase (p70S6K) and the 4E-binding protein 1 (4E-BP1) (19,20). Activated p70S6K phosphorylates the 40S ribosomal protein S6 to initiate the translation of 5'
terminal oligopyrimidine tract containing mRNAs encoding components of the protein synthesis machinery. 4E-BP1 is phosphorylated and inactivated by mTOR in
response to a growth signal. Phospho-4E-BP1 dissociates from the eukaryotic initiation factor 4E (eIF-4E), a translation initiation factor that subsequently binds the
cap structure of 5' mRNAs and initiates the translation of transcripts encoding genes involved in cell cycle control. This signal transduction cascade may represent a
target for innovative therapeutic treatments of AML patients. This study aims to evaluating the potential of the combination of a novel nucleoside analog - Clofarabine
- and an mTOR inhibitor - Temsirolimus - in AML. Various mechanisms of resistance to mTOR inhibitors have been described, including FKBP-12 mutations, high
eIF-4/4E-BP1 ratio, defective regulation of p27kip1, or c-myc amplification.21,22 Whether these mechanisms are found in AML remains to be determined. To address
this issue, we will perform high-troughput genome wide analysis and gene expression analysis in blast cells from elderly (>60 years old) AML patients before and
after treatment with Clofarabine and Temsirolimus. Moreover, we will analyze gene expression profiles of AML cell lines and primary blasts displaying activation of
PI3K/Akt/mTOR signaling. This kind of studies has been performed in other types of neoplasia, where it led to the identification of novel genes whose expression is
under the control of this signaling pathway. However, this kind of studies has never been performed in AML cells. This information could then be used for improving
targeted therapies aimed at improving the outcome of AML patient treatment.
MUR - BANDO 2007 - * MODELLO B *
-7-
Ministero dell ,Università e della Ricerca
Figure 1. Akt/mTOR signaling.
12 Riferimenti bibliografici
1.Appelbaum FR, Rowe JM, Radich J, Dick JE. Acute myeloid leukemia. Hematology (Am Soc Hematol Educ Program). Washington, DC: American Society of
Hematology. 2001;62-86.
2.Gilliland DG, Tallman MS. Focus on acute leukemias. Cancer Cell. 2002;1:417-420.
3.Stirewalt DL, Kopecky KJ, Meshinchi S, et al. FLT3, RAS, and TP53 mutations in elderly patients with acute myeloid leukemia. Blood. 2001;97:3589-3595.
4.Radich JP, Kopecky KJ, Willman CL, et al. N-ras mutations in adult de novo acute myelogenous leukemia: prevalence and clinical significance.Blood.
1990;76:801-807.
5.Farr CJ, Saiki RK, Erlich HA, McCormick F, Marshall CJ. Analysis of RAS gene mutations in acute myeloid leukemia by polymerase chain reaction and
oligonucleotide probes. Proc Natl Acad Sci U S A. 1988;85:1629-1633.
6.Care RS, Valk PJ, Goodeve AC, et al. Incidence and prognosis of c-KIT and FLT3 mutations in core binding factor (CBF) acute myeloid leukaemias. Br J
Haematol. 2003;121:775-777.
7.Yokota S, Kiyoi H, Nakao M, et al. Internal tandem duplication of the FLT3 gene is preferentially seen in acute myeloid leukemia and myelodysplastic syndrome
among various hematological malignancies: a study on a large series of patients and cell lines. Leukemia. 1997;11:1605-1609.
8.Hayakawa F, Towatari M, Kiyoi H, et al. Tandemduplicated Flt3 constitutively activates STAT5 and MAP kinase and introduces autonomous cell growth in
IL-3-dependent cell lines. Oncogene. 2000;19:624-631.
9.Guzman ML, Neering SJ, Upchurch D, et al. Nuclear factor-kappaB is constitutively activated in primitive human acute myelogenous leukemia cells. Blood.
2001;98:2301-2307.
10.Birkenkamp KU, Geugien M, Schepers H, Westra J, Lemmink HH, Vellenga E. Constitutive NF-kappaB DNA-binding activity in AML is frequently mediated by a
Ras/PI3-K/PKB-dependent pathway. Leukemia. 2004;18:103-112.
11.O'Gorman DM, Cotter TG. Molecular signals in anti-apoptotic survival pathways. Leukemia. 2001;15:21-34.
12.Guzman ML, Swiderski CF, Howard DS, et al. Preferential induction of apoptosis for primary human leukemic stem cells. Proc Natl Acad Sci U S A.
2002;99:16220-16225.
13.Xu Q, Simpson SE, Scialla TJ, Bagg A, Carroll M. Survival of acute myeloid leukemia cells requires PI3 kinase activation. Blood. 2003;102:972-980.
14.Min YH, Eom JI, Cheong JW, et al. Constitutive phosphorylation of Akt/PKB protein in acute myeloid leukemia: its significance as a prognostic variable.
Leukemia. 2003;17:995-997.
15.Grandage VL, Gale RE, Linch DC, Khwaja A. PI3-kinase/Akt is constitutively active in primary acute myeloid leukaemia cells and regulates survival and
chemoresistance via NF-kappaB, Mapkinase and p53 pathways. Leukemia. 2005;19:586-594.
16.Bardet V, Tamburini J, Ifrah N, et al. Single cell analysis of phosphoinositide 3-kinase/Akt and ERK activation in acute myeloid leukemia by flow cytometry.
Haematologica. 2006;91:757-764.
17.Schmelzle T, Hall MN. TOR, a central controller of cell growth. Cell. 2000;103:253-262.
18.Gingras AC, Raught B, Sonenberg N. Regulation of translation initiation by FRAP/mTOR. Genes Dev. 2001;15:807-826.
19.Reinhard C, Thomas G, Kozma SC. A single gene encodes two isoforms of the p70 S6 kinase: activation upon mitogenic stimulation. Proc Natl Acad Sci U S A.
1992;89:4052-4056.
20.Ming XF, Burgering BM, Wennstrom S, et al. Activation of p70/p85 S6 kinase by a pathway independent of p21ras. Nature.1994;371:426-429.
21.Hosoi H, Dilling MB, Liu LN, et al. Studies on the mechanisms of resistance to rapamycin in human cancer cells. Mol Pharmacol. 1998;54:815-824.
22.Huang S, Bjornsti MA, Houghton PJ. Rapamycins: mechanism of action and cellular resistance.Cancer Biol Ther. 2003;2:222-232.
13 - Descrizione del programma e dei compiti dell'Unità di Ricerca
Testo italiano
Sebbene le cellule di LAM abbiano una attivazione costitutiva di PI3K/Akt/mTOR, che svolge un ruolo chiave nella sopravvivenza di queste cellule, molto poco si
conosce degli effettori di questa via di segnalazione. Gli obiettivi principali dello studio sono quelli di caratterizzare su base molecolare la LAM ed identificare i geni
che possano essere predittivi della risposta o della resistenza agli inibitori di mTOR. Inoltre, ci proponiamo di effettuare uno studio che porti all'identificazione di
MUR - BANDO 2007 - * MODELLO B *
-8-
Ministero dell ,Università e della Ricerca
quei geni che, in cellule di LAM, sono sotto il controllo dell'asse PI3K/Akt/mTOR.
Le fasi principali del progetto comprendono:
a) la raccolta e la conservazione del materiale biologico dopo l'ottenimento del consenso informato del paziente, prima del trattamento e ad intervalli di tempo
regolari durante il trattamento da campioni di midollo osseo e/o sangue periferico;
b) l'identificazione dei differenti sottotipi di LAM mediante studi molecolari;
c) analisi genomica ad alta risoluzione;
d) analisi di espressione genica;
e) validazione dei dati di microarray mediante la tecnica di Real-Time RT-PCR quantitativa ad alta produttività (MicroFluidic Cards, Applera).
a) Raccolta e conservazione del materiale biologico per studi molecolari
In seguito all'ottenimento del consenso informato dei pazienti arruolati nei vari studi clinici, l'Unità di Ricerca di Bologna-Martinelli creerà una banca di:
1) campioni biologici, processati e conservati prima dell'inizio del trattamento e successivamente ad intervalli di tempo regolari da campioni di midollo osseo e/o
sangue periferico da pazienti con LAM; il materiale biologico comprende: cellule mononucleate, cellule CD34+, lisati proteici, RNA e DNA.
2) dati clinici, raccolti in modo accurato in formato elettronico prima del trattamento e successivamente ad intervalli di tempo regolari, che consentiranno di fare
correlazioni tra i risultati biologici ottenuti e l'andamento clinico.
b) Studi di biologia molecolare
La LAM è considerata il risultato della cooperazione di differenti tipi di mutazioni. Una categoria di alterazioni comprende le mutazioni che interferiscono con la
trascrizione e portano ad un arresto della differenziazione. Questo processo può essere mediato dalla diretta alterazione dei fattori di trascrizione dovuta alla fusione
di due geni, come le leucemie coinvolgenti il core-binding-factor (CBF) o quelle esprimenti il gene di fusione PML-RARalfa, o dalla diretta interferenza con la
trascrizione, come nel caso dei riarrangiamenti coinvolgenti il gene MLL. Un'altra importante categoria è costituita dalle mutazioni attivanti che determinano
aumentata proliferazione cellulare e alla stimolazione di tirosino-chinasi.
1) Per identificare i differenti sottotipi di AML saranno eseguite reazioni di RT-PCR di specifici geni di fusione, come AML1-ETO, CBFalfa-MYH11.
2) Inoltre, verranno amplificati i geni FLT3 (WT, ITD, TKD), NPM e C-KIT mediante RT-PCR seguita da digestione mediante enzima di restrizione (Eco-RV for
FLT3, HINF1 for c-KIT) e/o DHPLC e/o sequenziamento automatico secondo le procedure già descritte in letteratura.
3) Analisi mutazionale di p53. P53 è un gene appartenente alla classe dei soppressori tumorali. Le mutazioni con perdita di funzione in questi geni sono oncogeniche.
L'inattivazione del gene p53 mediante mutazioni puntiformi, delezioni o riarrangiamenti è stata descritta in molti tumori umani, ma nelle neoplasie ematologiche
sembra sia un evento raro. Più del 90% dei pazienti con LAM hanno p53 wild-type, ma se p53 è mutato o deleto la malattia si associa spesso alla resistenza alla
chemioterapia e ad una più bassa percentuale di remissioni. Lo studio del network di p53 rappresenta quindi un' ottima strategia per identificare nuovi bersagli
terapeutici. Lo stato molecolare di p53 sarà analizzato nei blasti di pazienti con leucemia mieloide acuta prima e dopo trattamento mediante RT-PCR seguita da
DHPLC e/o sequenziamento.
c) Analisi genomica dei campioni con LAM
Molti pazienti con LAM hanno un cariotipo normale o presentano anomalie cromosomiche non ricorrenti ed in questi casi i meccanismi della patogenesi non sono
chiari. L'analisi genomica dei polimorfismi a singolo nucleotide (SNP) mediante GeneChip array, consente di rilevare con elevata risoluzione la variazione del
contenuto genico a livelli microscopici e la perdita di eterozigosi (LOH) dovuta alla monosomia e alla disomia uniparentale (UPD). Al fine di identificare a livello
microscopico lesioni genetiche responsabili del processo di leucemogenesi e della resistenza al trattamento con Clofarabina+Temsirolimus, effettueremo un'ampia
analisi genomica utilizzando il GeneChip® Mapping 500K array set (Nsp and Sty Assays) che è capace di genotipizzare in una sola volta e in tutto l'intero genoma ad
alta risoluzione circa 500.000 SNPs. Il materiale biologico di partenza è costituito da DNA genomico estratto da pazienti con LAM prima e dopo del trattamento. Il
DNA deve essere integro. Il DNA sarà estratto dai campioni leucemici e processato seguendo in dettaglio i protocolli forniti dalla azienda. L'analisi verrà effettuata
utilizzando i seguenti programmi: Affymetrix GeneChip Operating Software v 3.0 e Affymetrix GeneChip Genotyping Analysis Software (GTYPE) v 4.0. Sarà possibile
identificare alterazioni geniche nel numero di copie mediante confronto con il genoma di un set di 48 individui normali e un set di campioni ottenuti da pazienti con
LAM in remissione. Le regioni di amplificazione e delezione saranno visualizzate utilizzando il programma Integrated Genome Browser e mappate su RefSeq per
identificare in dettaglio i geni specifici coinvolti nella lesione. Le alterazioni ritenute importanti per la patogenesi e lo sviluppo della resistenza verranno confermate
mediante FISH, mediante la tecnica di Real-Time RT-PCR quantitativa ad alta produttività (GeneCards, Applera) e sequenziamento.
d) Analisi dei profili di espressione genica
Recentemente, l'analisi del profilo di espressione genica nella LAM ha consentito di distinguere differenti sottoclassi di LAM e di predire l'esito clinico,
rappresentando una tecnica promettente per meglio anticipare l'evoluzione della malattia. La precoce identificazione dell'espressione genica in risposta alla terapia
potrebbe rappresentare un nuovo metodo per monitorare lo stato e la fase della malattia. L'immediata risposta genica potrebbe indicare se il farmaco di scelta è
efficace nell'eradicare le cellule leucemiche o se è opportuno cambiare la terapia. Inoltre, la precoce analisi di alterazioni nell'espressione genica potrebbe facilitare
l'identificazione di nuovi bersagli terapeutici, consentendo la migliore terapia per il paziente. Al fine di identificare vie di segnalazione molecolari che possono essere
alterate in seguito al trattamento con Clofarabina+Temsirolimus, eseguiremo una analisi mediante DNA microarray sui blasti di pazienti con LAM prima e dopo il
trattamento (campioni forniti da Unità Roma-Amadori). La stessa analisi verrà condotta su linee cellulari di LAM (HL60 e sulle MOLM, etc., fornite da Unità
Bologna-Martelli) con attivazione della via di segnalazione PI3K/Akt/mTOR, che su blasti LAM raccolti sia a Bologna (Unità Martinelli) che a Torino (Unità Cilloni)
prima e dopo trattamento con l'inibitore del pathway, LY294002. Questo approccio ci consentirà di determinare i meccanismi responsabili della potenziale efficacia
del trattamento combinato di Clofarabina+Temsirolimus nelle cellule dei pazienti soggetti al trial clinico. Inoltre, l'analisi del profilo di espressione genica ci
consentirà di identificare un profilo genomico predittivo che possa essere associato alla sensibilità o resistenza al trattamento. L'RNA totale sarà estratto usando il
kit Qiagen RNeasy (Qiagen, Valencia, CA, USA) seguendo il protocollo del produttore. Mediante dosaggio allo spettrofotometro ND-1000 (NanoDrop Technologies,
Wilmington, DE, USA) verrà determinata la concentrazione dell'RNA estratto, mentre la sua integrità verrà saggiata caricandone 1 microg su un gel di agarosio al
2%. Le reazioni di biotinilazione saranno eseguite usando i protocolli standard forniti dal produttore (Affymetrix). I campioni saranno ibridizzati sul chip
dell'Affymetrix HG133 2.0 Plus per 16 ore, lavati e processati seguendo il protocollo standard Eukaryotic GE WS2v4 ed utilizzando un'amplificazione del segnale
mediata da anticorpo. Le immagini dai chips scannerizzati saranno analizzate utilizzando il programma Affymetrix Microarray Analysis Suite 5.0 (MAS 5.0). La
quantità di un determinato trascritto, così come la sua assenza o presenza, saranno determinate utilizzando l'algoritmo MAS 5.0 per l'analisi. Sarà possibile
identificare geni che sono espressi in modo differenziale e i pazienti verranno suddivisi in gruppi utilizzando il software GeneSpring 7.3 (Silicon Genetics, Redwood
City, CA). L'utilizzo dei programmi EASE e Ingenuity® consentirà di identificare processi biologici, funzioni molecolari e componenti cellulari dei geni analizzati.
Questa parte del progetto sarà svolta in collaborazione con il Laboratorio di Ematologia ed Oncologia Pediatrica "Lalla Seràgnoli" - Policlinico S.Orsola/Malpighi
(www.pharmagen.it) e con il CINECA (www.cineca.it). Questa collaborazione consentirà di creare una piattaforma informatizzata ed una importante banca dati in
cui verranno raccolti tutti i risultati ottenuti dall'analisi di espressione genica e dall'analisi genomica mediante SNP array e a cui sarà possibile accedere in remoto.
e) Validazione quantitativa dei geni selezionati attraverso la tecnica di RealTime PCR quantitativa ad alta produttività (GeneCards, Applera)
La validazione dei risultati ottenuti dall'analisi di espressione genica e di genotipizzazione sarà effettuata mediante la tecnica di RealTime PCR quantitativa ad alta
produttività. L'utilizzo dei TaqMan Low-Density Arrays consente di valutare contemporaneamente l'espressione di un massimo di 384 geni in un singolo esperimento.
A tal fine, 1 microg di RNA totale sarà retrotrascritto in cDNA usando dei random esameri. Le reazioni di PCR quantitativa saranno eseguite sullo strumento ABI
PRISM 7900 Sequence Detector utilizzando reagenti, protocolli e softwares specifici (Applied Biosystems, Foster City, CA). In particolare analizzeremo l'espressione
di geni ABC, tirosino-chinasi e geni coinvolti in vie di segnalazione del segnale di differenziazione.
f) Inoltre questa Unità sarà responsabile di fornire all'Unità Bologna-Martelli campioni di LAM all'esordio o da pazienti ricaduti refrattari che serviranno per gli
studi traslazionali in vitro sul trattamento combinato Clofarabina+Temsirolimus.Fornirà anche campioni all'Unità Torino-Cilloni per l'analisi
proteomica-fosfoproetomica.
L'intero progetto verrà completato in 24 mesi, secondo l'organizzazione in fasi e le tempistiche specificate nella figura seguente:
MUR - BANDO 2007 - * MODELLO B *
-9-
Ministero dell ,Università e della Ricerca
Testo inglese
Description of the project
AML cells have constitutive activation of PI3K/Akt/mOTR signaling which is important for their survival. However, the downstream effectors of this signalling
neywork in leukemic cells are poorly understood. The main aim of this study is to better characterize the molecular signature of acute AML and to identify genes
which may predict sensitivity or resistance to mTOR inhibitors. Moreover, we aim to analyzing gene expression profiles in AML cell lines and AML blasts with
upregulated PI3K/Akt/mTOR signalling.
To this purpose, we will:
a) collect and store biological material after written informed consent of the patients, at baseline and at regular time points during treatment from bone marrow
and/or peripheral blood samples;
b) perform molecular studies to identify the subtype of AML;
c) perform high-resolution genome wide analysis;
d) perform gene expression analysis;
e) validate microarray results by high-through-put Real-Time RT-PCR (MicroFluidic cards of Applied Biosystems).
a) Collection and storage of biological material for molecular studies
Upon written informed consent of the patients enrolled in the trial, the Research Unit will create a bank of:
1) biological samples, processed and stored at baseline and at regular time points during treatment from bone marrow and/or peripheral blood samples from patients
with AML, as well as from patients in relapse; material will include mononuclear cells, leukemic CD34+ cells, protein lysates, RNA, DNA.
2) clinical data, accurately recorded in an electronic format at baseline and at regular timepoints during treatment and available for correlations between biological
findings and clinical outcome.
b) Molecular biology study
AML is considered to result from the cooperation of different types of mutations. One category is represented by mutations that interfere with transcription and lead
to a stop in differentiation. This can be mediated by direct alteration of transcription factors due to gene fusions, such as in the core-binding-factor (CBF)-leukemias
or in PML-RARalpha positive AML, or by indirect interference with transcription, such as in case of the MLL-rearrangements. A further main category is represented
by activating mutations which lead to increased cell proliferation such as by stimulation of tyrosine kinases.
1) RT-PCR of specific fusion proteins, such as AML1-ETO, CBFalpha-MYH11, will be performed in order to identify the AML subtype.
2) Molecular status of FLT3 (WT, ITD, TKD), NPM and C-KIT will be performed by RT-PCR followed by restriction enzyme digestion (Eco-RV for FLT3, HINF1 for
c-KIT) and/or DHPLC and/or sequencing analysis as reported in standard procedures.
3) Mutational analysis of p53 will be performed. P53 belongs to the tumor suppressor class of genes whose loss-of-function mutations are oncogenic. Inactivation of
the p53 gene by point mutation, deletion, or rearrangement have been found in a wide range of human tumors, but in hematological malignancies this is a rare
feature. In AML more than 90% of the patients express wild type p53 in their cancer cells, but if p53 is mutated or deleted the disease is often found to be
chemoresistant and to have lower complete remission rates. The p53 network is therefore a good candidate in the search of new targets for treatment. Molecular
status of p53 will be performed on blast cells from acute myeloid leukemia patients before and after treatment by RT-PCR followed by DHPLC and/or sequencing
analysis.
c) Gene wide analysis of AML samples
A large proportion of AML has either a normal karyotype or nonrecurrent chromosomal abnormalities and the underlying pathogenesis remains obscure. The
MUR - BANDO 2007 - * MODELLO B *
- 10 -
Ministero dell ,Università e della Ricerca
introduction of array-based analysis of single nucleotide polymorphism (SNP) allows for the identification of copy number changes at the sub-megabase level and the
identification of loss of heterozygosity (LOH) due to monosomy and uniparental disomy (UPD). In order to identify, at submicroscopic level, genetic lesions driving
leukemogenesis and resistance to Clofarabine+Temsirolimus therapy, we will perform a genome wide analysis using the GeneChip® Mapping 500K array set (Nsp
and Sty Assays) which is able to genotyping at once across the whole genome and at high resolution about 500,000 SNPs. The starting biological material is
represented by genomic DNA extracted from mononuclear cells of AML patients before beginning of the treatment and after treatment with
Clofarabine+Temsirolimus (samples provided by Dr. Amadori-Rome). DNA must be: double-stranded, free of PCR inhibitors, not contaminated with other human
genomic DNA sources, or with genomic DNA from other organism and not degraded. DNA is extracted from leukaemia samples and processed according to the
manufacturer's instructions. CEL files are generated using either Affymetrix GeneChip Operating Software v 3.0 or Affymetrix GeneChip Genotyping Analysis
Software (GTYPE) v 4.0. Raw signal data are analyzed by BRLMM algorithm and copy number state is calculated with respect to a set of 48 Hapmap normal
individuals and a diploid reference set of samples obtained from AML cases in remission. Regions of amplification and deletion are visualized by Integrated Genome
Browser and mapped to RefSeq to identify the specific genes involved in the lesion. Copy number variations (CNVs) will be confirmed by FISH, high-performance
reverse transcription-quantitative polymerase chain reaction (RQ-PCR) and nucleotide sequencing.
d) Gene expression profiling analysis
Gene expression profiling in AML has, in recent years, proven able to distinguish AML subclasses and predict clinical outcome and is, as such, a promising technique
for improved disease evaluation. The early detection of gene expression in response to therapy may be a novel way of monitoring disease management. The immediate
gene response may be an indication of whether the drug of choice is efficient in leukemic cell eradication and may early indicate the need for other therapeutic
measures. Furthermore, these early alterations in gene expression could facilitate identification of new treatment targets, thereby enabling better patient care and
follow-up in the future. In order to identify molecular pathways that may be down/up-regulated by exposure to Clofarabine +Temsirolimus, we will perform a DNA
microarray analysis on blast cells from AML patients before and after treatment (from Dr. Amadori-Rome). We will also perform a gene expression analysis on HL60
cell line and MOLM cell lines (from Dr. Martelli-Bologna), which have a constitutive activation of PI3K/Akt/mTOR pathway, as well as in AML blasts (recruited in
Bologna and Turin) before and after treatment with LY294002 inhibitor. This approach will allow us to elucidate the mechanisms responsible of the potential efficacy
of combination of Clofarabine+Temsirolimus in AML cells. We will also use this gene expression profiling strategy to identify a predictive genomic profile that may
be associated with sensitivity or resistance to treatment. In addition, we will identify genes which are under the control of the PI3K/Akt/mTOR network in AML cells,
an issue which has not been addressed before. Total RNA will be isolated from blast cells of each patient before treatment using RNeasy RNA Isolation Mini Kit
(Qiagen, Valencia, CA, USA), following the manufacturer's recommendations. The concentration of extracted RNA will be measured using a ND-1000
spectrophotometer (NanoDrop Technologies, Wilmington, DE, USA) and its integrity will be checked by loading 1 microg on an ethidium bromide-stained 2%
agarose gel. Biotin-labeled target synthesis reactions will be performed using standard protocols supplied by the manufacturer (Affymetrix). Briefly, 100 ng of the
RNA will be converted into double-stranded cDNA by reverse transcription using the cDNA synthesis kit, following the protocol supplied by the manufacturer, with a
T7-(dT)24 primer (Affymetrix). After the second-strand synthesis, cRNA will be generated from the purified cDNA sample (Gene Chip Sample Cleanup Module;
Affymetrix) by an in vitro transcription reaction (MEGAscript® T7 Kit, Ambion). The cRNA will be then purified (Gene Chip Sample Cleanup Module; Affymetrix)
and a second cycle of "retrotransciption, second-strand synthesis and sample cleanup" will be performed, followed by a final Biotin-Labeled cRNA synthesis
(GeneChip IVT Labeling Kit, Affymetrix). The labeled cRNA will be purified using the Affymetrix spin columns and the concentration of biotin-labeled cRNA will be
determined by Nanodrop, while the quality check will be performed by means of the Bioanalyzer. 5 microg of each biotinylated cRNA preparation will be fragmented
and put in the hybridization cocktail. Samples will be hybridized to Affymetrix HG133 2.0 Plus Gene Chip Arrays for 16 hours. Gene chips will be than washed and
stained following the instrument's standard Eukaryotic GE WS2v4 protocol and using antibody-mediated signal amplification. The images from the scanned chips will
be processed by means of Affymetrix Microarray Analysis Suite 5.0 (MAS 5.0). The amount of a transcript mRNA will be determined with the MAS 5.0 absolute
analysis algorithm, as well as the presence or the absence of a transcript. The identification of differentially expressed genes and the patients clustering will be
performed with GeneSpring 7.3 software (Silicon Genetics, Redwood City, CA). The identification of biologic processes, molecular functions and cellular components
of genes will be assessed by EASE and Ingenuity® softwares. This part of the project will be performed in collaboration with the Laboratory of Hematology and
Pediatric Oncology "Lalla Seràgnoli" - Policlinico S.Orsola/Malpighi (www.pharmagen.it)and with the CINECA (www.cineca.it). This collaboration will allow us to
create a computerized platform and an important data bank to storage the results from gene expression and SNP array analysis. Furthermore, we will have a remote
access service.
e) High-performance quantitative Real-Time RT-PCR (MicroFluidic cards)
Microarray data will be further validated by high-through-put Real-Time RT-PCR (MicroFluidic cards of Applied Biosystems). With the TaqMan Low-Density
Arrays, it is possible to simultaneously assay the RNA expression levels of up to 384 genes on a single card. 1 microg of total RNA will be reverse transcribed into
total cDNA using random examer. Real-time RT-PCR will be performed on an ABI PRISM 7900 Sequence Detector using dedicated reagents, protocols and softwares
(Applied Biosystems, Foster City, CA). Genes which will be investigated include: ABC genes, tyrosine kinase and genes involved in different pathways.
f) This Unit will also collect AML samples (either untreated or relapsed/refractory) which will be then employed by the Unit of Dr. Martelli-Bologna, for the
translational studies about the in vitro efficacy of Clofarabine+Temsirolimus treatment. Moreover, this Unit will ship AML samples to the Unit of Dr. Cilloni-Turin
for proteome/phosphoproteome analysis.
The whole project will be performed in two years according to the phases and the timing specified in the scheme below:
MUR - BANDO 2007 - * MODELLO B *
- 11 -
Ministero dell ,Università e della Ricerca
14 - Descrizione delle attrezzature già disponibili ed utilizzabili per la ricerca proposta
Testo italiano
nº anno di acquisizione
2007
1.
2007
2.
3.
4.
5.
Descrizione
Sistema completo di prefrazionamento bidimensionale di proteine Proteomelab PF2D (Beckman)
2006
LightCycler 480 - Real Time PCR System è una piattaforma per la quantificazione o genotipizzazione in tempo
reale PCR (Roche)
Piattaforma AFFYMETRIX per l'analisi dell'espressione genica e dei polimorfismi a singolo nucleotide (SNP)
2006
ABI PRISM 7900 Sequence Detector (Applied Biosystems)
2004
Sequenziatore automatico ABI PRISM 310 (Applied Biosystems)
Testo inglese
nº anno di acquisizione
2007
1.
2007
2.
3.
4.
5.
Descrizione
Complete system of bidimensional predivision of proteins Proteomelab PF2D (Beckman)
2006
LightCycler 480 Real-Time PCR System is a high throughput gene quantification or genotyping real-time PCR
platform (Roche)
AFFYMETRIX platform for gene expression profiling and single nucleotide polymorphism analysis (SNPs)
2006
ABI PRISM 7900 Sequence Detector (Applied Biosystems)
2004
ABI PRISM 310 Automatic sequencer (Applied Biosystems)
MUR - BANDO 2007 - * MODELLO B *
- 12 -
Ministero dell ,Università e della Ricerca
15 - Descrizione delle Grandi attrezzature da acquisire (GA)
Testo italiano
Nessuna
Testo inglese
Nessuna
16 - Mesi persona complessivi dedicati al Progetto
Numero
Componenti della sede dell'Unità di Ricerca
Componenti di altre Università/Enti vigilati
Titolari di assegni di ricerca
Titolari di borse
Dottorato
Post-dottorato
Scuola di Specializzazione
Personale a contratto
Assegnisti
Borsisti
Altre tipologie
Dottorati a carico del PRIN da destinare a questo specifico progetto
Altro personale
TOTALE
Impegno
1° anno
1
0
0
2
0
2
0
0
0
0
5
10
Impegno
2° anno
Totale
mesi
persona
6
6
12
8
8
16
8
8
16
0
19
0
19
0
38
41
41
82
17 - Costo complessivo del Progetto dell'Unità articolato per voci
Voce di spesa Spesa
in
Euro
Materiale
inventariabile
0
Grandi
Attrezzature
65.500
Materiale di
consumo e
funzionamento
Spese per
calcolo ed
elaborazione
dati
Personale a
contratto
Dottorati a
carico del PRIN
da destinare a
questo specifico
progetto
Servizi esterni
Missioni
Pubblicazioni
Descrizione dettagliata
(in italiano)
57.500 Euro (materiale plastico monouso, kit per separazione
CD34, kit per estrazione RNA (Rneasy Mini Kit, Qiagen) e DNA
(Qiamp DNA Blood Mini Kit, Qiagen), kit per retrotrascrizione ,
kit per amplificazione, kit di purificazione (PCR Purification Kit
QIAquick, Qiagen) e di sequenziamento (Dye-Ex, 2.0 Spin kit,
Qiagen) filtri, colonna e soluzioni per D-HPLC, Affymetrix HG133
2.0 Plus Gene Chip Arrays, GeneChip® Human Mapping 250K
Nsp Array, GeneChip® Human Mapping 250K Nsp Assay Kit,
Ingenuity Pathways Analysis 5.5, Ingenuity Sistems) + 10% "oneri
di gestione" pari a 8.000 Euro
10.000 Raccolta, archiviazione ed elaborazione dati studi clinici ed
acquisizione software PARTEK e INGENUITY
Descrizione dettagliata
(in inglese)
57.500 Eur (disposable plasticware, kits for CD34 cell selection,
RNA (Rneasy Mini Kit, Qiagen) and DNA (Qiamp DNA Blood
Mini Kit, Qiagen) extraction kits, reverse transcription kits,
amplification kits, purification (PCR Purification Kit QIAquick,
Qiagen) and sequencing kits (Dye-Ex, 2.0 Spin kit, Qiagen),
solutions, filters and column for D-HPLC, Affymetrix HG133 2.0
Plus Gene Chip Arrays, GeneChip® Human Mapping 250K Nsp
Array, GeneChip® Human Mapping 250K Nsp Assay Kit,
Ingenuity Pathways Analysis 5.5, Ingenuity Sistems) + 10%
(8.000 Eur) handling expenses
Recording and handling of data from clinical trial, use of
PARTEK and INGENUITY softwares.
0
0
500 Partecipazione a incontri per
garantire la cooperazione e il coordinamento del lavoro
2.000 Pubblicazioni scientifiche e abstracts inerenti i risultati della
ricerca
MUR - BANDO 2007 - * MODELLO B *
Participation to meetings
in order to assure cooperation and
coordination about the present project
Scientific publications and abstracts reporting the results of the
present research
- 13 -
Ministero dell ,Università e della Ricerca
Partecipazione / 2.000
Organizzazione
convegni
Altro
TOTALE
80.000
Iscrizione a convegni nazionali ed internazionali per la
divulgazione dei
risultati ottenuti
Registration fees for attending national and
international meetings and presenting the results of the research
18 - Prospetto finanziario dell'Unità di Ricerca
Voce di spesa
a.1) finanziamenti diretti, disponibili da parte di
Università/Enti vigilati di appartenenza dei ricercatori
dell'unità operativa
a.2) finanziamenti diretti acquisibili con certezza da parte di
Università/Enti vigilati di appartenenza dei ricercatori
dell'unità operativa
b.1) finanziamenti diretti disponibili messi a disposizione da
parte di soggetti esterni
b.2) finanziamenti diretti acquisibili con certezza, messi a
disposizione da parte di soggetti esterni
c) cofinanziamento richiesto al MUR
Totale
Importo in Euro
24.000
56.000
80.000
19 - Certifico la dichiarata disponibilità e l'utilizzabilità dei finanziamenti a.1) a.2) b.1) b.2)
SI
Firma _____________________________________
(per la copia da depositare presso l'Ateneo e per l'assenso alla diffusione via Internet delle informazioni riguardanti i programmi
finanziati e la loro elaborazione necessaria alle valutazioni; D. Lgs, 196 del 30.6.2003 sulla "Tutela dei dati personali")
Firma _____________________________________
MUR - BANDO 2007 - * MODELLO B *
Data 23/10/2007 ore 17:36
- 14 -
Ministero dell ,Università e della Ricerca
ALLEGATO
Curricula scientifici dei componenti il gruppo di ricerca
Testo italiano
1.
GIULIANI Silvia
Curriculum:
Silvia Giuliani
Nata a Pescara il 08/08/1975
Residente in Via Yuri Gagarin 23, 40100 Bologna
Email: [email protected]
Nazionalità italiana
Percorso di studi:
Dottoranda al 3° anno in Biochimica, Biologia Molecolare e Biotecnologie presso l' Università degli studi di Ferrara
Master in Bioinformatica presso l' Università degli studi Bicocca di Milano
Laurea in Chimica e Tecnologie Farmaceutiche presso l' Università degli studi di Chieti
Diploma classico presso il liceo classico "G. D'Annunzio" di Pescara
Esperienze professionali:
CINECA dal 2004 fino ad oggi. Sono impegnata nella realizzazione di una infrastruttura informatica per la gestione e l' analisi dei dati di microarray.
Relativamente alla mia attività di dottorato ancora in corso sono coinvolta in uno studio di dinamica molecolare riguardante il fattore di trascrizione NFkB.
Tirocinio in Biochimica presso l' Università degli studi di Chieti. Mi sono occupata delle studio conformazionale delle proteine prioniche, oggetto della mia
tesi di laurea "La mutazione patologica D202N destabilizza la conformazione dell' elica 3 della proteina prionica".
Lingue straniere e conoscenze informatiche:
Inglese discreto, francese discreto
Sistemi operativi: windows, linux
Linguaggi di programmazione: perl, java, mysql
pubblicazioni non disponibili
2.
IACOBUCCI Ilaria
Curriculum:
DATI PERSONALI
Nome e cognome:Ilaria Iacobucci
Data di nascita:18/09/1980.Termoli, Campobasso.
Nazionalità: Italiana
Indirizzo:Via Renato Simoni, 4 Bologna 40127
Italia
Telefoni:0039-051 6363791
0039-348 7622848
Fax:0039-051 6364037
E-mail: [email protected]
ISTRUZIONE
°09/1994-06/1998Liceo Scientifico Statale "A.. Romita", Campobasso.
Conseguimento del diploma con voto finale 100/100.
°09/1999-07/2004Università di Bologna
Laureta con lode in Biotecnologie Mediche (Facoltà di Medicina) il 9/07/2004.
Titolo della tesi: "La quantificazione del trascritto Bcr-Abl è utile per predire la risposta all'imatinib nella Leucemia mieloide Cronica".
Tesi sperimentale presso:
Istituto di Ematologia ed Oncologia Medica "L. e A. Seràgnoli",
Policlinico "S Orsola", Bologna
Prof. Michele Baccarani
°01/2005-in corso
Università di Bologna
Dottorato in Ematologia Clinica e Sperimentale
°08/2005-02/2006
III Medizinische Klinik Hamatologie/Oncologie
Universitat Heidelberg, Mannheim, Germany
Prof. Dr. Andreas Hochhaus
Svolgimento del progetto Marco Polo "standardization of BCR-ABL quantification and analysis of BCR-ABL mutations in patients treated with tyrosine
kinase inhibitors".
CONOSCENZE E COMPETENZE PROFESSIONALI
Tecniche di biologia molecolare finalizzate alla diagnostica ed alla ricerca oncoematologica:
-separazione di cellule mononucleate da midollo osseo o sangue periferico mediante gradiente di densità;
-estrazione di RNA da cellule conservate in guanidinio-isotiocianato (GITC) e sangue intero;
-estrazione di DNA da GITC, sangue intero, tessuti criopreservati e inclusi in paraffina;
-dosaggio spettrofotometrico di proteine ed acidi nucleici;
-PCR, RT-PCR;
-PCR quantitativa (tecnologia TaqMan);
-clonaggio di sequenze di DNA in vettori plasmidici;
-sequenziamento automatico;
culture cellulari
esperimenti di microarry.
MUR - BANDO 2007 - * MODELLO B *
- 15 -
Ministero dell ,Università e della Ricerca
LINGUE STRANIERE
° Inglese
Livello Europeo:Ascolto B2
Lettura B2
Scritto B2
Parlato B2
CONOSCENZE INFORMATICHE
-Ottima conoscenza dei softwares del pacchetto Office: Word, Power Point, Access, Excel.
-Ottima conoscenza di programmi di design grafico (GraphPad, PhotoShop).
-Ottima conoscenza di Endnote.
-Uso di database on line (GeneBank, SwissProt, PDB).
-Uso dei softwares Chromas e Olig.
-Uso dei softwares Affymetrix.
-Allineamento di sequenze nucleotidiche mediante BLAST/FASTA.
ATTIVITA' DIDATTICA
-"Modalità di prelievi" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", Aprile 10-12 2006).
-"Manipolazione del campione" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", Aprile 10-12 2006).
-"Estrazione dell'acido nucleico e retrotrascrizione" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", April 10-12 2006).
-"Report dei risultati" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", Aprile 10-12 2006).
ATTIVITA' SCIENTIFICA, PARTECIPAZIONE A CONGRESSI E SEMINARI
1.Adult Stem Cells. A Challenge for the future, Bologna, March 21-23, 2004
2.46th Annual Meeting of the American Society of Hematology (ASH). San Diego, California December 4-7, 2004.
3.Applicazioni in Medicina Molecolare, Bologna, April 4, 2005.
4.GIMEMA CML Working Party, Bologna-Jolly Hotel De La Gare, April 18, 2005
5.10th Congress of the European Hematology Association Stockholm, Sweden. June 2-5, 2005
6.40° Congresso Nazionale SIE, Bergamo, July 3-6, 2005.
7.New drugs in Hematology, Bologna, October 9-11, 2005.
8.Third ESH International Conference on chronic Myeloid leukaemia in the 21st century. Genoa, Novenber 11-13, 2005-Genoa.
9.47th Annual Meeting of the American Society of Hematology (ASH). December 10-13, 2005 Atlanta, GA.
10.10th Congress of the European Hematology Association Amsterdam. 15 - 18 June, 2006.
11.IX Congresso Nazionale SIES, Napoli, September 20-22, 2006.
12.Dasatinib CA180-043 Investigator Meeting, Rome October 16, 2006.
13.7900 HT Real Time PCR System, Bologna September 18-19, 2006.
14.48th Annual Meeting of the American Society of Hematology (ASH). December 9-12, 2006. Orlando, Florida.
15.AACR, American Association for Cancer Research Annual Meeting 2007. April 14-18, 2007. Los Angeles, California.
16.12th Congress of the European Hematology Association. Vienna, Austria, June 7-10, 2007.
Pubblicazioni:
BACCARANI M, CILLONI D, RONDONI M, OTTAVIANI E, MESSA F, MERANTE S, TIRIBELLI M, BUCCISANO F, TESTONI N, GOTTARDI E, DE
VIVO A, GIUGLIANO E, IACOBUCCI I., PAOLINI S, SOVERINI S, ROSTI G, RANCATI F, ASTOLFI C, PANE F, SAGLIO G, MARTINELLI G. (2007). The
efficacy of imatinib mesylate in patients with FIP1L1-PDGFRalpha-positive hypereosinophilic syndrome. Results of a multicenter prospective study.
HAEMATOLOGICA. vol. 92(9), pp. 1173 - 1179 ISSN: 0390-6078. [ARTICOLO].
BUSSOLARI R, CANDINI O, COLOMER D, CORRADINI F, GUERZONI C, MARIANI SA, CATTELANI S, SILVESTRI C, PECORARI L, IACOBUCCI I.,
SOVERINI S, FASANO T, MARTINELLI G, CERVANTES F, CALABRETTA B. (2007). Coding sequence and intron-exon junctions of the c-myb gene are
intact in the chronic phase and blast crisis stages of chronic myeloid leukemia patients. LEUKEMIA RESEARCH. vol. 31(2), pp. 163 - 167 ISSN: 0145-2126.
[ARTICOLO].
IACOBUCCI I., GALLETTI L, AMABILE M, SOVERINI S, BACCARANI M, MARTINELLI G. (2007). Monitoring BCR-ABL transcript levels by real-time
quantitative polymerase chain reaction: a linear regression equation to convert from BCR-ABL/B2M ratio to estimated BCR-ABL/ABL ratio.
HAEMATOLOGICA. vol. 92(3), pp. 429 - 430 ISSN: 0390-6078. [ARTICOLO].
ROSTI G, IACOBUCCI I., BASSI S, CASTAGNETTI F, AMABILE M, CILLONI D, POERIO A, SOVERINI S, PALANDRI F, REGE CAMBRIN G,
IULIANO F, ALIMENA G, LATAGLIATA R, TESTONI N, PANE F, SAGLIO G, BACCARANI M, MARTINELLI G. (2007). Impact of age on the outcome of
patients with chronic myeloid leukemia in late chronic phase: results of a phase II study of the GIMEMA CML Working Party. HAEMATOLOGICA. vol. 92,
pp. 101 - 105 ISSN: 0390-6078. [ARTICOLO].
SOVERINI S, COLAROSSI S, GNANI A, CASTAGNETTI F, ROSTI G, BOSI C, PAOLINI S, RONDONI M, PICCALUGA PP, PALANDRI F, GIANNOULIA
P, MARZOCCHI G, LUATTI S, TESTONI N, IACOBUCCI I., CILLONI D, SAGLIO G, BACCARANI M, MARTINELLI G. (2007). Resistance to dasatinib in
Philadelphia-positive leukemia patients and the presence or the selection of mutations at residues 315 and 317 in the BCR-ABL kinase domain.
HAEMATOLOGICA. vol. 92(3), pp. 401 - 404 ISSN: 0390-6078. [ARTICOLO].
SOVERINI S, IACOBUCCI I., BACCARANI M, MARTINELLI G. (2007). Targeted therapy and the T315I mutation in Philadelphia-positive leukemias.
HAEMATOLOGICA. vol. 92(4), pp. 437 - 439 ISSN: 0390-6078. [ARTICOLO].
IACOBUCCI I., SAGLIO G, ROSTI G, TESTONI N, PANE F, AMABILE M, POERIO A, SOVERINI S, BASSI S, CILLONI D, BASSAN R, BRECCIA M,
LAURIA F, IZZO B, MERANTE S, FRASSONI F, PAOLINI S, MONTEFUSCO E, BACCARANI M, MARTINELLI G. (2006). Achieving a major molecular
response at the time of a complete cytogenetic response (CCgR) predicts a better duration of CCgR in imatinib-treated chronic myeloid leukemia patients.
CLINICAL CANCER RESEARCH. vol. 12, pp. 3037 - 3042 ISSN: 1078-0432. [ARTICOLO].
MARTINELLI G, IACOBUCCI I., ROSTI G, PANE F, AMABILE M, CASTAGNETTI F, CILLONI D, SOVERINI S, TESTONI N, SPECCHIA G, MERANTE
S, ZACCARIA A, FRASSONI F, SAGLIO G, BACCARANI M. (2006). Prediction of response to imatinib by prospective quantitation of BCR-ABL transcript in
late chronic phase chronic myeloid leukemia patients. ANNALS OF ONCOLOGY. vol. 17, pp. 495 - 502 ISSN: 0923-7534. [ARTICOLO].
MARTINELLI G, IACOBUCCI I., SOVERINI S, CILLONI D, SAGLIO G, PANE F, BACCARANI M. (2006). Monitoring minimal residual disease and
controlling drug resistance in chronicmyeloid leukaemia patients in treatment with imatinib as a guide to clinicalmanagement. HEMATOLOGICAL
ONCOLOGY. vol. 24, pp. 196 - 204 ISSN: 0278-0232. [ARTICOLO].
MARTINELLI G, IACOBUCCI I., ROSTI G, PANE F, AMABILE M, CASTAGNETTI F, CILLONI D, SOVERINI S, TESTONI N, SPECCHIA G, MERANTE
S, ZACCARIA A, FRASSONI F, SAGLIO G, BACCARANI M. (2006). Prediction of response to imatinib by prospective quantitation of BCR-ABL transcript in
late chronic phase chronic myeloid leukemia patients. ANNALS OF ONCOLOGY. vol. 17(3), pp. 495 - 502 ISSN: 0923-7534. [ARTICOLO].
SOVERINI S., TASCO G., GRAFONE T., COLAROSSI S., GNANI A., ROSTI G., CASTAGNETTI F., PALANDRI F., RONDONI M., POERIO A.,
MUR - BANDO 2007 - * MODELLO B *
- 16 -
Ministero dell ,Università e della Ricerca
IACOBUCCI I., AMABILE M., BACCARANI M., CASADIO R., MARTINELLI G. (2006). Binding Mode Of The Novel Dual SRC and ABL Inhibitor SKI-606
To The BCR-ABL Kinase As Predicted By Molecular Docking Studies. HAEMATOLOGICA. vol. 91(s3), pp. 31 ISSN: 0390-6078. [ATTO DI CONVEGNO].
SOVERINI S., TASCO G., GRAFONE T., COLAROSSI S., GNANI A., ROSTI G., CASTAGNETTI F., PALANDRI F., RONDONI M., POERIO A.,
IACOBUCCI I., AMABILE M., BACCARANI M., CASADIO R., MARTINELLI G. (2006). Dual Src/Abl inhibitor SKI-606 binding mode in BCR-ABL kinase
hypothesized on the basis of molecular docking studies. HAEMATOLOGICA. vol. 91(s1), pp. 27 ISSN: 0390-6078. [ATTO DI CONVEGNO].
SOVERINI S, COLAROSSI S, GNANI A, ROSTI G, CASTAGNETTI F, POERIO A, IACOBUCCI I., AMABILE M, ABRUZZESE E, ORLANDI E,
RADAELLI F, CICCONE F, TIRIBELLI M, DI LORENZO R, CARACCIOLO C, IZZO B, PANE F, SAGLIO G, BACCARANI M, MARTINELLI G, ON
BEHALF OF THE GIMEMA WORKING PARTY ON CHRONIC MYELOID LEUKEMIA (GIMEMA-CML). (2006). Contribution Of ABL Kinase Domain
Mutations To Imatinib Resistance In Different Subsets Of Philadelphia-Positive Patients. By The GIMEMA Working Party On Chronic Myeloid Leukemia.
CLINICAL CANCER RESEARCH. vol. 12(24), pp. 7374 - 7379 ISSN: 1078-0432. [ARTICOLO].
SOVERINI S, MARTINELLI G, COLAROSSI S, GNANI A, CASTAGNETTI F, ROSTI G, BOSIC, PAOLINI S, RONDONI M, PICCALUGA PP, PALANDRI
F, GIANNOULIA P, MARZOCCHI G, LUATTI S, TESTONI N, IACOBUCCI I., CILLONI D, SAGLIO G, BACCARANI M. (2006). Presence or the
emergence of a F317L BCR-ABL mutation may be associated withresistance to dasatinib in Philadelphia chromosome-positive leukemia. JOURNAL OF
CLINICAL ONCOLOGY. vol. 24, pp. e51 - e52 ISSN: 0732-183X. [ARTICOLO].
3.
PAOLINI Stefania
Curriculum:
CURRICULUM VITAE
STEFANIA PAOLINI
Nata a Bologna il 01/10/1978
Residente in via Gramsci, 8
40012 Calderara di Reno (BO)
339-2231947
[email protected]
TITOLI DI STUDIO
Diploma di maturità scientifica conseguito presso il Liceo Scientifico "A. Righi" di Bologna nell' anno accademico 1996-1997
Laurea in Medicina e Chirurgia conseguita presso l'Università degli Studi di Bologna il 18/07/2003 con voto 110/110 con lode
Scuola di Specializzazione in Ematologia presso l'Istituto di Ematologia e Oncologia Medica L&A Seragnoli, Azienda ospedaliera "S.Orsola- Malpighi"
Bologna (2003-2007)
PUBBLICAZIONI
1) Iacobucci I, Saglio G, Rosti G, Testoni N, Pane F, Amabile M, Poerio A, Soverini S, Bassi S, Cilloni D, Bassan R, Breccia M, Lauria F, Izzo B, Merante S,
Frassoni F, Paolini S, Montefusco E, Baccarani M, Martinelli G; GIMEMA Working Party on Chronic Myeloid Leukemia.
Achieving a major molecular response at the time of a complete cytogenetic response (CCgR) predicts a better duration of CCgR in imatinib-treated chronic
myeloid leukemia patients.
Clin Cancer Res. 2006 May 15;12(10):3037-42.
PMID: 16707599 [PubMed - indexed for MEDLINE]
2) Stanzani M, Vianelli N, Bandini G, Paolini S, Arpinati M, Bonifazi F, Giannini B, Agostinelli C, Baccarani M, Ricci P. Related Articles, Links
Successful treatment of disseminated Fusariosis after allogeneic hematopoietic stem cell transplantation with the combination of voriconazole and liposomal
amphotericin B.
J Infect. 2006 Dec;53(6):e243-6. Epub 2006 Apr 11.
PMID: 16584783 [PubMed - indexed for MEDLINE]
3) Malagola M, Martinelli G, Rondoni M, Paolini S, Gaitani S, Arpinati M, Piccaluga PP, Amabile M, Basi C, Ottaviani E, Candoni A, Gottardi E, Cilloni D,
Bocchia M, Saglio G, Lauria F, Fanin R, Visani G, Marre MC, Maderna M, Rancati F, Vinaccia V, Russo D, Baccarani M. Related Articles, Links
Imatinib mesylate in the treatment of c-kit-positive acute myeloid leukemia: is this the real target?
Blood. 2005 Jan 15;105(2):904; author reply 905. No abstract available.
PMID: 15632213 [PubMed - indexed for MEDLINE]
4) Piccaluga PP, Malagola M, Amabile M, Rondoni M, Paolini S, Pane F, Russo D, Visani G, Baccarani M, Martinelli G. Related Articles, Links The
achievement of molecular complete remission during treatment with imatinib mesylate correlates with relapse-free survival in bcr/abl-positive acute lymphoid
leukemia patients.
Haematologica. 2004 Oct;89(10):1269-71.
PMID: 15477218 [PubMed - indexed for MEDLINE]
5) Piccaluga PP, Malagola M, Rondoni M, Amabile M, Paolini S, Soverini S, Gaitani S, Visani G, Baccarani M, Martinelli G. Related Articles, Links
Dose increase of imatinib mesylate may overcome acquired resistance in bcr/abl-positive acute lymphoid leukaemia.
Eur J Haematol. 2004 Apr;72(4):302-3. No abstract available.
PMID: 15089772 [PubMed - indexed for MEDLINE]
6)Piccaluga PP, Rondoni M, Paolini S, Rosti G, Martinelli G, Baccarani M
Imatinib mesylate in the treatment of hematologic malignancies.
Expert Opin Biol Ther. 2007 Oct;7(10):1597-611. Review.
PMID: 17916051 [PubMed - indexed for MEDLINE]
7)Piccaluga PP, Paolini S, Martinelli G
Tyrosine kinase inhibitors for the treatment of Philadelphia chromosome-positive adult acute lymphoblastic leukemia.
Cancer. 2007 Sep 15;110(6):1178-86. Review.
Pubblicazioni:
IACOBUCCI I, SAGLIO G, ROSTI G, TESTONI N, PANE F, AMABILE M, POERIO A, SOVERINI S, BASSI S, CILLONI D, BASSAN R, BRECCIA M,
LAURIA F, IZZO B, MERANTE S, FRASSONI F, PAOLINI S., MONTEFUSCO E, BACCARANI M, MARTINELLI G. (2006). Achieving a major molecular
response at the time of a complete cytogenetic response (CCgR) predicts a better duration of CCgR in imatinib-treated chronic myeloid leukemia patients.
CLINICAL CANCER RESEARCH. ISSN: 1078-0432.
MUR - BANDO 2007 - * MODELLO B *
- 17 -
Ministero dell ,Università e della Ricerca
STANZANI M, VIANELLI N, BANDINI G, PAOLINI S., ARPINATI M, BONIFAZI F, GIANNINI B, AGOSTINELLI C, BACCARANI M, RICCI P. (2006).
Successful treatment of disseminated Fusariosis after allogeneic hematopoietic stem cell transplantation with the combination of voriconazole and liposomal
amphotericin B. JOURNAL OF INFECTION. ISSN: 0163-4453.
MALAGOLA M, MARTINELLI G, RONDONI M, PAOLINI S., GAITANI S, ARPINATI M, PICCALUGA PP, AMABILE M, BASI C, OTTAVIANI E,
CANDONI A, GOTTARDI E, CILLONI D, BOCCHIA M, SAGLIO G, LAURIA F, FANIN R, VISANI G, MARRE MC, MADERNA M, RANCATI F, VINACCIA
V, RUSSO D, BACCARANI M. (2005). Imatinib mesylate in the treatment of c-kit-positive acute myeloid leukemia: is this the real target?. BLOOD. ISSN:
0006-4971.
PICCALUGA PP, MALAGOLA M, AMABILE M, RONDONI M, PAOLINI S., PANE F, RUSSO D, VISANI G, BACCARANI M, MARTINELLI G. (2004).
The achievement of molecular complete remission during treatment with imatinib mesylate correlates with relapse-free survival in bcr/abl-positive acute
lymphoid leukemia patients. HAEMATOLOGICA. ISSN: 0390-6078.
PICCALUGA PP, MALAGOLA M, RONDONI M, AMABILE M, PAOLINI S., SOVERINI S, GAITANI S, VISANI G, BACCARANI M., MARTINELLI G.
(2004). Dose increase of imatinib mesylate may overcome acquired resistance in bcr/abl-positive acute lymphoid leukaemia. EUROPEAN JOURNAL OF
HAEMATOLOGY. vol. 72 (4), pp. 302 - 303 ISSN: 0902-4441. [ARTICOLO].
4.
PAPAYANNIDIS Cristina
Curriculum:
Cristina Papayannidis
nata a Cesenatico il 20/12/1980
Diploma di Maturità scientifica nel 1999, con votazione 98/100.
Laurea in Medicina e Chirurgia presso l'Università di Bologna il 10/10/2005, con votazione di 110/110 e lode.
Dal 01/07/2006 iscritta alla scuola di Specializzazione in Ematologia dell'Università di Bologna.
Principali aree di interesse e di ricerca clinica: Sindromi Mieloproliferative e Linfoproliferative Acute e Croniche.
Inserita in trials clinici riguardanti: inibitori di Aurora Kinasi, inibitori Istone deacetilasi.
Pubblicazioni:
PICCALUGA PP, ASCANI S, AGOSTINELLI C, PAOLINI S, LATERZA C, PAPAYANNIDIS C., MARTINELLI G, VISANI G, BACCARANI M, PILERI SA.
(2007). Myeloid sarcoma of liver: an unusual cause of jaundice. Report of three cases and review of literature. HISTOPATHOLOGY. ISSN: 0309-0167.
PICCALUGA PP, FINELLI C, VIGNA E, AGOSTINELLI C, BACCI F, PAOLINI S, PAPAYANNIDIS C., LATERZA C, MARTINELLI G, PILERI SA,
BACCARANI M. (2007). Paraplegia due to a paravertebral extramedullary haemopoiesis in a patient with polycythaemia vera. JOURNAL OF CLINICAL
PATHOLOGY. ISSN: 0021-9746.
PICCALUGA PP, VIGNA E, PLACCI A, AGOSTINELLI C, LATERZA C, PAPAYANNIDIS C., LEONE O, MARTINELLI G, ZINZANI PL, BACCARANI M,
PILERI SA. (2006). Primary cardiac non-Hodgkin lymphoma presenting with atrial flutter and pericardial effusion. BRITISH JOURNAL OF
HAEMATOLOGY. ISSN: 0007-1048.
Testo inglese
1.
GIULIANI Silvia
Curriculum:
Silvia Giuliani
Date and place of birth: 08/08/1975, Pescara
Address: via Juri Gagarin 23 40100 Bologna, Italy
Email: [email protected]
Nationality: Italy
Education
PhD student in Biochemistry at the University of Ferrara
Master in Bioinformatics at the University Bicocca of Milano
Degree in chemistry at the University of Chieti
Professional Experience:
CINECA since 2004. I' m involved in a project to create an informatic infrastructure to store and analyze data microarray. I' m also involved in the molecular
dynamics study of the protein NFkB.
Stage in biochemistry at the University of Chieti where I was involved in the conformation study of prion proteins.
Computer and language skills
Inglesh & French
Operative System: windows, linux
Programming Language: perl, java, mysql
2.
IACOBUCCI Ilaria
Curriculum:
PERSONAL DATA
Name and surname:Ilaria Iacobucci
Date and placeof birth:September, 18, 1980.Termoli, Campobasso.
Nationality: Italian
Address(es):4, Renato Simoni Bologna 40127
Italy
Telephone(s):0039-051 6363791
0039-348 7622848
Fax:0039-051 6364037
E-mail: [email protected]
MUR - BANDO 2007 - * MODELLO B *
- 18 -
Ministero dell ,Università e della Ricerca
EDUCATION AND TRAINING
°09/1994-06/1998Liceo Scientifico Statale "A.. Romita", Campobasso.
High school, final mark 60/60
°09/1999-07/2004University of Bologna
Graduated cum laude in Biotechnology (Faculty of Medicine) on the 9th of July 2004.
Thesis title: "La quantificazione del trascritto Bcr-Abl è utile per predire la risposta all'imatinib nella Leucemia mieloide Cronica".
Experimental thesis in Dept. of Hematology:
Institute of Hematology and Medical Oncology "L. e A. Seràgnoli",
Policlinico "S Orsola", Bologna
Prof. Michele Baccarani
°01/2005-on going
University of Bologna
PhD in Clinical and Experimental Hematology
°08/2005-02/2006
III Medizinische Klinik Hamatologie/Oncologie
Universitat Heidelberg, Mannheim, Germany
Prof. Dr. Andreas Hochhaus
Marco Polo project focused on "standardization of BCR-ABL quantification and analysis of BCR-ABL mutations in patients treated with tyrosine kinase
inhibitors".
TECHNICAL EXPERTISE
Molecular biology techniques for diagnostics and hematologic and cancer research:
-Mononuclear cell separation from bone marrow and peripheral blood;
-Extraction of DNA and RNA from human blood or cells in GITC and Trizol;
-Spettrophotometric dosage of proteins and nucleic acids;
-RT-PCR; Nested PCR;
-Real-time quantitative PCR;
-Cell cultures;
-Nucleic acid sequencing;
-Microarry experiments.
FOREIGN LANGUAGES
° English
European Level:Listening B2
Reading B2
Writing B2
Spoken interaction B2
COMPUTER SKILLS AND COMPETENCES
-Competent with most Microsoft Office programmes (e.g. Word, Excel, Access and
-Good knowledge of graphic design applications (GraphPad, PhotoShop).
-Good knowledge of bibliographic design applications (Endnote).
-Use of databeses online (GeneBank, SwissProt, PDB).
-Use of Chromas and Oligo softwares.
-Use of Affymetrix softwares.
-Alignment of sequences by BLAST/FASTA.
PowerPoint);
TEACHING ACTIVITY
-"Modalità di prelievi" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", April 10-12 2006).
-"Manipolazione del campione" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", April 10-12 2006).
-"Estrazione dell'acido nucleico e retrotrascrizione" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", April 10-12 2006).
-"Report dei risultati" in "X-Phile, monitoring the CML:from bench to bedside" (Istituto "Seràgnoli", April 10-12 2006).
SCIENTIFIC ACTIVITY, PARTECIPATIONS TO CONGRESSES
1.Adult Stem Cells. A Challenge for the future, Bologna, March 21-23, 2004
2.46th Annual Meeting of the American Society of Hematology (ASH). San Diego, California December 4-7, 2004.
3.Applicazioni in Medicina Molecolare, Bologna, April 4, 2005.
4.GIMEMA CML Working Party, Bologna-Jolly Hotel De La Gare, April 18, 2005
5.10th Congress of the European Hematology Association Stockholm, Sweden. June 2-5, 2005
6.40° Congresso Nazionale SIE, Bergamo, July 3-6, 2005.
7.New drugs in Hematology, Bologna, October 9-11, 2005.
8.Third ESH International Conference on chronic Myeloid leukaemia in the 21st century. Genoa, Novenber 11-13, 2005-Genoa.
9.47th Annual Meeting of the American Society of Hematology (ASH). December 10-13, 2005 Atlanta, GA.
10.10th Congress of the European Hematology Association Amsterdam. 15 - 18 June, 2006.
11.IX Congresso Nazionale SIES, Napoli, September 20-22, 2006.
12.Dasatinib CA180-043 Investigator Meeting, Rome October 16, 2006.
13.7900 HT Real Time PCR System, Bologna September 18-19, 2006.
14.48th Annual Meeting of the American Society of Hematology (ASH). December 9-12, 2006. Orlando, Florida.
15.AACR, American Association for Cancer Research Annual Meeting 2007. April 14-18, 2007. Los Angeles, California.
16.12th Congress of the European Hematology Association. Vienna, Austria, June 7-10, 2007.
Publications:
BACCARANI M, CILLONI D, RONDONI M, OTTAVIANI E, MESSA F, MERANTE S, TIRIBELLI M, BUCCISANO F, TESTONI N, GOTTARDI E, DE
VIVO A, GIUGLIANO E, IACOBUCCI I., PAOLINI S, SOVERINI S, ROSTI G, RANCATI F, ASTOLFI C, PANE F, SAGLIO G, MARTINELLI G. (2007). The
MUR - BANDO 2007 - * MODELLO B *
- 19 -
Ministero dell ,Università e della Ricerca
efficacy of imatinib mesylate in patients with FIP1L1-PDGFRalpha-positive hypereosinophilic syndrome. Results of a multicenter prospective study.
HAEMATOLOGICA. vol. 92(9), pp. 1173 - 1179 ISSN: 0390-6078. [ARTICOLO].
BUSSOLARI R, CANDINI O, COLOMER D, CORRADINI F, GUERZONI C, MARIANI SA, CATTELANI S, SILVESTRI C, PECORARI L, IACOBUCCI I.,
SOVERINI S, FASANO T, MARTINELLI G, CERVANTES F, CALABRETTA B. (2007). Coding sequence and intron-exon junctions of the c-myb gene are
intact in the chronic phase and blast crisis stages of chronic myeloid leukemia patients. LEUKEMIA RESEARCH. vol. 31(2), pp. 163 - 167 ISSN: 0145-2126.
[ARTICOLO].
IACOBUCCI I., GALLETTI L, AMABILE M, SOVERINI S, BACCARANI M, MARTINELLI G. (2007). Monitoring BCR-ABL transcript levels by real-time
quantitative polymerase chain reaction: a linear regression equation to convert from BCR-ABL/B2M ratio to estimated BCR-ABL/ABL ratio.
HAEMATOLOGICA. vol. 92(3), pp. 429 - 430 ISSN: 0390-6078. [ARTICOLO].
ROSTI G, IACOBUCCI I., BASSI S, CASTAGNETTI F, AMABILE M, CILLONI D, POERIO A, SOVERINI S, PALANDRI F, REGE CAMBRIN G,
IULIANO F, ALIMENA G, LATAGLIATA R, TESTONI N, PANE F, SAGLIO G, BACCARANI M, MARTINELLI G. (2007). Impact of age on the outcome of
patients with chronic myeloid leukemia in late chronic phase: results of a phase II study of the GIMEMA CML Working Party. HAEMATOLOGICA. vol. 92,
pp. 101 - 105 ISSN: 0390-6078. [ARTICOLO].
SOVERINI S, COLAROSSI S, GNANI A, CASTAGNETTI F, ROSTI G, BOSI C, PAOLINI S, RONDONI M, PICCALUGA PP, PALANDRI F, GIANNOULIA
P, MARZOCCHI G, LUATTI S, TESTONI N, IACOBUCCI I., CILLONI D, SAGLIO G, BACCARANI M, MARTINELLI G. (2007). Resistance to dasatinib in
Philadelphia-positive leukemia patients and the presence or the selection of mutations at residues 315 and 317 in the BCR-ABL kinase domain.
HAEMATOLOGICA. vol. 92(3), pp. 401 - 404 ISSN: 0390-6078. [ARTICOLO].
SOVERINI S, IACOBUCCI I., BACCARANI M, MARTINELLI G. (2007). Targeted therapy and the T315I mutation in Philadelphia-positive leukemias.
HAEMATOLOGICA. vol. 92(4), pp. 437 - 439 ISSN: 0390-6078. [ARTICOLO].
IACOBUCCI I., SAGLIO G, ROSTI G, TESTONI N, PANE F, AMABILE M, POERIO A, SOVERINI S, BASSI S, CILLONI D, BASSAN R, BRECCIA M,
LAURIA F, IZZO B, MERANTE S, FRASSONI F, PAOLINI S, MONTEFUSCO E, BACCARANI M, MARTINELLI G. (2006). Achieving a major molecular
response at the time of a complete cytogenetic response (CCgR) predicts a better duration of CCgR in imatinib-treated chronic myeloid leukemia patients.
CLINICAL CANCER RESEARCH. vol. 12, pp. 3037 - 3042 ISSN: 1078-0432. [ARTICOLO].
MARTINELLI G, IACOBUCCI I., ROSTI G, PANE F, AMABILE M, CASTAGNETTI F, CILLONI D, SOVERINI S, TESTONI N, SPECCHIA G, MERANTE
S, ZACCARIA A, FRASSONI F, SAGLIO G, BACCARANI M. (2006). Prediction of response to imatinib by prospective quantitation of BCR-ABL transcript in
late chronic phase chronic myeloid leukemia patients. ANNALS OF ONCOLOGY. vol. 17, pp. 495 - 502 ISSN: 0923-7534. [ARTICOLO].
MARTINELLI G, IACOBUCCI I., SOVERINI S, CILLONI D, SAGLIO G, PANE F, BACCARANI M. (2006). Monitoring minimal residual disease and
controlling drug resistance in chronicmyeloid leukaemia patients in treatment with imatinib as a guide to clinicalmanagement. HEMATOLOGICAL
ONCOLOGY. vol. 24, pp. 196 - 204 ISSN: 0278-0232. [ARTICOLO].
MARTINELLI G, IACOBUCCI I., ROSTI G, PANE F, AMABILE M, CASTAGNETTI F, CILLONI D, SOVERINI S, TESTONI N, SPECCHIA G, MERANTE
S, ZACCARIA A, FRASSONI F, SAGLIO G, BACCARANI M. (2006). Prediction of response to imatinib by prospective quantitation of BCR-ABL transcript in
late chronic phase chronic myeloid leukemia patients. ANNALS OF ONCOLOGY. vol. 17(3), pp. 495 - 502 ISSN: 0923-7534. [ARTICOLO].
SOVERINI S., TASCO G., GRAFONE T., COLAROSSI S., GNANI A., ROSTI G., CASTAGNETTI F., PALANDRI F., RONDONI M., POERIO A.,
IACOBUCCI I., AMABILE M., BACCARANI M., CASADIO R., MARTINELLI G. (2006). Binding Mode Of The Novel Dual SRC and ABL Inhibitor SKI-606
To The BCR-ABL Kinase As Predicted By Molecular Docking Studies. HAEMATOLOGICA. vol. 91(s3), pp. 31 ISSN: 0390-6078. [ATTO DI CONVEGNO].
SOVERINI S., TASCO G., GRAFONE T., COLAROSSI S., GNANI A., ROSTI G., CASTAGNETTI F., PALANDRI F., RONDONI M., POERIO A.,
IACOBUCCI I., AMABILE M., BACCARANI M., CASADIO R., MARTINELLI G. (2006). Dual Src/Abl inhibitor SKI-606 binding mode in BCR-ABL kinase
hypothesized on the basis of molecular docking studies. HAEMATOLOGICA. vol. 91(s1), pp. 27 ISSN: 0390-6078. [ATTO DI CONVEGNO].
SOVERINI S, COLAROSSI S, GNANI A, ROSTI G, CASTAGNETTI F, POERIO A, IACOBUCCI I., AMABILE M, ABRUZZESE E, ORLANDI E,
RADAELLI F, CICCONE F, TIRIBELLI M, DI LORENZO R, CARACCIOLO C, IZZO B, PANE F, SAGLIO G, BACCARANI M, MARTINELLI G, ON
BEHALF OF THE GIMEMA WORKING PARTY ON CHRONIC MYELOID LEUKEMIA (GIMEMA-CML). (2006). Contribution Of ABL Kinase Domain
Mutations To Imatinib Resistance In Different Subsets Of Philadelphia-Positive Patients. By The GIMEMA Working Party On Chronic Myeloid Leukemia.
CLINICAL CANCER RESEARCH. vol. 12(24), pp. 7374 - 7379 ISSN: 1078-0432. [ARTICOLO].
SOVERINI S, MARTINELLI G, COLAROSSI S, GNANI A, CASTAGNETTI F, ROSTI G, BOSIC, PAOLINI S, RONDONI M, PICCALUGA PP, PALANDRI
F, GIANNOULIA P, MARZOCCHI G, LUATTI S, TESTONI N, IACOBUCCI I., CILLONI D, SAGLIO G, BACCARANI M. (2006). Presence or the
emergence of a F317L BCR-ABL mutation may be associated withresistance to dasatinib in Philadelphia chromosome-positive leukemia. JOURNAL OF
CLINICAL ONCOLOGY. vol. 24, pp. e51 - e52 ISSN: 0732-183X. [ARTICOLO].
3.
PAOLINI Stefania
Curriculum:
STEFANIA PAOLINI
Born in Bologna on 01/10/1978
Address is: via Gramsci, 8
40012 Calderara di Reno (BO)
339-2231947
[email protected]
EDUCATION
1996-1997: Liceo Scientifico "A. Righi", Bologna high school, final mark 60/60
1997-2003: University of Bologna
Graduated cum laude in Medicine on 18/07/2003
2003-2007 : Specialization in Hematology at Istituto di Ematologia e Oncologia Medica L&A Seragnoli, Azienda ospedaliera "S.Orsola- Malpighi" Bologna
Publications
1)Iacobucci I, Saglio G, Rosti G, Testoni N, Pane F, Amabile M, Poerio A, Soverini S, Bassi S, Cilloni D, Bassan R, Breccia M, Lauria F, Izzo B, Merante S,
Frassoni F, Paolini S, Montefusco E, Baccarani M, Martinelli G; GIMEMA Working Party on Chronic Myeloid Leukemia.Clin Cancer Res. 2006 May
15;12(10):3037-42.
PMID: 16707599 [PubMed - indexed for MEDLINE]
2) Stanzani M, Vianelli N, Bandini G, Paolini S, Arpinati M, Bonifazi F, Giannini B, Agostinelli C, Baccarani M, Ricci P. Related Articles, Links
Successful treatment of disseminated Fusariosis after allogeneic hematopoietic stem cell transplantation with the combination of voriconazole and liposomal
amphotericin B.
J Infect. 2006 Dec;53(6):e243-6. Epub 2006 Apr 11.
PMID: 16584783 [PubMed - indexed for MEDLINE]
3) Malagola M, Martinelli G, Rondoni M, Paolini S, Gaitani S, Arpinati M, Piccaluga PP, Amabile M, Basi C, Ottaviani E, Candoni A, Gottardi E, Cilloni D,
Bocchia M, Saglio G, Lauria F, Fanin R, Visani G, Marre MC, Maderna M, Rancati F, Vinaccia V, Russo D, Baccarani M. Related Articles, Links
Imatinib mesylate in the treatment of c-kit-positive acute myeloid leukemia: is this the real target?
Blood. 2005 Jan 15;105(2):904; author reply 905. No abstract available.
PMID: 15632213 [PubMed - indexed for MEDLINE]
MUR - BANDO 2007 - * MODELLO B *
- 20 -
Ministero dell ,Università e della Ricerca
4) Piccaluga PP, Malagola M, Amabile M, Rondoni M, Paolini S, Pane F, Russo D, Visani G, Baccarani M, Martinelli G. Related Articles, Links The
achievement of molecular complete remission during treatment with imatinib mesylate correlates with relapse-free survival in bcr/abl-positive acute lymphoid
leukemia patients.
Haematologica. 2004 Oct;89(10):1269-71.
PMID: 15477218 [PubMed - indexed for MEDLINE]
5) Piccaluga PP, Malagola M, Rondoni M, Amabile M, Paolini S, Soverini S, Gaitani S, Visani G, Baccarani M, Martinelli G. Related Articles, Links
Dose increase of imatinib mesylate may overcome acquired resistance in bcr/abl-positive acute lymphoid leukaemia.
Eur J Haematol. 2004 Apr;72(4):302-3. No abstract available.
PMID: 15089772 [PubMed - indexed for MEDLINE]
6)Piccaluga PP, Rondoni M, Paolini S, Rosti G, Martinelli G, Baccarani M
Imatinib mesylate in the treatment of hematologic malignancies.
Expert Opin Biol Ther. 2007 Oct;7(10):1597-611. Review.
PMID: 17916051 [PubMed - indexed for MEDLINE]
7)Piccaluga PP, Paolini S, Martinelli G
Tyrosine kinase inhibitors for the treatment of Philadelphia chromosome-positive adult acute lymphoblastic leukemia.
Cancer. 2007 Sep 15;110(6):1178-86. Review.
Publications:
IACOBUCCI I, SAGLIO G, ROSTI G, TESTONI N, PANE F, AMABILE M, POERIO A, SOVERINI S, BASSI S, CILLONI D, BASSAN R, BRECCIA M,
LAURIA F, IZZO B, MERANTE S, FRASSONI F, PAOLINI S., MONTEFUSCO E, BACCARANI M, MARTINELLI G. (2006). Achieving a major molecular
response at the time of a complete cytogenetic response (CCgR) predicts a better duration of CCgR in imatinib-treated chronic myeloid leukemia patients.
CLINICAL CANCER RESEARCH. ISSN: 1078-0432.
STANZANI M, VIANELLI N, BANDINI G, PAOLINI S., ARPINATI M, BONIFAZI F, GIANNINI B, AGOSTINELLI C, BACCARANI M, RICCI P. (2006).
Successful treatment of disseminated Fusariosis after allogeneic hematopoietic stem cell transplantation with the combination of voriconazole and liposomal
amphotericin B. JOURNAL OF INFECTION. ISSN: 0163-4453.
MALAGOLA M, MARTINELLI G, RONDONI M, PAOLINI S., GAITANI S, ARPINATI M, PICCALUGA PP, AMABILE M, BASI C, OTTAVIANI E,
CANDONI A, GOTTARDI E, CILLONI D, BOCCHIA M, SAGLIO G, LAURIA F, FANIN R, VISANI G, MARRE MC, MADERNA M, RANCATI F, VINACCIA
V, RUSSO D, BACCARANI M. (2005). Imatinib mesylate in the treatment of c-kit-positive acute myeloid leukemia: is this the real target?. BLOOD. ISSN:
0006-4971.
PICCALUGA PP, MALAGOLA M, AMABILE M, RONDONI M, PAOLINI S., PANE F, RUSSO D, VISANI G, BACCARANI M, MARTINELLI G. (2004).
The achievement of molecular complete remission during treatment with imatinib mesylate correlates with relapse-free survival in bcr/abl-positive acute
lymphoid leukemia patients. HAEMATOLOGICA. ISSN: 0390-6078.
PICCALUGA PP, MALAGOLA M, RONDONI M, AMABILE M, PAOLINI S., SOVERINI S, GAITANI S, VISANI G, BACCARANI M., MARTINELLI G.
(2004). Dose increase of imatinib mesylate may overcome acquired resistance in bcr/abl-positive acute lymphoid leukaemia. EUROPEAN JOURNAL OF
HAEMATOLOGY. vol. 72 (4), pp. 302 - 303 ISSN: 0902-4441. [ARTICOLO].
4.
PAPAYANNIDIS Cristina
Curriculum:
Cristina Papayannidis,
born in Cesenatico on December 20th 1980.
Degree at Bologna University in Medicine and Surgery on 10th October 2005, cum laude (110/110).
Diploma at Scientific Secondary School with mark of 98/100.
Since 1st July 2006 she's attending the post-graduate school of Hematology at the Institute of Hematology and Medical Oncology in Bologna University.
Main areas of research interest: Myeloproliferative and Lymphoproliferative Acute and Chronic disorders.
Sub-investigator in many clincal trials, regarding: Aurora Kinase Inhibitors, Histone deacetylase Inhibitors.
Publications:
PICCALUGA PP, ASCANI S, AGOSTINELLI C, PAOLINI S, LATERZA C, PAPAYANNIDIS C., MARTINELLI G, VISANI G, BACCARANI M, PILERI SA.
(2007). Myeloid sarcoma of liver: an unusual cause of jaundice. Report of three cases and review of literature. HISTOPATHOLOGY. ISSN: 0309-0167.
PICCALUGA PP, FINELLI C, VIGNA E, AGOSTINELLI C, BACCI F, PAOLINI S, PAPAYANNIDIS C., LATERZA C, MARTINELLI G, PILERI SA,
BACCARANI M. (2007). Paraplegia due to a paravertebral extramedullary haemopoiesis in a patient with polycythaemia vera. JOURNAL OF CLINICAL
PATHOLOGY. ISSN: 0021-9746.
PICCALUGA PP, VIGNA E, PLACCI A, AGOSTINELLI C, LATERZA C, PAPAYANNIDIS C., LEONE O, MARTINELLI G, ZINZANI PL, BACCARANI M,
PILERI SA. (2006). Primary cardiac non-Hodgkin lymphoma presenting with atrial flutter and pericardial effusion. BRITISH JOURNAL OF
HAEMATOLOGY. ISSN: 0007-1048.
MUR - BANDO 2007 - * MODELLO B *
- 21 -