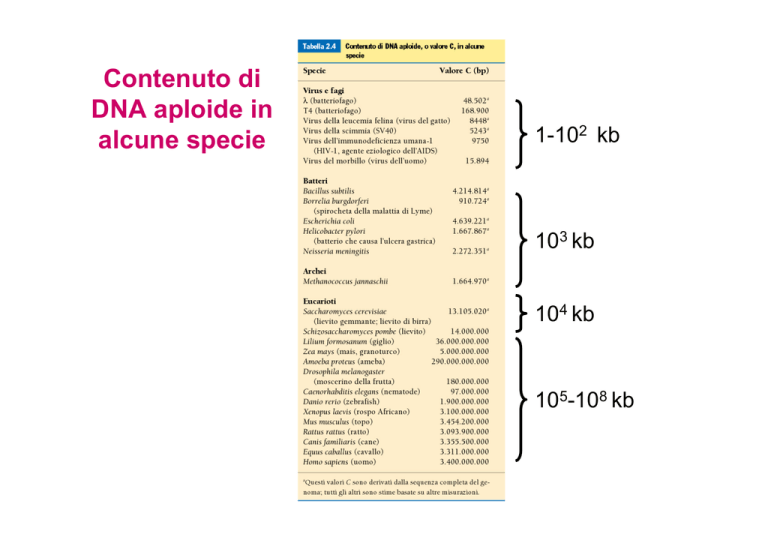
Contenuto di
DNA aploide in
alcune specie
1-102 kb
103 kb
104 kb
105-108 kb
Dimensioni del genoma
Paradosso del valore C
● Non c’è una correlazione tra la quantità di DNA e
la complessità di un organismo
● Non c’è una correlazione tra il numero di geni e
la complessità di un organismo
Struttura dei geni nei procarioti ed eucarioti
Distribuzione del numero di esoni per gene
Specie
Hemophilus influenzae
Methanococcus jannaschii
S. cerevisiae
Filamentous fungi
C. elegans
D. melanogaster
Chicken
Mammals
Numero
medio di
esoni
1
1
1
3
4
4
9
7
Lunghezza
media del
gene (kb)
1.0
1.0
1.6
1.5
4.0
11.3
13.9
16.6
Lunghezza
media del
mRNA (kb)
1.0
1.0
1.6
1.5
3.0
2.7
2.4
2.2
Classificazione del DNA eucariotico
La diversa natura delle sequenze ripetute
Geni presenti nel genoma degli eucarioti
in copie multiple
I geni delle proteine istoniche sono presenti nel
genoma in centinaia di copie ripetute in tandem
Geni per l’rRNA: l’organizzatore nucleolare
Le due famiglie dei geni per le globine α e β
Altri esempi di famiglie geniche sono:
I geni delle actine (da 5 a 30)
I geni delle cheratine (circa 20)
I geni della regione variabile delle immunoglobuline (500)
DNA satellite
Lunghezza della sequenza ripetuta: 10 bp circa
Numero di ripetizioni: fino a milioni di copie
Localizzazione: principalmente nei centromeri
Sequenze ripetitive di DNA
A
B
LA REPLICAZIONE
DEL DNA
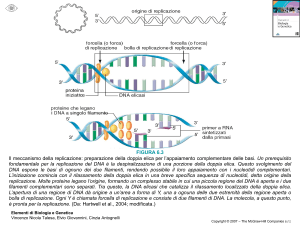
La replicazione del DNA
Il modello semiconservativo
La replicazione del DNA è il
processo mediante il quale una
cellula raddoppia il suo contenuto
di DNA prima di dividersi
La biochimica della replicazione del DNA
Componenti richiesti:
• DNA stampo
• DNA polimerasi
• dNTP
• ioni Mg2+
• Innesco
La biochimica della replicazione del DNA
Le attività delle DNA polimerasi
9 Attività di polimerasi, che catalizza l’allungamento
della catena polinucleotidica in direzione 5’→ 3’
9 Attività di esonucleasi 3’→ 5’, che rimuove le basi non
appaiate correttamente (correzione di bozze)
9 Attività di esonucleasi 5’→ 3’, che rimuove gli inneschi
durante la sintesi del filamento ritardato
Le tappe della replicazione del DNA
Inizio
Allungamento
Termine
Inizio della
replicazione del DNA
Enzimi coinvolti:
• le proteine iniziatrici
• DNA elicasi
• le SSB
• le topoisomerasi
Ori: origine di replicazione in E. Coli
(Sequenza di 254 bp con 4 siti di legame per
le proteine iniziatrici)
ARS: sequenze a
autonoma del lievito
replicazione
La funzione delle topoisomerasi
La direzione della replicazione
la direzione di sintesi è
sempre 5’→3’
La replicazione unidirezionale e bidirezionale
L’allungamento: la replicazione è semidiscontinua
Enzimi coinvolti:
• DNA primasi
• le DNA polimerasi
• la DNA ligasi
Allungamento (catena veloce)
La sintesi della catena veloce incomincia con la sintesi da parte di una
primasi di un breve primer di RNA (da 10 a 60 nucleotidi) all’origine
della replicazione.
I deossiribonucleotidi vengono poi aggiunti al primer dalla DNA
polimerasi III.
Una volta iniziata, la sintesi della catena veloce procede in modo
continuo, avanzando di pari passo con la forcella di replicazione.
Allungamento (catena lenta)
La sintesi della catena lenta viene compiuta tramite corti frammenti di
Okazaki.
Inizialmente il primer è sintetizzato dalla primasi.
Come nella sintesi della catena veloce, la DNA polimerasi III si lega al
primer e aggiunge deossiribonucleotidi fino al primer successivo
La DNA polimerasi I degrada i primer grazie alla sua attività
esonucleotidica 5’ – 3’ e li sostituisce con nuovi deossiribonucleotidi.
La DNA ligasi salda le interruzioni nello scheletro di zucchero-fosfato
lasciate dalle DNA polimerasi, catalizzando la formazione di un legame
fosfodiestere senza aggiungere altri nucleotidi alla catena.
Il DNA della catena lenta si piega
ad anello, di modo che i due
percorsi di polimerizzazione
possano procedere insieme
La terminazione
Origine
Forcella in
senso
antiorario
Trappola per
la forcella in
senso
orario
Forcella in
senso
orario
Trappola per
la forcella in
senso
antiorario
La proteina Tus (terminus utilization substance)
lega la sequenza Ter e blocca il movimento
dell’elicasi
causando
l’interruzione
della
replicazione
Le due forcelle di replicazione del cromosoma circolare di E. coli si incontrano
in una regione terminale contenente copie multiple di una sequenza di 20
coppie di basi detta Ter (“termine”). Quando una delle due forcelle di
replicazione incontra un complesso funzionale Tus-Ter, si ferma; l’altra forcella
si ferma quando incontra la prima forcella già ferma
Enzimi e fattori proteici richiesti nella replicazione
L’intero complesso è detto replisoma
• Proteine iniziatrici: denaturano il DNA in corrispondenza delle sequenze
Ori
• Elicasi: si muovono lungo il DNA e separano le catene usando l’energia
dell’ATP
• Topoisomerasi: risolvono la tensione topologica nella struttura ad elica del
DNA che si genera con la separazione delle catene
• Proteine che legano il DNA a singolo filamento: stabilizzano le catene
separate
• DNA primasi: sintetizza i primer (generalmente brevi frammenti di RNA)
• DNA polimerasi III: sintetizza il filamento guida e i frammenti di Okazaki
• DNA polimerasi I: rimuove i primer e li sostituisce con DNA
• DNA ligasi: ripara le interruzioni dei legami fosfodiesterici che rimangono
dopo l’azione delle DNA polimerasi
Le cinque DNA polimerasi di Escherichia coli
DNA
Polimerasi
Polimerasi
5’→3
Esonucleasi
3’→5’
Esonucleasi
5’→3’
Funzione
I
si
si
si
Rimozione e
sostituzione
degli inneschi
II
si
si
no
Riparazione del
DNA
III
si
si
no
Allungamento
del DNA
IV
si
no
no
Riparazione del
DNA
V
si
no
no
Riparazione del
DNA
La replicazione teta
La replicazione a circolo rotante
La replicazione lineare negli eucarioti
• Diversi tipi di DNA polimerasi: α, β, γ, δ, ε
• Origini di replicazione multiple
• Assemblaggio dei nucleosomi subito dopo la
replicazione
• Replicazione dei telomeri
Disassemblaggio e assemblaggio dei nucleosmi
La replicazione dei telomeri
La replicazione
dei telomeri
Caratteristiche delle replicazioni teta,
a circolo rotante e lineare
Modello di
replicazione
DNA
stampo
Teta
Rottura dei
filamenti
nucleotidici
Numero di
repliconi
Direzione della
replicazione
Prodotti della
replicazione
Circolare No
1
Unidirezionale
o bidirezionale
Due molecole
circolari
A circolo
rotante
Circolare Si
1
Unidirezionale
Una molecola
circolare e una
lineare che può
circolarizzarsi
Lineare
eucariotica
Lineare
Molti
Bidirezionale
Due molecole
lineari
No
La fedeltà nella replicazione del DNA
Domande
Le sequenze genomiche degli eucarioti
1. Che cosa è il valore C e il paradosso del valore C?
2. Fare un esempio di geni ripetuti in tandem
La replicazione del DNA
1. Perché è semiconservativa
2. Perché è semidiscontinua
3. Quali sono gli enzimi coinvolti
4. Cosa sono i frammenti di Okazaki
5. Differenze tra la replicazione nei batteri e quella negli eucarioti




![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)



![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)


