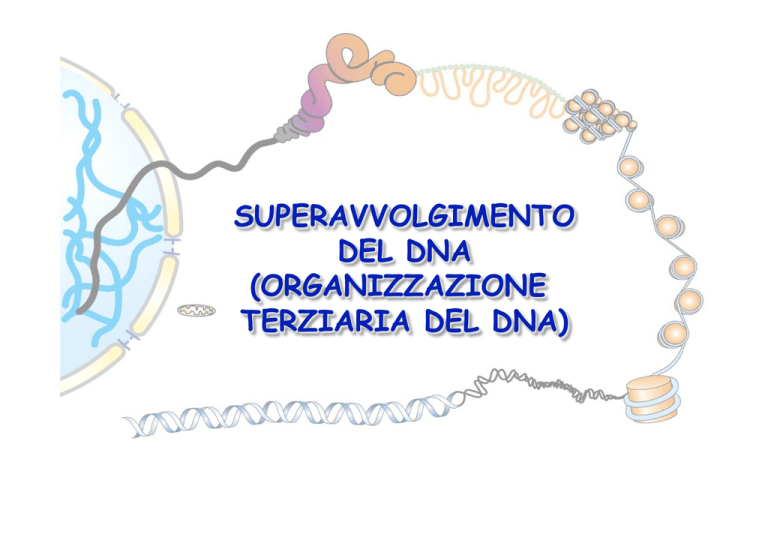
SUPERAVVOLGIMENTO
DEL DNA
(ORGANIZZAZIONE
TERZIARIA DEL DNA)
ORGANIZZAZIONE TERZIARIA DEL DNA
Il DNA cellulare contiene porzioni geniche e intergeniche, entrambe
necessarie per le funzioni vitali della cellula
GENE: regione del DNA che codifica la sequenza primaria di un
prodotto genico finale, che può essere un polipeptide o una molecola di
RNA con una specifica funzione strutturale o catalitica
Oltre ai geni il DNA contiene segmenti o sequenze con funzioni
regolatrici
SEQUENZE REGOLATRICI: segnali che indicano l inizio o la fine
dei geni, che influenzano la trascrizione dei geni, funzionano da inizio
della replicazione o della ricombinazione
Le molecole di DNA sono molto più lunghe degli involucri che le
contengono
Rivestimento proteico del batteriofago T2
circondato dalla sua molecola lineare di DNA
(batteriofago lisato)
Nel batteriofago non lisato tutto il DNA è
normalmente impaccato nella testa del fago
Le molecole di DNA sono molto più lunghe degli involucri che
le contengono
• La lunghezza totale del DNA di E.coli è circa 1 mm mentre la
lunghezza della cellula batterica è circa 1µm
• La lunghezza totale della molecola di DNA umano è circa 2m,
ma questa molecola deve stare in un nucleo di circa 10-15µm di
diametro
Il DNA cellulare deve essere strettamente
compattato, il che implica un elevato grado
di organizzazione strutturale
Il meccanismo di ripiegamento non deve
soltanto impacchettare il DNA, ma deve
anche permettere l accesso all informazione
contenuta nel DNA per lo svolgimento di
processi quali replicazione e trascrizione
Un importante proprietà intrinseca della
struttura terziaria del DNA è il
SUPERAVVOLGIMENTO = AVVOLGIMENTO
DI QUALCOSA GIA AVVOLTO
Il DNA è avvolto in forma di una doppia elica
nella quale entrambe le catene avvolte ruotano
intorno a un asse. Un ulteriore ripiegamento o
una torsione di tale asse su se stesso
determinano un superavvolgimento del DNA.
Il superavvolgimento del DNA è in genere la
manifestazione di una tensione strutturale; è
ubiquitario nel DNA cellulare ed è strettamente
regolato in ogni cellula.
ORGANIZZAZIONE DEL DNA
EUCARIOTICO IN CROMOSOMI
Nella cellula, il DNA è associato a proteine per formare un
complesso chiamato CROMOSOMA
Il termine cromosoma indica in generale una molecola di acido
nucleico depositaria dell informazione genetica di un virus, un
batterio, una cellula eucariotica o un organello.
La parola deriva dal greco: χρῶµα (chroma, colore) and σῶµα (soma,
corpo) a causa della proprietà dei cromosomi di essere colorate
intensamente da particolari coloranti.
Per questa ragione questo termine è stato originariamente usato
per indicare le inclusioni densamente colorate all interno dei nuclei
eucariotici che possono essere visualizzate al microscopio ottico dopo
che le cellule sono state sottoposte a colorazione.
L organizzazione strutturale del DNA
eucariotico a livelli superiori alla struttura
secondaria comporta la sua
SPIRALIZZAZIONE
Il livello di condensazione è diverso nelle
varie fasi del ciclo cellulare
La struttura dei cromosomi può essere
studiata solo durante la mitosi, quando essi
formano strutture più compatte. Durante
l interfase la maggior parte dei cromosomi
sono troppo despiralizzati e sottili perché si
possa osservarne l organizzazione
strutturale.
La determinazione del tipo di spiralizzazione
del DNA in ciascun cromosoma sembra
essere una delle funzioni delle regioni non
codificanti del DNA, di cui non è ancora
completamente noto il ruolo funzionale
La spiralizzazione del DNA nelle cellule eucariotiche è
importante
- per disporre in modo ordinato all interno del
nucleo le lunghissime catene di DNA
- la precisione con cui in una determinata cellula una
regione del genoma è ripiegata può influenzare in modo
decisivo l attività dei geni in questa regione
Un ruolo fondamentale nel processo di ripiegamento del DNA
negli eucarioti è svolto da proteine specializzate
Le proteine associate al DNA possono essere:
- ENZIMI che catalizzano importanti reazioni quali la sintesi di
RNA
- PROTEINE CON FUNZIONI STRUTTURALI che servono ad
organizzare il DNA all’interno del nucleo
Le proteine che, negli eucarioti, si legano al DNA sono classificate
in
- ISTONI (proteine strutturali)
- PROTEINE CROMOSOMICHE NON ISTONICHE (migliaia di
proteine diverse con numerose differenti funzioni)
ISTONI
Presenti nelle cellule in quantità enorme (circa 60 milioni di copie di
ciascun tipo per cellula)
La loro massa complessiva è quasi uguale a quella del DNA presente nella
cellula
Proteine relativamente piccole contenenti una quantità elevata di aa con
cariche positive (Arg e Lys) che favoriscono la formazione di uno stretto
legame con il DNA, indipendentemente dalla sequenza nucleotidica del
DNA stesso.
Il DNA legato agli istoni si chiama CROMATINA
-ISTONI NUCLEOSOMICI: piccole proteine responsabili del
ripiegarsi del DNA nei nucleosomi
H2A, H2B, H3, H4
Sono fra le proteine meglio conservate durante l evoluzione
-ISTONE H1: eccettuato il nucleo centrale della molecola, le
sequenze aminoacidiche sono meno conservate.
L unità strutturale della
cromatina è il NUCLEOSOMA,
responsabile dell aspetto a filo
di perle che la cromatina mostra
al microscopio elettronico quando
è stata trattata in modo da
distendere le ripiegature di ordine
superiore
Il nucleosoma è stato descritto da Roger Kornberg nel 1974
nucleo istonico: ottamero formato da due copie
di ciascun istone H2A, H2B, H3, H4
tratto di DNA che avvolge il nucleo istonico:
147bp
tratto di DNA di collegamento: 20-60bp
Le code N-terminali degli istoni nucleosomici
sporgono esternamente al nucleosoma
Il compattamento del DNA in nucleosomi produce
un filo di cromatina di circa 10nm di diametro
Il legame dell istone H1 porta ad un maggior
compattamento del nucleosoma
H1 si lega sia al nucleosoma che al DNA linker
Le molecole di istone H1 si legano al DNA in
maniera COOPERATIVA
Gli istoni H1 aiutano i
nucleosomi a condensarsi
nella fibra di 30nm
Le code N-terminali degli istoni del nucleosoma stabilizzano la fibra
di cromatina di 30nm.
Poiché la condensazione in fibre di cromatina di 30 nm riduce le
dimensioni della molecola di DNA di circa 40 volte, è necessaria
un ulteriore condensazione
I livelli di ripiegamento successivi alla fibra di 30 nm non sono ancora
stati completamente chiariti.
Implicano la presenza di REGIONI AD ANSA: alcune regioni del DNA si
associano con un impalcatura nucleare costituita da diverse proteine non
ancora completamente identificate (probabilmente H1 e topoisomerasi)
Le proteine si legano al DNA riconoscendo probabilmente sequenze
specifiche che formano il collo di ciascuna ansa
Il tratto di DNA che forma un ansa può contenere un gruppo di geni
correlati tra loro ed è lungo da 20000 e 100000 bp; ciò significa che
un tipico cromosoma umano può contenere circa 2600 regioni ad ansa
ed ogni ansa di cromatina è formata da un tratto della fibra di 30 nm
lungo in media circa 4000 nm
A questo punto la molecola di DNA avrebbe la lunghezza di circa 100
µm; deve quindi ripiegarsi ulteriormente per poter rimanere all interno
del nucleo
Esistono evidenze sperimentali a favore dell esistenza di
ulteriori livelli di organizzazione nei cromosomi eucariotici,
ognuno dei quali determina un aumento esponenziale del grado
di compattezza.
La spiralizzazione del DNA è accompagnata dalla
fosforilazione di tutte le molecole di H1 della cellula a livello
di cinque diverse specifiche molecole di serina.
I progressivi gradi di compattezza variano probabilmente da
cromosoma a cromosoma, da una regione all altra di un singolo
cromosoma, da un istante all altro nella vita della cellula.
Ad oggi, nessun modello è in grado di descrivere
adeguatamente questa struttura.
LA COMPATTEZZA DEL DNA DEL CROMOSOMA
EUCARIOTICO E VEROSIMILMENTE DOVUTA AD
AVVOLGIMENTI SUCCESSIVI CHE SI SOVRAPPONGONO
AD AVVOLGIMENTI GIA PRESENTI
Modello di compattamento del DNA in un
cromosoma eucariotico
Oltre alle proteine istoniche, altre proteine si legano al DNA a livello
di sequenze specifiche (PROTEINE CROMOSOMICHE NON
ISTONICHE)
Sono PROTEINE REGOLATRICI
Regolano il processo di trascrizione, l inizio della sintesi del DNA, il
ripiegamento della molecola in regioni funzionalmente distinte
I nucleosomi limitano l accesso delle proteine regolatrici alle specifiche
sequenze di DNA alle quali esse si legano.
Nel caso di alcune proteine regolatrici, il legame è possibile solo se
quel certo tratto di DNA è completamente privo di nucleosomi o se il
loro sito di legame si trova nel DNA linker.
Questo fa ipotizzare che in alcune regioni della cromatina i nucleosomi
non sono disposti a caso, ma in modo da lasciare relativamente libere
quelle sequenze di DNA che devono essere riconosciute da altre
proteine cellulari.
La disposizione dei nucleosomi a livello di specifiche sequenze è detta
MESSA IN FASE O POSIZIONAMENTO DEI NUCLEOSOMI
L’interazione del DNA con l’ottamero istonico è dinamica
Il nucleosoma può muoversi per permettere l’accesso delle proteine regolatrici alle
loro sequenze di legame in risposta alle diverse richieste di accessibilità
(Rimodellamento del nucleosoma).
Modificazioni delle code N-terminali degli istoni influenzano
l’accessibilità alla cromatina
In che modo le modificazioni degli istoni alterano la funzione dei
cromosomi?
- L’acetilazione o la fosforilazione riducono le cariche positive e quindi l’affinità
delle code istoniche per il DNA
- Modificazioni delle code istoniche (in particolare l’acetilazione) riducono la
capacità del DNA di condensarsi nella fibra di 30 nm.
- Alcune modificazioni favoriscono la formazione di siti di legame per proteine
regolatorie
Le modificazioni degli istoni formano un codice che può essere
letto dalle proteine coinvolte nell’espressione genica o in altre
reazioni a carico del DNA
Quindi…..
le modificazioni a livello delle code istoniche e i
processi di rimodellamento dei nucleosomi lavorano
insieme per determinare l’accessibilità delle diverse
regioni del DNA
Passaggio da:
eterocromatina trascrizionalmente silente
a:
eucromatina trascrizionalmente attiva
La metilazione del DNA avviene per trasferimento di un gruppo metile
da S-adenosilmetionina alla posizione 5 della citidina da parte di
enzimi della famiglia delle DNA metiltrasferasi (DNMT).
Nei mammiferi sono normalmente metilate citidine seguite da guanosine
(CpG).
Possono essere metilate anche citidine seguite da altre basi, in
particolare adenine (CpA).
Nei mammiferi sono metilate tra il 60% ed il 90% delle sequenze CpG.
Sequenze CpG non metilate sono raggruppate in clusters chiamati
”Isole CpG " che sono presenti nelle regioni regolatorie al 5’ di molti
geni.
Gli effetti della metilazione del DNA sull’espressione
genica si esplicano attraverso meccanismi di diverso tipo e
portano generalmente alla riduzione dell’espressione
genica, chiamata
In generale:
silenziamento genico
La metilazione del DNA si inserisce nel contesto
delle modificazioni chimiche delle proteine
istoniche.
I due processi formano un modello di regolazione
flessibile ma preciso che è essenziale per le
attività fisiologiche delle cellule e dei tessuti.


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)







![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)