LEZIONE DEL 06/04/2017
DNA: il responsabile per la riproduzione genetica
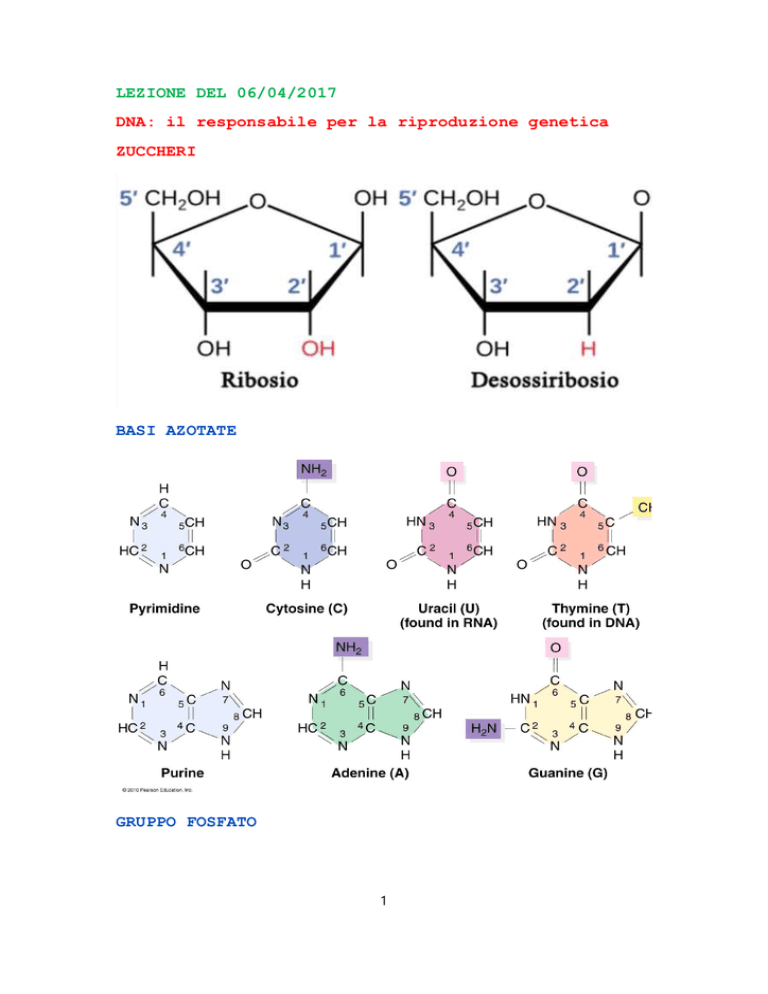
ZUCCHERI
BASI AZOTATE
GRUPPO FOSFATO
1
NUCLEOTIDI DNA
Un nucleotide è l'unità monomerica di una catena di
DNA/RNA (polinucleotidica) formata da zucchero,
gruppo fosfato e base azotata. Tale unità strutturale
si lega ad altre per formare una catena tramite un
legame fosfodiesterico, che implica l'interazione tra
il il C5 dello zucchero di un nucleotide e il C3
dello zucchero del nucleotide soprastante con il
gruppo fosfato in medias res.
NUCLEOTIDI RNA
Anche nell'RNA ci sono la guanina e l'adenina
(purine) e citosina (pirimidina)come basi azotate, ma
al posto della timina (pirimidina) c'è l'uracile.
L'acidità è data dal gruppo fosfato: il pK del gruppo
fosfato è minore di 7.
2
Per quanto riguarda le basi azotate, esse possono
assumere varie forme tautomeriche, che differiscono
nella localizzazione dei legami idrogeno e dei doppi
legami. In genere, i doppi legami coniugati mi
portano all'assorbimento di luce.
ENDONULEASI ED ESONUCLEASI
Affinché il legame fosfodiestereo possa avverire si
deve spendere energia: questo ruolo da "moneta di
scambio energetico" lo compie l'ATP che si trasforma
in AMP durante la formazione del legame
fosfodiesterico.
3
Il processo di per sé è molto lento dal punto di
vista cinetico: servono dei catalizzatori biologici
(enzimi) che lo accelerino: endonucleasi ed
esonucleasi.
Vi riporto una spiegazione breve della funzione dei
due enzimi presa da Sapere.it (mi spiace, ma non
riesco ad astenermi dal congratularmi con la di
Venere per la scoperta di un nuovo antiparallelismo,
oserei dire un chiasmo semantico che attribuisce a
due grecismi prefissali significati opposti agli
usuali [ per lei "endo-" sta per "fuori" ed "eso-"
sta per "dentro" ]) - ho messo di proposito le
parentesi quadre dentro le tonde, tanto quel che di
norma sta fuori, ora sta dentro - :
[ sf. [endo-+nucleo+ -asi]. Enzima che rompe i legami
fosfodiesterici interni di una molecola di DNA o di
RNA; a differenza dell'esonucleasi non scinde gli
acidi nucleici in corrispondenza delle estremità
delle molecole nucleotidi terminali. Le prime
endonucleasi scoperte rompevano i legami interni al
DNA senza particolare specificità di azione; dagli
anni Settanta in poi, invece, sono state individuate
moltissime endonucleasi di origine batterica, cui è
stato dato il nome di enzimi di restrizione, che
svolgono la propria azione solo in corrispondenza di
sequenze nucleotidiche molto specifiche. Tali zone
del DNA hanno come caratteristica comune una
4
simmetria doppia intorno a un punto, in modo che una
stessa sequenza, che prende il nome di sito di
restrizione, sia presente su entrambe le eliche del
DNA. I numerosi enzimi individuati e caratterizzati
in oltre cento specie differenti di batteri, sono
dotati di specificità differente sia per sito di
restrizione, sia per il tipo di taglio che in questa
determinano; possono infatti generare due estremità a
doppio filamento o piccole regioni a singola elica.
Gli enzimi di restrizione, che hanno in natura il
compito di proteggere l'organismo produttore
dall'ingresso di molecole di DNA estraneo,
rappresentano una forma molto primitiva di “sistema
immunitario” e trovano il loro utilizzo in tutte le
tecniche di base della biologia molecolare e
dell'ingegneria genetica: permettono di trasformare
molecole circolari di DNA in molecole lineari dalle
estremità ben definite; una rapida identificazione
indiretta di particolari sequenze; l'isolamento di
piccoli frammenti che possono essere introdotti prima
in vettori, poi in cellule e riprodotti per
clonazione.]
NATURA E SIGNIFICATO DELLA STRUTTURA PRIMARIA
1. Direzionalità di catena polinucleotidica;
2. Individualità di catena polinucleotidica
determinata dalla sequenza di nucleotidi (struttura
primaria).
pApCpGpTACGTT
La sequenza primaria mi dà l'informazione genetica.
DIFFRAZIONE DEI RAGGI X
Si tratta di una tecnica volta ad individuare
elementi ripetuti nella struttura della molecola.
[cito: "DNA essiccato"]
5
MODELLO DI WATSON E CRICK
La struttura secondaria si riferisce alla
conformazione che l'acido nucleico assume come
risultato della struttura primaria. B-DNA, A-DNA, e
Z-DNA sono forme di struttura secondaria. La B-DNA è
la forma che predomina in ambiente acquoso cellulare.
La forma A è favorita in soluzioni povere d'acqua. Il
DNA si organizza in una doppia elica destrorsa più
larga, che contiene 11 residui per ogni giro. La
forma Z è una doppia elica sinistrorsa che consta di
12 residui per ogni giro d'elica.
Il confornto tra strutture tridimensionali di del DNa
mostra che la dorma A è più larga mentre la forma Z
più stretta rispetto alla forma B.
La solubilità in genere è data dalla presenza dello
zucchero nelle unità nucleotidiche.
6
Il modello di Watson e Crick della doppia elica e le
regole di Chargaff, secondo cui A + G = T + C,
suggeriscono che una A è sempre accoppiata ad una T e
una G ad una C. Le basi interagiscono tra loro
mediante legami idrogeno:
- G e C formano tre legami idrogeno;
- A e T formano due legami idrogeno.
Ogni giro di elica contiene 10.5 coppie di basi e una
periodicità di 36 Å (ångström). Le due catene che
costituiscono la doppia elica hanno diversa
7
composizione, in particolare si definiscono:
- complementari, ad ogni A su una catena corrisponde
una T sull'altra catena;
- antiparallele, i legami fosfodiesterei 5' 3'
corrono in direzioni opposte (quindi i legami di
concatenamento nel secondo filamento saranno
fosfodiesterei 3' 5').
REPLICAZIONE DNA (padre -> figlio)
CONSERVATIVA - SEMI-CONSERVATIVA - DISPERSIVA
8
L'enzima che solitamente interagisce con un'elica di
DNA è il DNA polimerasi.
La lunghezza complessiva del DNA genomico di una
cellula umana è di circa 2 m. Un corpo umano adulto
contiene circa 1014 cellule, e quindi la lunghezza
totale corrisponde a circa 2 x 1011 km. La
circonferenza della Terra è pari a 4 x 104 km. La
distanza tra la Terra e il sole equivale a 1.5 x 108
km.
IL SUPERAVVOLGIMENTO DEL DNA
Il DNA è in forma di una doppia elica in cui entrambe
le catene ruotano intorno ad un asse. Un ulteriore
ripiegamento dell'elica genera il superavvolgimento
del DNA. Ivi non si creino superavvolgimenti, l'elica
rimante in uno stato rilassato (centrale in figura).
9
Un cerchio di DNA può formarsi in seguito all'unione
delle estremità di un DNA lineare per un DNA di 260
coppie di basi.
In entrambe le forme, lineare e circolare, i due
filamenti si attraversano 25 volte, il che è chiamato
numero di legame, Lk, e non cambia indentemente da
come sia incurvato l'anello. L'anello non è
superavvolto e viene chiamato rilassato. Il
superavvolgimento è indotto se il DNA viene svolto
prima che si formi l'anello come mostrato sopra.
C'è bisogno di aggiungere due ulteriori termini
topologici per descrivere la nostra molecola di DNA
superavvolta. Uno è la torsione, Tw, l'altro il
contorcimento, Wr. La torsione è il numero di giri
che un filamento compie attorno all'asse dell'elica.
Per il B-DNA la torsione è data dal numero di coppie
di basi fratto 10.4 (numero di coppie di basi per
giro della doppia elica B-DNA). Per un DNA da 260
coppie Tw = 25. Il contorcimento è il numero di volte
che l'elica si passa sopra ed è una misura del
svravvolgimento. Un'elica da 260 coppie compie due
attraversamenti destrorsisu se stessa per cui Wr.
Poiché Lk è costante per ogni anello, per ogni
torsione della doppia elica aggiunta, DTw, deve
esserci un uguale ed opposta torsione di
superavvolgimento, -DWr. Il valore solito di Wr è
negativo, il che vuol dire che il superavvolgimento è
destrorso. Lo stato topologico di un DNA è descritto
dall'equazione Lk = Tw + Wr. Per un DNA da 260 coppie
Lk = 23 = 25 + (-2). Cambiando uno dei tre termini
topologici, gli altri due devono cambiare: dLk = DTw
10
+ DWr.
Gli enzimi che determinano l'aumento o la diminuzione
del grado di disavvolgimento del DNA sono chiamati
topoisomerasi, e la proprietà su cui agiscono è il
numero di legame. Le topoisomerasi hanno un ruolo
determinante in processi quali la replicazione e
l'impacchettamento del DNA. Ne abbiamo due classi:
- le topoisomerasi di tipo I agiscono rompendo
transitoriamente una delle catene del DNA, ruotando
un'estremità intorno alla catena integra e riunendo
le estremità interrotte. Modificano Lk con incrementi
o riduzioni di 1;
- le topoisomerasi di tipo II rompono entrambe le
catene del DNA e modificano la struttura con
intrementi o riduzioni di 2.
11



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)




