Prof. Giorgio Sartor
Metabolismo dei
nucleotidi
1:
2:
3:
4:
5:
6:
7:
introduzione
biosintesi de-novo delle purine
biosintesi de-novo delle pirimidine
catabolismo delle purine
catabolismo delle pirimidine
sintesi deossiribonucleotidi
regolazione della sintesi deossiribonucleotidi
Copyright
© 2001-2013
by Giorgio Sartor.
V.0.2 © gsartor
2001-2013
N01 - Versione 0.2 – may 2013
Metabolismo dei nucleotidi
-1
All rights reserved.
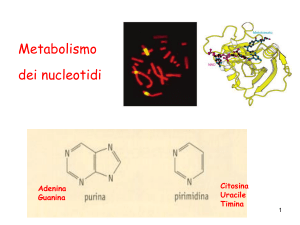
BIOSINTESI delle PIRIMIDINE
NH2
O
N
HO
O
O
P
O
O
HN
N
HO
O
HO
OH
CTP
V.0.2 © gsartor 2001-2013
O
O
O
P
O
O
HO
CH3
HN
N
HO
O
OH
UTP
Metabolismo dei nucleotidi
O
O
P
O
O
HO
N
O
H
dTTP
-2
1
Pirimidine
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
-3
Biosintesi de-novo delle pirimidine
• Contrariamente alle PURINE, le
PIRIMIDINE vengono sintetizzate
come anello purinico e quindi legate
al riboso-5-P;
• I precursori sono:
– Carbamilfosfato
– Aspartato
• Si forma il DIIDROOROTATO
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
-4
2
Biosintesi de-novo delle pirimidine
• Sei reazioni per sintetizzare
UMP;
• Sei substrati:
–
–
–
–
–
• Sei enzimi:
1. Carbamilfosfato sintasi II (EC
6.3.5.5)
2. Aspartato transcarbamilasi (EC
2.1.3.2)
3. Diidrorotasi (EC 3.5.2.3)
4. Diidrorotato deidrogenasi (EC
1.3.1.14)
5. Orotato fosforibosiltranferasi (EC
2.4.2.10)
6. OMP decarbossilasi (EC 4.1.1.23)
HCO3ATP
Due aminoacidi (Gln, Asp)
Ribosio (αPRPP)
NADP+ (CoQ, fumarato)
• Sei prodotti/intermedi:
1. Carbamilfosfato
2. Carbamilaspartato
3. Diidrorotato
4. Orotato
5. Orotinina-5’-monofosfato
(OMP)
6. UMP
V.0.2 © gsartor 2001-2013
• Nei procarioti i sei enzimi sono
separati;
• Negli eucarioti sono presenti in tre
proteine:
– CAD
– Diidrorotato deidrogenasi
– Uridina monofosfato sintasi
Metabolismo dei nucleotidi
-5
Biosintesi de-novo delle pirimidine
Gln + CO2 + 2 ATP
O
4
H
Carbamilfosfato
3
N
H
2
O
O
O
P
O
5
O
H
O
4
3
2
O
6
O
N1
H
O
Aspartato
5
N
O
N1
H
O
H
6
O
DIIDROROTATO
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
-6
3
Biosintesi de-novo delle pirimidine
• Nel citosol degli eucarioti i tre enzimi che
catalizzano le tre reazioni per la sintesi del
diidroorotato da Gln, CO2 e Asp:
– Carbamilfosfato sintasi II (EC 6.3.5.5) (CPSII)
– Aspartato transcarbamilasi (EC 2.1.3.2)
(ATCasi)
– Diidrorotasi (EC 3.5.2.3)
• sono presenti in unico peptide di 210 kD
codificato da un unico gene (CAD).
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
-7
Biosintesi de-novo delle pirimidine
O
Gln
O
H
Bicarbonato ATP
O
O
1
Carbamilfosfato
O
ATP
NH2
O
HO
O
N
H
ADP
P
O
ADP
O
P
O
OH
O
1
O
O
NH2
O
O
Pi
Carbonil-fosfato
O
O
O
O
OH
O
Glu
NH2
O
2
H 2N
Pi
O
O
QH2
Q
N
H
O
Orotato
V.0.2 © gsartor 2001-2013
H
O
H
4
O
O
H
OH
N
N
O
O
H2O
H
H
HN
O
N
HH
O
Diidroorotato
Metabolismo dei nucleotidi
3
O
O
N
H
O
Carbamilaspartato
-8
4
Biosintesi de-novo delle pirimidine
O
P
O
O
O
O
O
αPRPP
O
HO
O
P
OH
P
O
OO
O
Orotato
OH
HN
5
O
PPi
O
N
H
O
O
O
HN
O
O
P
O
O
O
HN
O
O
O
N
O
P
O
O
O
O
N
O
6
HO
CO2
OH
HO
Orotidil-5'-monofosfato
OMP
V.0.2 © gsartor 2001-2013
OH
Uridin-5'-monofosfato
UMP
Metabolismo dei nucleotidi
-9
CAD
O
Gln
O
H
Bicarbonato ATP
O
O
1
Carbamilfosfato
O
ATP
NH2
O
HO
O
N
H
ADP
P
O
ADP
O
P
O
OH
O
1
O
O
NH2
O
O
Pi
Carbonil-fosfato
O
O
O
O
OH
O
Glu
NH2
O
2
H 2N
Pi
O
O
QH2
Q
N
H
O
Orotato
V.0.2 © gsartor 2001-2013
H
O
H
4
O
O
H
OH
N
N
O
O
H2O
H
H
HN
O
N
HH
O
Diidroorotato
Metabolismo dei nucleotidi
3
O
O
N
H
O
Carbamilaspartato
- 10
5
1. Carbamilfosfato sintasi
• La sintesi del carbamilfosfato è catalizzata dalla
Carbamilfosfato sintasi che è diversa negli
eucarioti e nei procarioti:
• Eucarioti:
– Carbamilfosfato sintasi II (EC 6.3.5.5) (CPSII) con
diverso substrato (Gln) rispetto all’enzima mitocondriale
(CPS-I) (substrato NH3 nel ciclo dell’urea) con analogo
meccanismo;
– Il carbamilfosfato citosolico è inviato obbligatoriamente
nella sintesi di PIRIMIDINE;
– Il carbamilfosfato mitocondriale (ciclo dell’urea) va a
sintetizzare arginina via ornitina e citrullina.
• Procarioti
– Unico enzima Carbamilfosfato sintasi che ha come
substrato sia NH3 che Gln
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 11
1. Carbamilfosfato sintasi II (EC 6.3.5.5) (CPSII)
O
O
H
ATP
O
HO
H
ADP
ATP
NH2
P
O
ADP
O
O
OH
P
O
NH2
O
O
O
Bicarbonato
Carbamilfosfato
O
O
O
O
N
Pi
Carbonil-fosfato
O
O
O
O
O
NH2
• Enzima citosolico
• L’attività di CPSII è presente in un unico peptide da 210kD
che presenta anche l’attività ATCasica e della Diidroorotasi;
• Due molecole di ATP:
– la prima attiva il bicarbonato (CO2) per formare il carbonil-fosfato,
• Il gruppo amminico della Gln si lega e forma il carbammato
liberando il fosfato e Glu,
– La seconda molecola di ATP lega il carbammato attivandolo.
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 12
6
Carbamilfosfato sintasi I (EC 6.3.4.16) (CPSI)
H
H
ATP
H
ADP
O
N
ATP
O
HO
O
O
O
P
O
O
P
O
O
O
NH2
O
O
O
Bicarbonato
ADP
NH2
OH
O
Pi
Carbonil-fosfato
Carbammato
Carbamilfosfato
• Enzima mitocondriale (ciclo dell’urea) e nei procarioti
• Due molecole di ATP :
– la prima attiva in carbonato (CO2) per formare il carbonilfosfato,
– L’ammoniaca si lega e forma il carbammato liberando il
fosfato,
– La seconda molecola di ATP lega il carbammato
attivandolo.
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 13
Ciclo dell’urea
2ATP
2ADP + Pi
Carbamilfosfato
O
CO2 + NH 3
O
Carbamil-fosfato
sintasi
EC 6.3.4.16
O
P
O
Citrullina
(IN)
NH 2
NH 2
HN
O
O
NH3 +
Ornitina
(IN)
Ornitina
trans carbamilasi
EC 2.1.3.3
NH3 +
Trasportatore
della citrullina
NH3 +
O
O
O
O
NH3 +
O
2 Pi
H2N
ATP
Argininosuccinato
sintasi
EC 6.3.4.5
Ossalacetato
Ornitina
(OUT)
NH3+
O
O
NH 2
O
α-chetoacido Aminoacido
Trasportatore
dell'ornitina
NH3+
H
N
O
Citrullina
(OUT)
N
O
H2N
Aspartato
O
Urea
Malato
O
NH3+
O
H2 N
O
Arginasi
EC 3.5.3.1
H
N
O
O
N
OH
OH
P
O
O
NH2+
O
NH3 +
Citrullil-AMP
O
H2O
N
N
PPi
O
Fumarato
NH3 +
O
O
O
H
N
NH 2
NH 2+
Arginina
Argininosuccinato
sintasi
EC 6.3.4.5
O
AMP
O
O
O
O
NH3+
Argininosuccinato
liasi
EC 4.3.2.1
O
H
N
H
N
O
O
NH2+
O
Argininosuccinato
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 14
7
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
O
Gln
O
H
Bicarbonato ATP
H
ADP
O
N
Carbamilfosfato
O
ATP
NH2
O
ADP
P O
• HONegli eucarioti Ol’attività
OH ATCasica è
O
O
presente in un unicoO peptide da O
O
Pi
Carbonil-fosfato
210kD che presenta
anche l’attività
di
O
CPSII e diidroorotasica;
Glu NH
• Metallo enzima (Zn++)
• È codificato da un singolo gene
O
QH2
O
Q
HN
O
OH
2
O
OH
O
O
N
H
O
O
Diidroorotato
V.0.2 © gsartor 2001-2013
H
N
N
HH
O
Orotato
H2N
O
H
O
O
O
Pi
N
N
H
O
O
H
O
O
NH2
H2O
H
H
O
O
2
O
P
O
Carbamilaspartato
Metabolismo dei nucleotidi
- 15
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
• Diversa regolazione nei
procarioti
O
Gln
O
H
rispetto agli eucarioti
N
Bicarbonato ATP
ADP (E.
‒ Nei procarioti
O
HO
‒
‒O
‒
‒
Carbamilfosfato
O
H
ATP
ADP
Coli):
NH
O
è attivata daOATP
da CTP;
P O e inibita
OH
O
ATP e CTP competono
per lo stesso
sito;
O
O
O
Pi
regola la conversione
di carbamilfosfato
Carbonil-fosfato
O
in carbamilaspartato;
Glu NH
controlla la sintesi di arginina e dei
nucleotidi.
QH2
O
Q
H
N
H
O
Orotato
O
OH
O
O
2
H2N
Pi
O
O
H
H
OH
N
O
O
NH2
O
N
O
O
O
O
H2O
H
H
HN
P
O
2
O
V.0.2 © gsartor 2001-2013
O
2
N
HH
O
Diidroorotato
Metabolismo dei nucleotidi
O
O
N
H
O
Carbamilaspartato
- 16
8
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
O
Gln
O
H
Bicarbonato ATP
N
H
ADP
O
O
O
ATP
NH2
O
HO
Carbamilfosfato
O
P
O
ADP
O
P
O
OH
O
NH2
O
O
O
O
Pi
Carbonil-fosfato
O
O
O
O
OH
O
Glu
NH2
O
2
H2N
Pi
O
QH2
O
Q
HN
H2O
H
H
H
N
H
O
H
OH
N
O
O
Orotato
V.0.2 © gsartor 2001-2013
O
H
N
O
O
O
N
HH
O
Diidroorotato
O
O
N
H
O
Carbamilaspartato
Metabolismo dei nucleotidi
- 17
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 18
9
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
O
Gln
O
H
Bicarbonato ATP
N
H
ADP
O
P
O
O
ATP
NH2
O
HO
Carbamilfosfato
O
O
ADP
O
P
O
OH
O
NH2
O
O
O
O
Pi
Carbonil-fosfato
O
O
O
O
OH
O
Glu
NH2
O
2
H2N
Pi
O
QH2
O
Q
HN
H2O
H
H
H
O
H
O
N
H
O
O
Orotato
N
HH
O
Diidroorotato
V.0.2 © gsartor 2001-2013
H
OH
N
N
O
O
O
O
O
N
H
O
Carbamilaspartato
Metabolismo dei nucleotidi
- 19
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
•
•
Il sito catalitico è localizzato
all’interfaccia tra due catene catalitiche
nello stesso trimero;
Il legame dei substrati è stato studiato
utilizzando un inibitore la cui struttura
si suppone simile allo stato di
transizione:
‒ N-(fosfonoacetil)-L-aspartato
(PALA)
O
O
O
O
O
O
P
N
H
O
O
O
O
O
O
O
NH2
P
O
V.0.2 © gsartor 2001-2013
O
O
O
NH2
Metabolismo dei nucleotidi
- 20
10
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
O
Bicarbonato O
ATP
O
N
H
O
O
O
ATP
NH2
P
O
N
H
Carbamilfosfato
O
O
O
P
O
H
ADP
O
HO
Gln
O
O
O
ADP
OH
O
O
P
O
NH2
O
O
O
Pi
Carbonil-fosfato
N-(fosfonoacetil)-L-aspartato (PALA)
O
O
O
O
OH
O
Glu
NH2
O
2
H2N
Pi
O
O
O
O
O
O
O
O
O
O
O
QH2
Q
H2O
H
H
H
NH2
HN
P
O
O
O
N
O
Orotato
N
HH
O
O
N
H
O
O
Diidroorotato
V.0.2 © gsartor 2001-2013
OH
N
O
H
NH
2O
H
N
O
O
O
H
Carbamilaspartato
Metabolismo dei nucleotidi
- 21
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
O
O
O
O
O
O
Effettori
allosterici
P
N
H
O
O
nH
(coefficiente di
Hill)
KD(PALA)
(nM)
Nessuno
2.0
110
ATP
1.4
65
CTP
2.3
270
N-(fosfonoacetil)-L-aspartato (PALA)
O
O
O
O
O
O
NH2
P
O
O
V.0.2 © gsartor 2001-2013
O
NH2
Metabolismo dei nucleotidi
- 22
11
2. Aspartato trans carbamilasi (EC 2.1.3.2) (ATCasi)
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 23
3. Diidroorotasi (EC 3.5.2.3)
O
Gln
O
H
Bicarbonato ATP
N
H
ADP
Carbamilfosfato
O
ATP
NH2
ADP
O
• L’attività della diidrooratasi è presente, negli
eucarioti, in un unico peptide da 210kD che
presenta anche Carbonil-fosfato
l’attività di CPSII e ATCasica;
O
O
HO
O
O
P
O
O
OH
O
O
Pi
O
P
O
O
HO
OH
O
OH
P
P
O
O
O
O
O
QH2
O
O
O
HN
O
O
N
O
HO
OH
O
H2N
N
H
H
Orotato
O
H
H
OH
N
N
O
O
O
H2O
H
O
O
O
P
O
Q
H
O
O
O
Pi
HN
PPi
O
NH2
αPRPP
O
NH2
O
Glu
O
O
O
O
O
P
O
O
N
HH
O
Diidroorotato
3
O
O
N
H
O
Carbamilaspartato
O
OH
V.0.2 © gsartor 2001-2013
Orotidil-5'-monofosfato
OMP
Metabolismo dei nucleotidi
- 24
12
4. Diidrorotato deidrogenasi (EC 1.3.1.14)
O
Gln
O
H
Bicarbonato ATP
N
H
ADP
O
P
O
1
O
ATP
NH2
O
HO
Carbamilfosfato
O
O
ADP
O
P
O
OH
O
1
O
O
NH2
O
O
Pi
Carbonil-fosfato
O
O
O
O
OH
O
Glu
NH2
O
2
H 2N
Pi
O
O
QH2
Q
HN
H
O
H
H
O
4
N
H
O
N
HH
O
Orotato
3
O
O
N
H
O
O
Diidroorotato
V.0.2 © gsartor 2001-2013
OH
N
N
O
O
H2O
H
H
O
Carbamilaspartato
Metabolismo dei nucleotidi
- 25
4. Diidrorotato deidrogenasi (EC 1.3.1.14)
•
•
Lega FMN, FAD e un cluster [2Fe-2S].
L’enzima consiste in due subunità;
O
Gln
‒ Una subunità catalica che
legaO FMN
H
Carbamilfosfato
N al trasferimento
‒ Una subunità deputata
di elettroni
che
O
H
Bicarbonato ATP
ADP
ATP
ADP
NH
O
contiene
FAD
e
un
centro
[2Fe-2S].
O
O
P O
NH
P O
O
OH
• LaHOreazione avvieneOnel
citosol
ed è la sola reazione
redox
nella
O
O
O
O
biosintesi de-novo dei nucleotidi
pirimidinici.
O
O
O
O
Pi
+
• Un altro enzima di Carbonil-fosfato
classe 1 usa fumarato
(EC 1.3.98.1)
o NADP
O
OH
O
O
(EC 1.3.1.15) come accettore di elettroni
O
Glu NH
H N
P
αPRPP
O •
O Un altro enzima di classe 2 legato alla membrana
Pi mitocondriale
O
O
O
OH
(ECO 1.3.5.2)
usa
il chinone come accettore di elettroni.
P
P
2
2
2
2
O
HO
OH
O
O
O
O
QH2
NADPH + H+
O
4
HN
O
O
O
HN
O
O
O
N
O
HO
N
H
H2O
H
H
Orotato
O
H
H
OH
N
N
O
O
O
O
P
O
H
O
PPi
O
Q
NADP+
N
HH
O
Diidroorotato
O
O
N
H
O
Carbamilaspartato
O
OH
V.0.2 © gsartor 2001-2013
Orotidil-5'-monofosfato
OMP
Metabolismo dei nucleotidi
- 26
13
4. Diidrorotato deidrogenasi (EC 1.3.1.14)
• Subunità catalitica:
– Orotato
– FMN
– Inibitore
3ZWS
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 27
Biosintesi de-novo delle pirimidine
O
P
O
O
O
O
αPRPP
O
O
HO
O
P
OH
P
O
OO
O
OH
Orotato
HN
5
O
PPi
O
N
H
O
O
O
HN
O
O
P
O
O
O
HN
O
O
O
N
O
O
O
P
O
O
N
O
6
HO
CO2
OH
Orotidil-5'-monofosfato
OMP
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
HO
OH
Uridin-5'-monofosfato
UMP
- 28
14
Biosintesi de-novo delle pirimidine
O
P
O
O
O
O
αPRPP
O
O
HO
O
P
OH
P
O
OO
O
OH
Orotato
HN
5
O
PPi
O
N
H
O
O
O
HN
O
O
P
O
O
O
HN
O
O
O
N
O
O
O
P
O
O
N
O
6
HO
CO2
OH
Orotidil-5'-monofosfato
OMP
V.0.2 © gsartor 2001-2013
HO
OH
Uridin-5'-monofosfato
UMP
Metabolismo dei nucleotidi
- 29
UMP sintasi
Orotato fosforibosiltranferasi (EC 2.4.2.10)
OMP decarbossilasi (EC 4.1.1.23)
•
•
•
Negli mammiferi un unico gene codifica per
l’enzima UMP sintasi (UMPS) che catalizza le ultime
due reazioni nella sintesi de-novo delle pirimidine
L’attività Orotato fosforibosiltranferasi (OPRT) è
localizzata nella porzione N-terminale mentre l’
OMP decarbossilasi (OMPDC) è presente al Cterminale
Anche il protozoo Leishmania (e specie correlate)
esprime un unico enzima (3QW4) ma con i domini
invertiti rispetto alla UMPS umana.
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 30
15
UMP sintasi di Leishmania
Orotato fosforibosiltranferasi (EC 2.4.2.10)
OMP decarbossilasi (EC 4.1.1.23)
3QW4
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 31
5. Orotato fosforibosiltranferasi (EC 2.4.2.10)
αPRPP
Orotato
OMP
1LH0
2WNS
• Dominio N-terminale di UMPS che
possiede l’attività OPRT
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 32
16
6. OMP decarbossilasi (EC 4.1.1.23)
UMP
• Dominio C-terminale di
UMPS con attività OMPDC:
– Studi strutturali su OMPDC
da microrganismi ha portato
all’ipotesi che la
decarbossilazione avvenga
attraverso la formazione di
intermedi non covalenti ad
alta energia;
– Dominio privo di cofattori
estremamente efficiente.
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
2QCD
- 33
Uridin-5'-monofosfato (UMP)
O
HN
O
O
P
O
O
N
O
HO
V.0.2 © gsartor 2001-2013
O
OH
Metabolismo dei nucleotidi
- 34
17
UTP
• A cosa serve:
– Precursore dei nucleotidi e dei
deossinucleotidi pirimidinici
– Coinvolto nel metabolismo glucidico:
• Lega il glucosio-1-fosfato per formare UDPG
– Glicogenosintesi
– Metabolismo del galattoso
– Ossidazione del glucoso a acido glucuronico …
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 35
Glicogenosintesi
• Il glucoso uridin-difosfato (UDPG) è il precursore per la sintesi di
glicogeno.
• Un residuo di glucoso è addizionato al glicogeno e viene rilasciato
un UDP.
• Gli zuccheri nucleotidi difosfati sono i precursori della sintesi di
carboidrati complessi, glicoproteine ecc.
• Viene sintetizzato da glucoso-1-fosfato e UTP ad opera della UDPGlucoso pirofosforilasi (EC 2.7.7.9).
O
O
O
O
P
P
O
O
O
O
P
O
HO
N
OH
O
+
NH
O
HO
O
O
O
P
HO
O
O
HO
O
O
OH
UDP-Glucoso pirofosforilasi
EC 2.7.7.9
O
P
O
O
P
O
O
O
+
O
O
HO
O
O
P
HO
O
HO
O
O
P
O
N
NH
OH
HO
V.0.2 © gsartor 2001-2013
O
O
O
Metabolismo dei nucleotidi
OH
O
- 36
18
Glicogenosintesi
• Il processo viene ripetuto fino a che si forma una corta catena di
glucoso (fino a cinque residui).
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 37
Glicogenosintesi
• Il processo viene ripetuto fino a che si forma una corta catena di
glucoso (fino a cinque residui).
OH HO
HO
O
HO
O
P
HO
O
HO
O
O
P
O
O
O
HO
O
O
O
N
R
+
NH
O
OH
HO
OH
O
O
N
H
Glicogenina
EC 2.4.1.186
x4
R
HO
R
HO
O
O
P
O
+
O
O
P
O
HO
V.0.2 © gsartor 2001-2013
O
O
O
N
OH
H
N
O
HO
O
HO
O
O
R
OH
HO
NH
O
OH
4
O
Metabolismo dei nucleotidi
- 38
19
Metabolismo del galattoso
Glicogeno
(n+1 unità)
Glicogeno
(n unità)
UDP
CH2 OH
CH2OH
O
O
OH
HO
Galattoso
ATP
O
ADP
O
HO
O
HO
EC 2.7.1.6
Galattochinasi
O
O
O
O
HO
OH
HO
OH
OH
n+1
O
P
HO
OH
OH
n
OH
O
OH
HO
HO
Galattoso-1-fosfato
HO
OH
O
P
O
O
O
HO
O
P
N
NH
O
O
HO
OH
UDP-glucoso
O
OH
OH
EC 2.7.7.10
Galattoso-1-fosfato
uridililtransferasi
EC 5.1.3.2
UDP-galattoso4-epimerasi
O
HN
O
O
P
O
OH
EC 5.4.2.2
Fosfoglucomutasi
O
O
HO
O
O
O
O
O
OH
P
O
HO
OH
OH
O
HO
O
HO
OH
O
P
HO
O
O
P
O
O
N
O
O
UDP-galattoso
OH
OH
HO
OH
Glucoso-1-fosfato
Glucoso-6-fosfato
GLICOLISI
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 39
Glucuronazione
• Coniugazione di xenobiotici con un H
labile con acido glucuronico da acido
uridindifosfoglicerico (UDPGA)
O
O
O
H
O
O
OH
O
O
HO
HO
OH
OH
O
O
O P O P O
O
HO
N
O
O
OH
UDPGA
V.0.2 © gsartor 2001-2013
O
N
Metabolismo dei nucleotidi
HO
OH
- 40
20
Formazione di UDPG
O
O
O
O P O P O P O
O
O
O
O
O
N
HO
NH
HO
+
HO
O
OH
O
O P O
O
OH
O
OH
UTP
EC 2.7.7.9
UTP-glucoso-1-fosfato
uridililtransferasi
O
O
O
O P O P OH + HO
O
O
HO
OH
V.0.2 © gsartor 2001-2013
O
O
O P O P O
O
O
HO
OH
UDPG
O
O
N
OH
NH
O
Metabolismo dei nucleotidi
- 41
Coniugazione con acido glucuronico
O
O
HN
O
O
O
O P O P O
O
O
OH
O
HO
HO
O
O
HO
N
+
R-OH
OH
EC 2.4.1.17
UDPglucuronato
β-D-glucuronosiltransferasi
O
O
O
O P O P O
O
O
V.0.2 © gsartor 2001-2013
O
HO
O
N
OH
NH
O
O
+
O
Metabolismo dei nucleotidi
HO
O
R
OH
OH
- 42
21
UTP e CTP
O
O
HN
O
O
O
P
O
O
ADP
ATP
P
O
ADP
ATP
O
O
O
O
O
O
O
O P
O
N
EC 2.7.4.4
Nucleoside-fosfato chinasi
O
O
N
O
EC 2.7.4.6
Nucleoside-difosfato chinasi
HO
OH
UMP
P
O
O
HN
O
O
P
O
P
O
HN
O
O
N
HO
O
O
HO
OH
UDP
OH
UTP
ATP
O
O
O
P
NH2
O
O P
O
Nei batteri la CTP sintasi
usa NH4+ come sorgente
di azoto
O
O
ADP
N
O
O
•
EC 6.3.4.2
CTP sintasi
P
O
O
H2N
N
O
O
NH2
O
Glu
OH
O
Gln
O
O
HO
O
NH2
CTP
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 43
Regolazione
HCO3- + Gln + 2ATP
EC 6.3.5.5
Carbamilfosfato Sintetasi
EC 6.3.5.5
Carbamilfosfato Sintetasi II
CP
EC 2.1.3.2
Aspartato trans carbamilasi
Arg
Carbamilaspartato
Orotato
α-PRPP
Procarioti
OMP
Eucarioti
UMP
UDP
UTP
V.0.2 © gsartor 2001-2013
CTP
Metabolismo dei nucleotidi
- 44
22
Regolazione
• CPS II è l’enzima chiave che controlla la
sintesi di pirimidine nei mammiferi ed è
sensibile all’inibizione da UMP,
• È stato dimostrato che la fosforilazione in
vitro di CPS II da parte delle MAP chinasi
abolisce l’inibizione da UMP, in vivo questo
tipo di controllo è innescato da EGF.
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 45
Regolazione
HCO3- + Gln + 2ATP
EC 6.3.5.5
Carbamilfosfato Sintetasi
EC 6.3.5.5
Carbamilfosfato Sintetasi II
CP
PO3--
EC 2.1.3.2
Aspartato trans carbamilasi
Arg
Carbamilaspartato
Orotato
α-PRPP
Procarioti
OMP
Eucarioti
UMP
UDP
UTP
V.0.2 © gsartor 2001-2013
CTP
Metabolismo dei nucleotidi
- 46
23
Interconversione delle pirimidine
NH2
O
O
N
HN
O
O
O
O
O
O
O
P
P
P
O
O
O
O
O
HO
H
NH2
O
O
O
O
O
O
O
O
P
P
P
O
O
O
O
N
O
HO
O
Riduzione
O
OH
N
Riduzione
O
O
O
O
O
O
O
P
P
P
O
O
O
OH
Trasferimento
NH2
O
O
O
O
O
O
O
P
P
P
O
O
O
O
N
O
Metilazione
H
NTP
dCTP
CTP
UTP
dUTP
dTTP
NDP
dCDP
CDP
UDP
dUDP
dTDP
NMP
dCMP
CMP
UMP
dUMP
dTMP
Nucleoside
dCitidina
Citidina
Citosina
H
N
O
HO
O
O
O
O
O
O
P
P
P
O
O
O
O
HO
HN
N
O
HO
BASE
CH3
HN
N
Uridina
dTimidina
Uracile
Timina
Traut TW. Enzymes of nucleotide metabolism: the significance of subunit size and
polymer size for biological function and regulatory properties. CRC Crit Rev Biochem. 1988;23(2):121-69.
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 47
Crediti e autorizzazioni all’utilizzo
• Questo materiale è stato assemblato da informazioni raccolte dai seguenti testi di Biochimica:
– CHAMPE Pamela , HARVEY Richard , FERRIER Denise R. LE BASI DELLA BIOCHIMICA [ISBN 9788808-17030-9] – Zanichelli
– NELSON David L. , COX Michael M. I PRINCIPI DI BIOCHIMICA DI LEHNINGER - Zanichelli
– GARRETT Reginald H., GRISHAM Charles M. BIOCHIMICA con aspetti molecolari della Biologia
cellulare - PICCIN
– VOET Donald , VOET Judith G , PRATT Charlotte W FONDAMENTI DI BIOCHIMICA [ISBN 9788808-06879-8] – Zanichelli
• E dalla
–
–
–
–
consultazione di svariate risorse in rete, tra le quali:
Kegg: Kyoto Encyclopedia of Genes and Genomes http://www.genome.ad.jp/kegg/
Brenda: http://www.brenda.uni-koeln.de/
Protein Data Bank: http://www.rcsb.org/pdb/
Rensselaer Polytechnic Institute:
http://www.rpi.edu/dept/bcbp/molbiochem/MBWeb/mb1/MB1index.html
Questo ed altro materiale può essere reperito a partire da:
http://www.ambra.unibo.it/giorgio.sartor/ oppure da http://www. gsartor.org/
Il materiale di questa presentazione è di libero uso per didattica e ricerca e può essere usato senza
limitazione, purché venga riconosciuto l’autore usando questa frase:
Materiale ottenuto dal Prof. Giorgio Sartor
Università di Bologna – Alma Mater
Giorgio Sartor - [email protected]
V.0.2 © gsartor 2001-2013
Metabolismo dei nucleotidi
- 48
24