SDS-PAGE
• La tecnica elettroforetica maggiormente adoperata per le molecole
proteiche è l’elettroforesi su gel di poliacrilammide in sodio
dodecil solfato (SDS-PAGE)
• L’SDS-PAGE consente la separazione di molecole con un rapporto
carica/massa identico, ma di dimensioni molecolari diverse
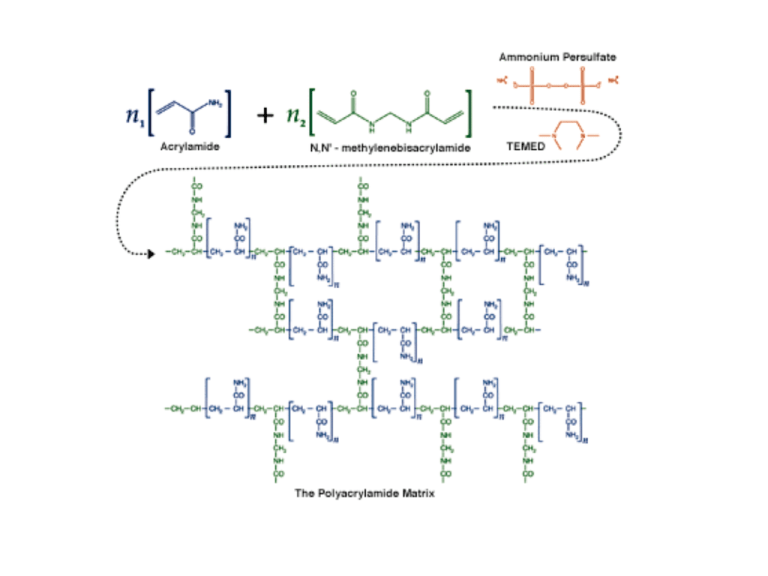
• Nell’SDS-PAGE la matrice del gel è una sostanza reticolata che agisce
come un setaccio, in cui le forze di attrito fanno diminuire la mobilità
elettroforetica delle molecole in relazione alle loro dimensioni
Sodio Dodecil Solfato
CH3(CH2)10CH2OSO3- Na+
proteina
Nella SDS-PAGE la velocità di migrazione è inversamente
proporzionale alle dimensioni della proteina
(il rapporto CARICA/MASSA è uguale per tutte le proteine)
-
+
GEL IN GRADIENTE
Gel di poliacrilammide nel quale la percentuale di acrilammide varia in
modo uniforme dal 5% (alla sommità del gel) al 25% (sul fondo dello
stesso)
Il gradiente è formato con un formatore di gradiente
E’ possibile separare proteine con un intervallo di massa molecolare
relativa molto più ampio rispetto ad un gel a porosità costante
Proteine con masse molecolari relative simili possono essere separate
TECNICHE DI RIVELAZIONE
Dopo aver tolto la corrente, il gel è rimosso dalle due lastre di vetro e
messo a colorare , in agitazione, in una soluzione colorante
COOMASSIE BRILLIANT BLUE R-250
Le bande proteiche sono fissate e il gel è immerso in una
soluzione che contiene acido acetico e Coomassie
Brilliant Blue R-250 sciolto in metanolo
Per ottenere bande proteiche omogenee si colora il gel
fino a quando non è uniformemente blu (~ 30’ r.t.)
successivamente si rimuove il colorante in eccesso
immergendo il gel in una soluzione di acido acetico e
etanolo
Colorazione delle proteine con nitrato d’argento
Il gel è immerso in una soluzione diluita di metanolo/acido
acetico per fissare le proteine al gel
Il gel è incubato con una soluzione acida di nitrato di argento
che reagisce con le proteine
L’immagine si forma per riduzione dello ione argento alla sua
forma metallica per opera della formaldeide a pH
alcalino (sodio carbonato)
Lo sviluppo è fermato acidificando la soluzione con acido acetico
Il metodo è da 10 a 100 volte più sensibile della
colorazione con Coomassie
SDS-PAGE fissato con acido acetico
e colorato con BLU COOMASSIE
Applicazioni dell’SDS-PAGE
Analisi quantitativa delle proteine presenti
nella frazione di interesse
Identificazione del P.M. di proteine
sconosciute
Analisi qualitativa delle proteine presenti
nella frazione di interesse (colorazione con
Blu Coomassie o western blotting)
10 μg di proteine totali del campione da:
Proteina di interesse
Analisi qualitativa
di un SDS-PAGE
1.
2.
3.
4.
5.
STD
Omogenato 5 µl
I purif. 10 µl
II purif. 15 µl
III purif. 20 µl
Western blot
Dopo la corsa elettroforetica le proteine possono essere
trasferite su un supporto più stabile e “colorate” usando
anticorpi specifici
Il western blot è circa 100-200 volte più sensibile della
colorazione per Blu Coomassie
Il gel viene sottoposto ad una carica
elettrica “trasversale” che forza le
proteine a migrare fuori dal gel, fino
ad arrivare sulla membrana a cui
aderiscono (nitrocellulosa)
1.
2.
3.
4.
Il filtro viene saturato con BSA o “fat free
milk” poiché sulla membrana rimangono
dei siti liberi
La membrana viene immersa in una
soluzione che contiene l’Ab specifico per
la proteina di interesse
Un secondo Ab, che riconosce la fonte
biologica del I, lega un enzima che
catalizza una reazione cromogena
La reazione con il S dà luogo ad un P
luminescente che può essere rivelato per
autoradiografia (5)
•fosfatasi alcalina converte il 5-bromo-4-cloro-indolofosfato (BCIP),
incolore, in un prodotto di colore blu
•perossidasi di rafano utilizza acqua ossigenata come substrato e
ossida il 3-ammino-9-etilcarbazolo in un prodotto insolubile
marrone o il 4-cloro-1-naftolo in un prodotto insolubile blu
Enhanced chemiluminescence: in presenza di acqua ossigenata e di
luminolo, un substrato chemiluminescente, la perossidasi ossida il
luminolo con concomitante produzione di luce, la cui intensità
aumenta di 1.000 volte in presenza di un intensificatore
chimico. La luce emessa può essere rilevata esponendo il blot ad una
lastra fotografica
LogPM (Da)
Identificazione del PM di una proteina sconosciuta
mobilità relativa (mm)
Analisi qualitativa e quantitativa
di un SDS-PAGE
1.
2.
3.
4.
5.
STD
Omogenato 5 µl
I purif. 10 µl
II purif. 15 µl
III purif. 20 µl
0,5µg
2,5 µg
Alternativa:
densitometria a scansione
Resa=
mg o U di proteina di interesse nella frazione
mg o U di prot. di interesse all’inizio della purificazione
mg di proteina di interesse nella frazione
Purezza = mg di proteine totali presenti nella frazione
Attività
specifica
U di proteina di interesse nella frazione
=
mg di proteine totali presenti nella frazione
La messa a punto di un procedimento sperimentale di
purificazione di una proteina richiede la possibilità di:
I. un sistema per identificare e quantizzare la proteina di
interesse fra tutte le altre proteine del campione
[Gli ENZIMI possono essere identificati dalla reazione che essi
catalizzano (SAGGIO ENZIMATICO)]
II. un sistema per misurare la quantità di proteine totali nel
campione
III. un sistema per valutare la presenza di proteine
contaminati presenti nel campione di interesse
I. Saggio enzimatico
E’ necessario disporre di un opportuno saggio che consenta di
seguire la proteina durante i vari passaggi del procedimento di
purificazione
rapido (eseguibile su molti campioni)
quantitativo
“economico” (eseguibile con una piccola quantità di proteina)
specifico
SAGGI DI ATTIVITA’ ENZIMATICA
• Gli enzimi possono essere usati come strumenti analitici per
identificare o quantificare sostanze chimiche specifiche in
soluzione
• E’ possibile determinare la presenza e la quantità di un
determinato enzima in un fluido biologico misurandone l’attività
con substrati specifici
CONDIZIONI SPERIMENTALI DEL SAGGIO
Controllo del pH con una soluzione tampone
Controllo della temperatura
Assenza di inibitori (agenti chelanti, metalli)
In un saggio di attività enzimatica si misura il prodotto della
reazione, più di rado il consumo di substrato
• SAGGI DIRETTI
E + S E + P (prodotto colorato – cromoforo o fluorescente – fluoroforo)
FOSFATASI
R-O-PO32- + H2O
R-O-H + HO-PO32-
Gruppo di enzimi che
catalizzano l’idrolisi
di monoesteri del fosfato
NO2
NO2
O -PO3
2-
+ H2O
FOSFATASI
O-H + HO-PO32-
p-nitrofenil-fosfato
p-nitrofenolo
INCOLORE
GIALLO
• SAGGI DIRETTI
O
C
O-
C
O
+
+(ossidato)
NADH
CH3
Piruvato
O
C
H C
Lattato
Deidrogenasi
(LDH)
OOH
CH3
Lattato
+
NAD+
(ridotto)
Le reazioni che richiedono nucleotidi purinici sono molto usate
nei metodi enzimatici di analisi
Tali coenzimi (NAD, NADH, NADP, NADPH) sono ideali perché
sono usati stechiometricamente in un gran numero di reazioni di
ossido-riduzione
Composto ridotto + NAD+
NAD ossidato
(non assorbe a 340 nm)
Composto ossidato + NADH
NAD ridotto
(assorbe a 340 nm)
misurazione dell’assorbimento a 340 nm
saggio enzimatico non specifico: evidenzia tutti gli
enzimi che usano il NAD+
• SAGGI ACCOPPIATI
E1 + S E1 + P (prodotto non colorato o fluorescente)
E2 + P E2 + Q (secondo prodotto, Q, colorato)
Esempio:
COO+H N
3
H3C
C
L-treonina
COO-
aldolasi
H
+H N
3
CH2
C
+
H
O
H3C
C
H
H
OH
glicina
L-treonina
O
H3C
C
+
acetaldeide
alcol
deidrogenasi
NADH
H
OH
H3C
acetaldeide
CH2
alcol etilico
ACCOPPIAMENTO CON REAZIONE IRREVERSIBILE
A
E1
E2
B C
+
NAD+
Metodi radioisotopici
• Si basano sull’utilizzo di forme radiomarcate di un substrato
• Sono metodi molto sensibili ma limitati alle applicazioni in cui è possibile
separare facilmente i substrati dai prodotti
Esempio: misura dell’attività di un’amminotrasferasi
R2
R1
1a FASE
+H N
3
14
C
COO-
+
C
R1
COO -
14
C
O
H
R2
COO-
+
+H N
3
C
COO-
O
H
Miscela
di
reazione
2a FASE
(separazione cromatografica
e conteggio della radioattività)
lavaggio
eluizione
colonna
cromatografica
a scambio anionico amminoacidi chetoacidi
Conteggio
radiattività












