Metabolismo Biosintetico
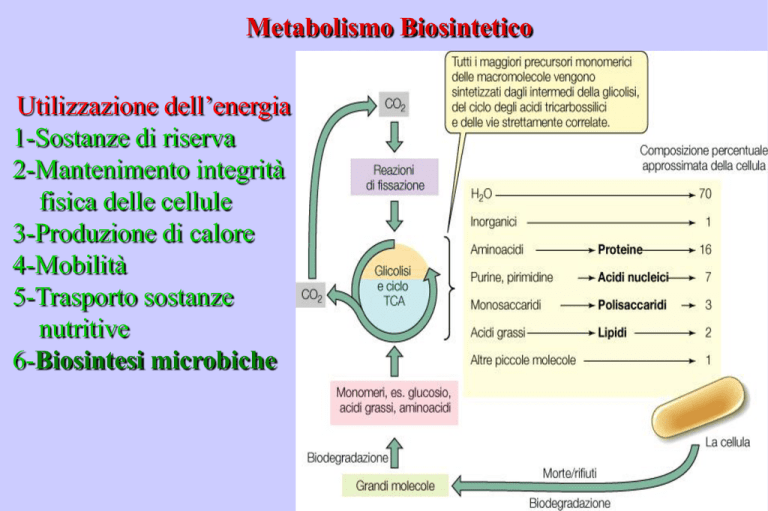
Utilizzazione dell’energia
1-Sostanze di riserva
2-Mantenimento integrità
fisica delle cellule
3-Produzione di calore
4-Mobilità
5-Trasporto sostanze
nutritive
6-Biosintesi microbiche
Principali precursori
delle reazioni
biosintetiche
-Glucosio 6 fosfato
-Fruttosio 6 fosfato
-Ribosio 5 fosfato
-Eritrosio 4 fosfato
-Gliceraldeide 3 fosfato
-3fosfoglicerato
-Fosfoenolpiruvato
-Piruvato Acetil-CoA
-Ossalacetato
-a-Chetoglutarato
-Succinil-CoA
Utilizzazione
dell’energia
Ribulosio
1,5-difosfato
carbossilasi
Ciclo di
Calvin
1-Carbossilazione
Fissazione
Autotrofa
della CO2
3-Rigenerazione
2-Riduzione
1-Carbossilazione
3-Rigenerazione
Ciclo di Calvin
2-Riduzione
Assimilazione del Fosforo
P i > ATP:
-Fosforilazione ossidativa
-Fotofosforilazione
-Fosforilazione a livello
del substrato
Assimilazione dei Solfati
H2S + O-Acetilserina >
Cisteina + Acetato (Batteri)
H2S + Serina > Ac.CoA >
Cisteina + H2O (Funghi)
H2S
Assimilazione dell’N2
A-Assimilazione dell’NH3
1-Glutammato deidrogenasi
2-Glutammina sintetasi
3-Amminazione riduttiva
Alanina-deidrogenasi
Ac.Piruvico + NH4+ + NADPH
+ H+ > L-Alanina + NADP+ +
H2O
1
B-Assimilazione del NO3
NO3 + NADPH + H+ > NO2
+ NADP+ H2O (Nitrato
Reduttasi)
Reazioni di transaminazione
NO2 + 2e- + H+ > NH2OH >
NH3 (Nitrito Reduttasi)
2
Fissazione dell’N2
Struttura dell’enzima Nitrogenasi
Dinitrogenasi
Reduttasi
28-32 Fe
4 Fe
FeMo-co
Dinitrogenasi
Struttura FeMo-co
Fissazione dell’N2
Donatori di elettroni:
-reazioni luminose
-NADH
Ferredossina
-H2
-Piruvato
Struttura genetica del Regulone nif (Klebsiella pneumonie)
Regolazione N fissazione
Enzima Nitrogenasi > inibito dall’O2, N combinato (NH3, NO3-)
Aerobi liberi:
Organismi azoto fissatori
Chemioorganotrofi– Azotobacter, Azomonas, Klebsielle,
Beijerinckia, Bacillus polymyxa, Methylomonas,
Methylococcus, Azospirillum, Mycobacterium, Citrobacter.
Fototrofi – Cianobatteri
Chemiolitotrofi – Thiobacillus, Streptomyces, Alcaligenes
Anaerobi liberi: Chemioorganotrofi – Clostridium, Desulvibrio,
Desulfotomaculum
Fototrofi– Chromatium, Thiocapsa, Chlorobium, Rhodobacter,
Rhodospirillum, Heliobacterium ecc.
Chemiolitotrofi – Archea (Methanococcus, Methanosarcina,
Simbionti:
Methanobacterium, Methanospirillum)
Leguminose – Rhizobium, Bradyrhizobium, Sinorhizobium,
Azorhizobium
Non Leguminose - Frankia
Biosintesi dei carboidrati
Gluconeogenesi
Piruvico
carbossilasi
Fosfoenolpiruvico
carbossicinasi
Fruttosio
difosfatasi
Esosi
Decarbossilazione
Biosintesi dei carboidrati
Pentosi
Biosintesi dei carboidrati
Sintesi dei Polimeri
Attivazione degli esosi
Scheletro di carbonio + NH2
Biosintesi
degli
aminoacidi
Biosintesi delle purine e pirimidine
Precursore della purina
(Scheletro)
Acido orotico
Ribosio-5-P
Precursore della pirimidina
(Scheletro)
Acido Inosinico
Precursore nucleotidi purinici
Adenina, Guanina
Uridilato
Precursore nucleotidi
pirimidinici
Timina, Citosina, Uracile
Origine nove atomi anello purinico
Origine sei atomi anello pirimidinico
Biosintesi delle purine e pirimidine
Biosintesi dei lipidi Complesso acido grasso sintetasi
Acil-Carrier-Protein
Biosintesi dei Biopolimeri
Polisaccaridi:
Omopolimeri A-A-A-AEteropolimeri regolari A-B-A-B
Acidi Nucleici
Eteropolimeri irregolari A-C–B
Proteine
Eteropolimeri irregolari A-C-B
Biosintesi metaboliti basso
peso molecolare > 1 enzima
per ogni reazione
DNA
Complementarietà
Biosintesi macromolecole > messaggero
sistema ezimatico specifico
Codice genetico
per tipo di legame fra
monomeri
Trascrizione
Traduzione
Procarioti
Eucarioti
Struttura del DNA
Complementarietà e
natura antiparallela
del DNA
Replicazione del DNA
Sintesi del DNA
Semiconservativa–Filamento Stampo
Direzione 5’fosfato > 3’ossidrile
Sintesi del DNA Replicazione
Proteine per la replicazione
DNA girasi (Topoisomerasi II) Introduce i Superavvolgimento del DNA
Topoisomerasi I
Rimuove i Superavvolgimento del DNA
SSB (proteine)
Legano DNA a singolo filamento
DnaA
Fattore di inizio (proteina specifica)
DnaB
Srotola il DNA -DnaC, DnaT
HU
Proteina lega il DNA
PriA
Formazione Primisoma 3 <5
PriB
Formazione primisoma
PriC Elicasi
Svolge DNA 5 >3
Primasi
Sintesi Primer di RNA
DNA Polimerasi III Sintesi DNA
DNA Polimerasi I
Rimuove Primer RNA e sintetizza frammenti di DNA
DNA ligasi
Lega frammenti di Okazaki
Ter
Terminazione replicazione
Primer
Forca di replicazione
Replicazione del DNA
Replicazione del DNA
Replicazione bidirezionale
Proteine FtsZ
del Divisoma
Intervengono dopo
la replicazione per
ripartire il DNA
nelle cellule figlie
Replisoma
Replicazione del DNA
Correzione
Correzione errori della replicazione: Mutazioni
1 errore 10-8 – 10-11 appaiamenti
Sintesi dell’RNA - Trascrizione
Tipi di RNA
mRNA messaggero
rRNA ribosomale
tRNA trasferimento
Terminazione sintesi mRNA
-Terminatori
-Fattore rho (Proteina legata
all’RNA)
-Terminatori intriseci
(Specifiche sequenze
nucleotidiche del DNA)
Terminatori
Sintesi delle proteine Traduzione
Codice genetico
Codone inizio: AUG (codifica per N-formilmetionina, inizio di ogni mRNA)
Codoni di stop: UAA, UGA, UAG
Aminoacido > Codone > Anticodone-tRNA
Sintesi delle proteine
Struttura del tRNA
Batteri >
60 tRNA
diversi
Disposizione reale
Aminoacido - t RNA - aminoacil-tRNA sintetasi ( Enzima specifico)
1-Attivazione
Sintetasi+aa +
ATP >
Aminoacil-AMP
+Ppi
2 -AminoacilAMP + tRNA >
Aminoacil-tRNA
+AMP
2
1
Enzima Aminoacil-tRNA
Sintetasi
Riconosce sia il tRNA che
l’aa specifico al codone
tRNA
Trasporto verso i Ribosomi
Sintetasi
Meccanismo Sintesi Proteicaa a-Inizio b-Allungamento c-Terminazione
peptidico accettore
uscita
Catena nascente
Meccanismo Sintesi Proteica
Polisoma
Formazione dei legami peptidici - reazione di peptidiltransferasi
Catalizzata dagli rRNA - subnità 50S (rRNA 23S)
c-Terminazione (Fattori di rilascio RF – Tagliano il polipetide)
Ribosoma si muove lungo l’mRNA e raggiunge i codoni di stop
U A A, U A G, U G A


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)








