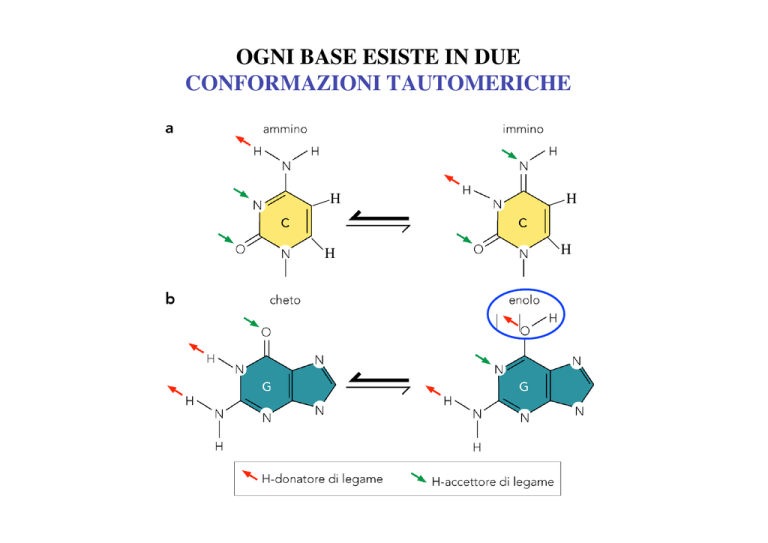
OGNI BASE ESISTE IN DUE
CONFORMAZIONI TAUTOMERICHE
H
H
H
H
La tautomeria amino-imminica
Imino
Amino
A
C
Imino
C
Amino
A
I tautomeri alternativi sono causa di errori durante la sintesi del DNA
La tautomeria cheto-enolica
Enolo
Chetone
T
G
Enolo
T
Chetone
G
Strutture secondarie insolite del DNA
Strutture scivolate
Strutture cruciformi
DNA a tripla elica
Il DNA triplex si forma grazie
alla formazione di legami H fra
le coppie di basi di Hoogsteen
6
7
4
3
6
7
4
3
DNA 105 pb
T= 10
W= 0
Lk0= 10
DNA 105 pb
T= 10
W=-1
Lk= 9
∆Lk = Lk - Lk0= 9 - 10 = -1
La quantità di
superavvolgimenti di un
DNA è detta differenza
di linking e corrisponde
al ∆Lk
Lk0= Linking number di un cccDNA rilassato
Le DNA TOPOISOMERASI umane
DNA
topoisomeras
i umane
I
Tipo
IB
Taglio
sul DNA
Ruolo strutturale
ssb
Rilassamento di DNA
superavvolto sia (+) che (-)
IIIα
IA
ssb
IIIβ
IA
ssb
IIα
IIβ
IIA
IIA
Rilassamento solo di DNA
superavvolto
negativamente
Rilassamento solo di DNA
superavvolto
negativamente.
Funzione
Replicazione
Trascrizione
Ricombinazione
Ricombinazione
Trascrizione dei geni
dell’RNA ribosomiale
Ricombinazione
dsb
Rilassamento di DNA
superavvolto sia (+) che
(-). Facilita lo scioglimento
di nodi o il decatenamento
Condensazione dei
cromosomi
Segregazione dei
cromosomi
Replicazione
dsb
Rilassamento di DNA
superavvolto sia (+) che
(-). Facilita lo scioglimento
di nodi o il decatenamento
Non ben definita
DNA Topoisomerase I
= n s.a.
= n – 1 s.a.
L’enzima taglia un filamento della doppia elica, fa passare il filamento integro
attraverso il taglio e poi richiude il taglio: il n° topologico (Lk) aumenta di
un’unità
DNA Gyrase

![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)




![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)

