FUNZIONI DEL DNA E FLUSSO DELL’ INFORMAZIONE GENETICA
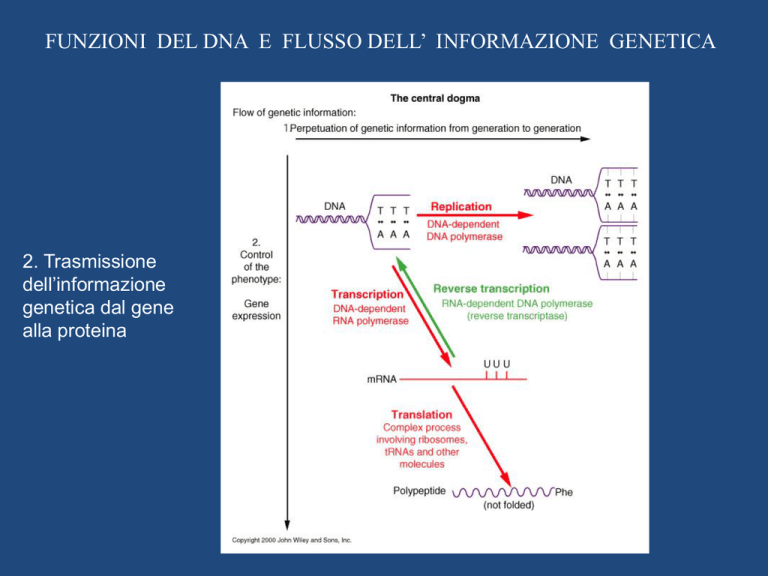
2. Trasmissione
dell’informazione
genetica dal gene
alla proteina
LA TRASCRIZIONE NEI PROCARIOTI E NEGLI EUCARIOTI
PROCARIOTI
EUCARIOTI
LA TRASCRIZIONE NEI PROCARIOTI E NEGLI EUCARIOTI
Filamento
stampo
Filamento
senso
Filamento
senso di
RNA
LA TRASCRIZIONE NEI PROCARIOTI E NEGLI EUCARIOTI
La sintesi di RNA sulla elica
stampo del DNA è catalizzata
dall’enzima RNA polimerasi
LA TRASCRIZIONE NEI PROCARIOTI
La sintesi delle proteine avviene sugli mRNA
(ad opera di più ribosomi) mentre l’mRNA
si sta ancora sintetizzando.
LA TRASCRIZIONE NEI PROCARIOTI
L’ RNA polimerasi dei Procarioti sintetizza: gli RNA messaggeri mRNA
gli RNA transfert tRNA
e gli RNA ribosomali rRNA
Nei batteri (per es. in E. coli) la RNA polimerasi è
costituita da 4 subunità del nucleo enzimatico e un
fattore sigma () dissociabile. Il fattore sigma
agisce nel riconoscimento del promotore e viene
rilasciato con l’allungamento dell’RNA.
Il Promotore dei Procarioti
La struttura di un promotore tipico di E. coli:
La sequenza a -10
TATAAT è chiamata
Pribnow box
L’RNA polimerasi si
lega alla sequenza -35
del promotore ed inizia
lo svolgimento dei
filamenti di DNA nella
sequenza -10
La
trascrizione
comincia nella bolla
di trascrizione in un
sito da cinque a nove
paia di basi oltre la
sequenza a -10.
LE FASI DELLA TRASCRIZIONE NEI PROCARIOTI
LA TERMINAZIONE DELLA TRASCRIZIONE
La terminazione delle catene di RNA nei
procarioti avviene quando l’RNA polimerasi
incontra un segnale di terminazione.
Questi segnali sono sequenze specifiche che
possono ripiegarsi assumendo una struttura
detta a forcina che causa la terminazione
della trascrizione (terminatori rhoindipendenti)
oppure possono ripiegarsi e interagire con
una proteina rho determinando la fine della
trascrizione (terminatori rho-dipendenti).
Successivamente si ha il distacco dell’RNA.
LA TRASCRIZIONE NEGLI EUCARIOTI
Cinque tipi di RNA trascritti negli EUCARIOTI:
mRNA -------> RNA messaggero, trasporta l’informazione genetica nel citoplasma per la sintesi
delle proteine
tRNA --------> RNA transfer, specifici per i vari aminoacidi, per la sintesi proteica
rRNA --------> RNA ribosomale, componente dei ribosomi, per la sintesi proteica
snRNA -------> (small nuclear RNA) piccoli RNA nucleari, per lo splicing dei messaggeri
miRNA ----- (micro RNA) altri piccoli RNA nucleari, per la regolazione post trascrizionale
Le RNA polimerasi degli EUCARIOTI sono tre
Enzima
Localizzazione
Prodotti
RNA polimerasi I
Nucleolo
rRNA 28S (25S), 18S e 5,8S
RNA polimerasi II
Nucleo
mRNA, alcuni snRNA, miRNA
RNA polimerasi III
Nucleo
tRNA, rRNA 5S, alcuni snRNA
INIZIO DELLA TRASCRIZIONE di un gene eucariotico
L’RNA pol II non riconosce il promotore senza
l’intervento dei Fattori di Trascrizione TFII
TFIID (Transcription Factor II D) si lega al TATA
box del promotore
seguono gli altri fattori di trascrizione A, B e F
la RNA polimerasi II
TFIIE si lega per ultimo
Si forma il complesso di inizio della trascrizione
La doppia elica di DNA si apre per l’azione di TFIIF
FASE DI ALLUNGAMENTO
interviene un altro fattore di
trascrizione TFII S e vengono
rilasciati il B e F
( i TFII D e A rimangono legati al
promotore per facilitare l’attacco di
una nuova RNApol II)
La RNA pol II catalizza la
formazione del legame
fosfodiesterico tra il 5’P del
ribonucleotide trifosfato da inserire
e il 3’OH dell’ultimo
ribonucleotide inserito
Il filamento di RNA cresce in
direzione 5’3’
LA TERMINAZIONE DELLA TRASCRIZIONE
La terminazione della trascrizione
NON avviene per arresto della
trscrizione in corrispondenza di
specifiche sequenze dette
Terminatori come nei Procarioti
Intervengono una endonucleasi e
una esonucleasi specifiche
Quando la esonucleasi raggiunge
la RNApol II la trascrizione si
ferma
PROCARIOTI
IL GENE CONTINUO
EUCARIOTI
IL GENE DISCONTINUO
PROPRIETA’ CHIMICO-FISICHE DEL DNA
Denaturazione del DNA
Riassociazione del DNA
Queste proprietà del DNA sono state sfruttate nei primi studi di caratterizzazione del genoma e oggi sono alla base di tutte
le moderne tecniche di biologia molecolare come l’ibridazione molecolare su filtro, l’ibridazione in situ, la PCR ecc.
STRUTTURA DEL GENE EUCARIOTICO Chambon e coll, 1978
Fotografia al microscopio elettronico dell’ibrido
fra mRNA e gene per l’ ovoalbumina di pollo
Interpretazione
Gene per l’ovoalbumina di pollo
in verde gli 8 esoni L, 1-7, che formano
l’ibrdo DNA – mRNA
e i 7 introni A-G, (da 251 a 1600 pb),
che non si ibridano con l’RNA
Gene codificante l’ovoalbumina di pollo
LE SEQUENZE NEL SITO DI GIUNZIONE ESONE–INTRONE
SONO CONSERVATE
STRUTTURA GENERALE DEL GENE EUCARIOTICO
Darnell: un gene è costituito da tutte le sequenze di acido nucleico (di solito
DNA) che sono necessarie alla produzione finale di un polipeptide o di un RNA
comprende quindi le sequenze promotrici che ne regolano l’espressione
Una volta chiarita la struttura del gene eucariotico
la domanda era:
gli introni non si ritrovano nell’mRNA perché non vengono trascritti
oppure vengono trascritti e poi rimossi?
Elenco dei partecipanti alla esercitazione
TRASCRIZIONE E MATURAZIONE DELL’RNA MESSAGGERO
NEGLI EUCARIOTI
L’RNA messaggero è sintetizzato dalla
RNApol II nel nucleo e poi trasportato
nel citoplasma
Gli RNA trascritti sono “monogenici”
Gli mRNA prima del trasporto nel
citoplasma subiscono tre modifiche
principali:
(a) aggiunta all’estremità 5’ di un
cappuccio (cap) di 7-metil guanosina;
(b) aggiunta all’estremità 3’ di una coda di
poli-A;
(c) quando presenti, le sequenze introniche
vengono rimosse tramite splicing.
Il cap in 5’ è importante per l’inizio della traduzione e sembra in grado di proteggere l’mRNA
dalla degradazione (Buchanan)
La coda di poliA viene sintetizzata dalla PoliAPolimerasi in 3’ a valle della sequenza AAUAAA
(dopo il taglio fatto da una endonucleasi che con la PAP e altre 4 proteine forma un complesso multipro)
La coda di poliA (20-250 A) facilita il trasporto dell’ mRNA nel citoplasma e lo stabilizza
L’RNA messaggero immaturo contiene le sequenze corrispondenti agli introni che poi vengono rimosse
MECCANISMO DI SPLICING DEI PRE-mRNA : “ TAGLIA E CUCI ”
SPLICEOSOMA o Complesso di splicing
è costituito da proteine associate a piccoli
RNA detti snRNA (small nuclear RNA)
U1, U2, U4, U5, U6
Il primo taglio avviene al 5’ dell’introne
U2 si lega alla sequenza di ramificazione
UACUUAUCC che forma la struttura a cappio
con l’estremità 5’ dell’introne GU
Si forma un legame fosfodiesterico “anomalo”
G-A
L’estremità 3’ dell’introne viene tagliata e i
due esoni congiunti
GLI mRNA MATURI CONTENGONO LA 5’ UTR e LA 3’ UTR
La sequenza guida o 5’ UTR serve per l’attacco al ribosoma e per iniziare la sintesi proteica
La sequenza di coda o 3’ UTR stabilizza il messaggero
Entrambe interagiscono con le proteine del trasporto dei mRNA e con proteine dei ribosomi
LA REGIONE DEL PROMOTORE
Struttura di un promotore riconosciuto dalla RNA polimerasi II.
Il box TATA serve a specificare il sito di inizio della trascrizione
è riconusciuta da un fattore di inizio trascrizione
Il box CAAT favorisce la trascrizione, se la seq è modificata si
ha riduzione del livello della trascrizione
I box GC servono a legare la RNApolimerasi II
GLI ENHANCER E I SILENCER
a
Eucarioti unicellulari
TATA box, UAS sequenza attivatrice a monte, Silencer
sequenza silenziatrice
Sono tre elementi circa a 200pb a momte del sito inizio
della trascrizione
b
Eucarioti pluricellulari
TATA box
INR sequenza iniziatrice trascrizione
RE elementi regolatori
costituiscono il promotore prossimale
Gli enhancer e i silencer sono elementi
distali del promotore, possono essere
localizzati anche 10-50 Kb a monte o a
valle della TATA box
ATTIVATORI, COATTIVATORI E REPRESSORI
Gli henancer esercitano la loro azione
tramite gli attivatori e i coattivatori
secondo il modello della figura
I repressori si legano ai silencer
interferendo con l’ azione degli
attivatori
TFIID, B, A, F, E e H sono i fattori
BASALI della trascrizione
FINE
I GENI CHE CODIFICANO GLI RNA RIBOSOMALI
I RIBOSOMI
4
6
I GENI CHE CODIFICANO GLI RNA RIBOSOMALI
Fotografia al microscopio elettronico di geni ribosomali
in trascrizione
Interpretazione
ORGANIZZAZIONE DEI GENI RIBOSOMALI 25S, 18S e 5,8S
3’
I geni ribosomali sono sequenze ripetute in “tandem”
5’
livello di ripetizione ca 103 copie/genoma
Ogni unità di ripetizione comprende un gene per il 25S uno per il 5,8S e uno per il 18S
tra loro dagli spaziatori interni (ITS)
separati
Ogni unità di ripetizione è separata dalle altre dallo spaziatore esterno (NTS+ETS)
Il promotore della trascrizione dell’rDNA
in X. laevis
I terminatori della trascrizione dell’rDNA.
TRASCRIZIONE E MATURAZIONE DEGLI RNA RIBOSOMALI 25S, 18S e 5,8S
25 S
I GENI PER GLI RNA RIBOSOMALI 5S
Organizzazione delle due unità di ripetizione del gene per l’rRNA 5S in frumento.
•
A e B spaziatore nelle unità di 410 pb (sopra) e di 500 pb (sotto): la regione B è formata da sequenze
perfettamente omologhe nelle due unità ribosomali.
•
La trascrizione è svolta dalla RNApol III. La posizione +1 indica il punto di inizio della trascrizione
•
Non hanno il promotore esterno ma due regioni ICR interne al gene
I GENI PER GLI RNA TRANSFER
Struttura
degli RNA transfer
LA TRASCRIZIONE E LA MATURAZIONE DEGLI RNA TRANSFER
La trascrizione dei geni per gli RNA transfer viene svolta dalla RNA pol III
Lo splicing dei tRNA avviene ad opera di una endonucleasi e di una ligasi di splicing
Splicing autocatalitico dell’rRNA
di Tetrahymena termophila
• Nelle piante lo splicing autocatalitico avviene in
alcuni precursori di tRNA e rRNA mitocondriali e
cloroplastici
•
Gli RNA capaci di compiere splicing autocatalitico
sono detti RIBOZIMI
FINE









![Lezione 12 Trascrizione e maturazione RNA [modalità compatibilità]](http://s1.studylibit.com/store/data/004956814_1-03488078208fbae40d6f98c42ec7fd9f-300x300.png)


