Meccanismi di riparazione del DNA
•
•
•
I danni al DNA sono una continua minaccia alla stabilità genomica di una cellula.
Gli organismi viventi hanno evoluto vari meccanismi per proteggere l’integrità del DNA
Questi meccanismi si basano sulla attività degli enzimi della riparazione del DNA.
I molteplici sistemi di riparazione del DNA si sono evoluti per salvaguardare l’integrità dell’informazione
genetica negli organismi viventi. Ogni via di riparazione è specializzata nel correggere specifici tipi di danno al
DNA
Esistono numerosi tipi di lesioni, un singolo processo di riparazione non potrebbe fare fronte a tutti i tipi di
danno. L’evoluzione ha modulato una grande varietà di sofisticati sistemi di riparazione che riescono a
recuperare la maggioranza degli insulti inflitti all’informazione genetica cellulare
I sistemi di riparazione del DNA devono essersi sviluppati molto presto nell’evoluzione, e per questo sono
sistemi altamente conservati
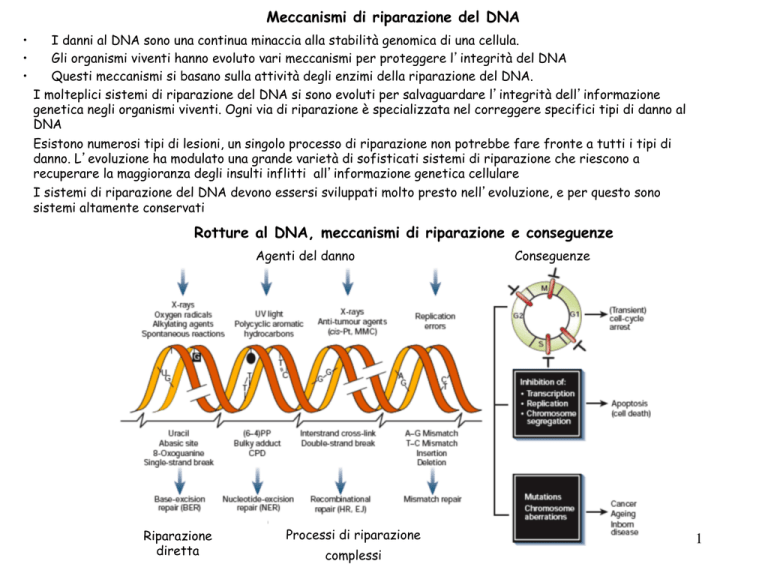
Rotture al DNA, meccanismi di riparazione e conseguenze
Agenti del danno
Riparazione
diretta
Processi di riparazione
complessi
Conseguenze
1
Wolters, Genome maintenance and transcription integrity in aging and disease. Frontiers in Genetics, 4, article 19, 110, 2013
doi: 10.3389/fgene.2013.00019
Fig 1. Diverse lesion types trigger DNA damage responses.
DNA damage can be caused by various
genotoxic agents, such as Reactive Oxygen
Species (ROS) produced during cellular
metabolism, Alkylating Agents that find
application in cancer therapy, Ionizing
radiation (IR), which is used for radio
therapy, or UltraViolet (UV) irradiation
presenting a daily threat as it is contained in
sunlight.
The inflicted lesions are just as diverse, since
- ROS usually lead to base modifications;
- alkylating agents form adducts, while
- bifunctional alkylating agents crosslink DNA
to form Interstrand CrossLinks (ICLs).
- IR typically induces Double-Strand Breaks
(DSBs), and
- UV light triggers the formation of
Cyclobutane Pyrimidine Dimers (CPDs) and
6,4-PhotoProducts (6,4-PPs).
Cells have a repertoire to sense the different
lesions and subsequently activate DNA damage
checkpoint proteins.
Ultimately, cells respond to the DNA damage
by chromatin remodeling, modified transcription,
fine-tuning of energy metabolism, cell cycle
arrest, activation of DNA repair pathways and
in case of irreparable damage load, induction
2
of senescence or apoptosis.
Gli agenti alchilanti si dividono in:
] bifunzionali: possono formare dei legami a ponte (cross-link) tra
due filamenti del DNA o all’interno dello stesso filamento
provocando la rottura completa della molecola di DNA,
oppure possono causare un blocco della trascrizione e della
crosslink at N-7 guanine
on both strands
(G-G crosslink)
nitrogen mustard
duplicazione (nitrogen mustard)
] monofunzionali: si legano ad una sola parte e non danno luogo a dei cross-links, ma
agiscono spezzando la catena del DNA inattivandolo (ethyl methanesulfonate EMS).
➾ Alcuni agenti alchilanti, come l’ethylmetane sulfonate (EMS) agiscono trasferendo gruppi
etilici e metilici al DNA, causando disappaiamento delle basi.
➾ Altri agenti alchilanti attaccano preferenzialmente adenina e guanina (purine) rendendo
più labile il legame con il deossiribosio e favorendo la depurinazione.
La replicazione del DNA apurinico può generare una mutazione per sostituzione casuale
della base.
3
Meccanismo
Lesione al DNA
Sistemi enzimatici
Rip diretta:
fotoriattivazione
Dimeri di pirimidina
DNA fotoliasi
Rip diretta:
transmetilazione
Metilazione-alchilazione
Alchil-metiltransferasi
Escissione di basi
(BER)
Basi danneggiate (es.
alchilate)
Glicosilasi+endonucleasi
Escissione di
nucleotidi (NER)
Dimeri di pirimidina
Grossi addotti delle basi
UvrA,B e C in E. coli
XPA, B, C, D, E, F, G in h.
sapiens
MisMatch repair
(MMR)
Errori di replicazione
(appaiamenti non corretti)
MutS, MutL, MutH in E. coli
MSH, MLH e PMS in h.
sapiens
Ricombinazione
omologa e non
omologa
Rotture a doppio filamento RecA e RecB in E. coli
Sistemi di ricombinazione
RISPOSTE CELLULARI AL DANNO AL DNA: I MECCANISMI DI RIPARAZIONE
• REVERSIONE DIRETTA DELLE LESIONI
• fotoriattivazione (in procarioti e eucarioti, ma assente nei mammiferi placentati)
• transmetilazione: riparazione di O6-metilguanina (nella maggior parte delle specie)
• ESCISSIONE DELLE LESIONI
• excisione di basi (BER)
• excisione di nucleotidi (NER)
• RIPARAZIONE DEGLI ERRORI DI APPAIAMENTO TRA LE BASI (MMR)
• RIPARAZIONE RICOMBINATIVA
• Ricombinazione omologa (HR)
• Ricombinazione non omologa (NHEJ)
4
Formation of the most toxic and mutagenic DNA lesion, cyclobutane-pyrimidine dimers by UV radiation.
Dimers can form between two adjacent pyrimidines.
CH⌃
3 CH3
thymine-thymine
cyclobutane-pyrimidine dimer (CPD)
dipyrimidine bases
pirimidina (6-4) pirimidone.
In questo caso i raggi UV aprono i
legami in C-5 C-6, uno degli anelli
pirimidinici ruota e il suo C-4 forma
un legame con il C-6 dell’ altro anello.
6-4PhotoProduct)
(6-4 pirimidina-pirimidone
thymine-cytosine dimer and their
photoreactivation by the enzyme
photolyase in the presence of light.
dipyrimidine bases
5
L’esposizione del DNA ai raggi UV determina il legame covalente tra due pirimidine adiacenti.
Queste strutture vengono chiamate dimeri ciclobutano pirimidina.
(a) Formation of thymine dimer;
(b) Distorted DNA.
FOTORIATTIVAZIONE
L’enzima DNA fotoliasi (deossiribodipirimidina foto-liasi) catalizza la monomerizzazione dei dimeri di pirimidina.
E’ associata a gruppi cromofori come
- FADH2 (flavina adenina dinucleotide ridotto) coenzima ossiriduttivo, partecipa a
innumerevoli reazioni che comportano il trasferimento di 1 o 2 elettroni ;
- pterina (composto aromatico, che deriva da una molecola di pteridina 2-amino,
4-cheto sostituita;
assorbono fotoni (luce da 300 a 500 nm) e catalizzano la destabilizzazione del legame tra le due pirimidine
mediante un trasferimento di elettroni.
La DNA fotoliasi cattura dal sole
l’energia necessaria per rompere
i legami covalenti che uniscono le
pirimidine adiacenti
FOTORIATTIVAZIONE
6
Transmetilazione: trasferimento del gruppo metilico (o alchilico)
La O6-metilguanina è una lesione altamente mutagenica che permette alla guanina di appaiarsi sia con la citosina che con la
timina. Confonde il sistema di riparazione degli appaiamenti errati delle basi (MMR) poiché innesca inutili cicli di rimozione
di mismatch e successiva reincorporazione della base sbagliata da parte della riparazione replicativa.
La metiltransferasi trasferisce il gruppo metilico
dalla guanina ad uno dei proprio residui di cisteina.
MGMT inattiva
RISPOSTE CELLULARI AL DANNO AL DNA: I MECCANISMI DI RIPARAZIONE
ESCISSIONE DELLE LESIONI
Base Excision Repair (BER)
Escissione di Basi e Riparo (BER)
1. Pathway principale per la riparazione di basi
modificate, incorporazioni di nucleotidi errati,
danni ossidativi, siti apurinici e rotture a singolo
filamento.
2. Varie DNA glicosilasi riconoscono le lesioni e
rimuovono la base al suo legame glicosidico generando
siti abasici o AP (apurinic/ apyrimidinic)
3. Una delle numerose AP endonucleasi incide lo scheletro
fosfodiesterico adiacente al sito AP
4. Il nucleotide AP viene rimosso da una esonucleasi/dRPase e il
tratto mancante di DNA viene riempito attraverso la sintesi
di DNA e ligazione.
7
La glicosilasi si lega
al DNA e lo distorce
per inserire la base
sospetta in una tasca
specifica
dove avviene il taglio
del legame glicosidico
tra base e
deossiribosio
Il sito AP è riconosciuto
da una AP endonucleasi
che incide al 5’ del
fosforibosio e genera un
gruppo
3’-OH per la polimerasi
che inserirà il nucleotide
corretto
Quando sono riparati
SSB intervengono la
poli-(ADP-ribosio)
polimerasi (PARP), la
polinucleotide chinasi
(PNK), a proteggere e
modificare le estremità
libere.
PCNA e la esoendo nucleasi
FEN1
determinano lo
scalzamento e la
rimozione del
filamento
danneggiato
PCNA, Proliferating
Cell Nuclear
Antigen)
8
Come fa la glicosilasi a rilevare la presenza di una base danneggiata mentre esamina il genoma?
Le glicosilasi scorrono lungo il solco minore del DNA finché non trovano la specifica
lesione. Quando la base si trova all’interno della doppia elica, la base danneggiata
viene ribaltata all’esterno così da protrudere fuori dalla doppia elica e sedersi nella
tasca enzimatica specifica
Nucleotide Excision Repair (NER)
1. Riconosce lesioni che bloccano la replicazione del DNA (es. fotodimeri di pirimidina indotti da UV e addotti
ingombranti indotti dagli idrocarburi policiclici aromatici)
2. Distorsioni nella doppia elica
3. In E. coli, mediata da UvrABCD
4. Negli eucarioti, molte proteine coinvolte
5. Può essere generale per tutto il genoma (Global Genome Repair-GGR) oppure strettamente associata alla
trascrizione (Transcription Coupled Repair-TCR)
9
Riparazione attraverso escissione nucleotidica in E. coli
ATP
A A
ADP+Pi
Piegamento del DNA
(bolla di denaturazione)
Taglio al 5’ ed al 3’
(8 nucl. a monte e
4-5 a valle)
UvrA
UvrB
Elicasi D
B
ATP
UvrC
ADP+Pi
C
12-mer
POLI
B
B
12 nt sostituiti
10
Transcription-Coupled Repair (TCR)
Il transcription Repair Coupling Factor (TCRF) lega il
complesso della RNA polimerasi bloccato dal danno e recluta il
complesso UvrA2B
11
TCR in mammifero
⇒ Formazione di un grosso complesso
proteico insieme alla RNA polimerasi
in stallo (complesso TCNER).
⇒ Il complesso disloca la polimerasi
favorendo l’accesso delle proteine
richieste per il BER o per il NER.
⇒ Dopo la riparazione, la trascrizione
riparte.
I geni che codificano CSA e CSB sono mutati nella
sindrome di Cockayne (CS) (fotosensibilità della pelle)
12
(Nucleotide Excision Repair)
NER nei mammiferi
1.
Il danno viene riconosciuto da XPC che è legato ad
hHR23B
2.
Successivamente si legano XPA, RPA, TFIIH e XPG. Due
DNA elicasi che costituiscono TFIIH formano una bolla
nel DNA
3.
Si lega ERCC1-XPF
4.
L’endonucleasi XPG taglia l’elica danneggiata al 3’ del sito
di danno. ERCC1-XPF taglia l’elica al 5’ del sito di danno
(incisione bimodale) generanado un frammento di 27-30 nt
5.
Escissione del frammento, sintesi del DNA e ligazione
I geni che codificano XPA, B, C, E, F e G sono
mutati nello Xeroderma Pigmentoso
13
Wolters, Genome maintenance and transcription integrity in aging and disease. Frontiers in Genetics, 4, article 19, 110, 2013
doi: 10.3389/fgene.2013.00019
(modified)
14
Malattie umane causate da alterazioni nella riparazione per escissione di nucleotidi (NER)
MALATTIA
SINTOMI
FUNZIONI ALTERATE
Xeroderma
Pigmentoso (XP)
Sensibilità alla luce del
sole; cancro della pelle,
lesioni oculari, frequenti
anormalità neuronali,
invecchiamento prematuro
Riparazione del DNA
danneggiato da UV o sostanze
chimiche (ipersensibilità ai
raggi UV e agenti chimici)
XPA,XPB, XPC,
XPD, XPF, XPG
Sindrome di
Cockayne (CS)
Nanismo; senilità precoce;
atrofia oculare; ritardo
mentale; sensibilità alla
luce solare, normalmente
non associata al cancro
Riparazione del danno
ossidativo associata alla
trascrizione.
CSA, CSB, XPB,
XPD, XPG
Tricotiodistrofia
(TTD)
Capelli e unghie fragili,
Riparazione del DNA
ritardo fisico e mentale,
danneggiato da UV.
normalmente non associata
al cancro
GENE MUTATO
XPB
XPD
15
Xeroderma Pigmentosum
• Autosomica recessiva.
• 1/250.000 (Western countries) - 1/40.000 (Giappone)
• Sviluppo precoce di carcinoma cutaneo e delle cellule squamose,
melanomi, angiomi e sarcomi (50% dei pazienti)
• Degenerazione neuronale (20%)
• 10 gruppi di complementazione (eterogeneità di locus)
• Difetti principalmente nella riparazione per escissione dei nucleotidi (NER).
Mutazioni nei geni:XPA, B, C, E, F e G
Sindrome di Cockayne
• Malattia autosomica recessiva
• È caratterizzata da: sensibilità alla luce, nanismo, microcefalia,
difetti neuronali, sordità, invecchiamento precoce.
• Non presenta predisposizione al cancro
• Difetti principalmente in TCR
- Mutazioni nei geni CSA e CSB TCR: (transcription-Coupled Repair)
16
The initial damage response in TCR is mediated by
the coupling factors CSA and CSB associated with
the RNA polymerase II transcription elongation
complex.
The initial damage response in GGR is mediated by
the damage-recognition factors XPE and XPC.
Following damage recognition, the damaged site is
remodelled by the helicase activities of XPB and
XPD and binding of XPA and replication
protein A (RPA).
The nucleases XPG and XPF–ERCC1 then cut
the damaged oligo nucleotide region either side of
the damage and this is excised.
The repair patch is synthesized by proliferating-cell
nuclear antigen (PCNA) and replicative
polymerase (Polδ).
Damage that isnot excised must be replicated by
the action of Polδ.
CNS: Central Nervous System
RISPOSTE CELLULARI AL DANNO AL DNA:
I MECCANISMI DI RIPARAZIONE
• RIPARAZIONE DEGLI ERRORI DI APPAIAMENTO TRA LE BASI (MMR)
• RIPARAZIONE RICOMBINATIVA
• Ricombinazione omologa (HR)
• Ricombinazione non omologa (NHEJ)
17
In un primo ciclo di replicazione l’erronea incorporazione
di una base può introdurre una mutazione. Se l’appaiamento
errato sfugge al sistema di correzione di bozze da parte
dell’attività esonucleasica 3’-5’ delle DNA polimerasi
replicative (aumenta la fedeltà della replicazione di un
fattore 100), in un secondo ciclo replicativo, la mutazione
viene definitivamente fissata nella sequenza di DNA.
MisMatch Repair (MMR) in E. coli (Principale responsabile della fedeltà della replicazione)
(aumenta la correttezza della sintesi del DNA di un fattore tre)
L’omodimero MutS analizza il DNA e
riconosce i mismatch dalla distorsione che
essi producono sulla doppia elica
MutS si chiude intorno al DNA, induce su
di esso una profonda curvatura e subisce
una modificazione conformazionale
18
MutS recluta la proteina MutL che a sua volta attiva
l’endonucleasi MutH, un enzima che taglia un
filamento nelle immediate vicinanze del mismatch
L’elicasi UvrD svolge il DNA a partire
dall’incisione mentre una esonucleasi digerisce
progressivamente la singola elica spostata.
Si produce una regione a singolo filamento
che viene riempita dalla Pol III. Infine una
DNA ligasi rinsalda il nick
19
Come fa il sistema di riparazione a riconoscere quale dei due nucleotidi
scorrettamente appaiati deve sostituire?
La Dam metilasi aggiunge un gruppo metilico alla N6
dell’adenina nelle sequenze GATC (sequenza presente una
volta ogni 256 basi)
Dopo la replicazione, per alcuni minuti, le molecole di DNA
figlie, risultano emimetilate. Il filamento di nuova sintesi è
quindi riconoscibile dall’assenza di metilazione
MutH introduce selettivamente un nick sul filamento non
metilato
20
Direzionalità del sistema MMR
Se il DNA viene tagliato al 5’ del mismatch l’esonucleasi
VII o RecJ che degrada il DNA in direzione 5’-3’ rimuove
il tratto di DNA compreso tra il sito di taglio e il
nucleotide errato
Se il DNA viene tagliato al 3’ del mismatch il DNA
viene rimosso dall’esonucleasi VI che degrada la
doppia elica in direzione 3’-5’
21
Mismatch repair nei mammiferi
• L’eterodimero hMSH2/6 riconosce i mismatch
e i loops a singola base
• L’eterodimero hMSH2/3 riconosce i loops da
inserzione/delezione
• hMutLa e hMutLb interagiscono con MSH e con
i fattori di replicazione
• Sembra che i frammenti di Okazaki forniscano
il segnale di discriminazione tra elica parentale
e figlia
Difetti nel MMR nei mammiferi predispongono al cancro,
principalmente al cancro del colon non poliposico (HNPCC),
ma anche a cancro uterino, ovarico e gastrico
22
Sindrome di Lynch I: (carcinoma ereditario colon-rettale non poliposico, Hereditary Non-Polyposis Colon Cancer – HNPCC
È una forma ereditaria di tumore al colon con trasmissione dominante, il che significa che ha una probabilità del
50% di manifestarsi nei figli di chi ne è affetto.
Diversamente dalla poliposi adenomatosa familiare, la predisposizione allo sviluppo della malattia non si manifesta
con la comparsa di polipi, ma direttamente con lo sviluppo del cancro al colon, in genere intorno ai 45 anni di età.
Sindrome di Lynch di tipo II: oltre al tumore al colon, comprende altre possibili neoplasie a livello:
dell'endometrio,
dell'ovaio, dello stomaco,
del tratto urinario,
dei dotti biliari.
Hereditary
cancer syndrome
Gene(s) for which testing
is available
Mutation detection rate by
sequencing
Lynch syndrome
(HNPCC)
MMR genes: MLH1, MSH2,
MSH6, PMS2
MLH1, MSH2: 85–90%
MSH6: about 7-10%
PMS2: fewer than 5%
Unknown genes ?
EPCAM (not an MMR gene)
inactivate MSH2 in about 1% of
individuals with Lynch syndrome
EPCAM (detected by
duplication/deletion analysis)
(MMR) genes: Mismatch Repair Genes
23
Riparazione delle rotture DSB
NHEJ:
• Predominante nei mammiferi
• Riparazione inaccurata
• Veloce meccanismo di emergenza
• Non dipende dai cromatidi fratelli
• Attiva in G1 e sia in cellule aploidi che diploidi
HR:
• Prevale nelle cellule in fase S e G2
• Riparazione accurata
• Veloce meccanismo di emergenza
24
Una serie di step sono alla base
della risposta ai DSB
ATM
➣ ATM = A-T mutated
➣ Serina-treonina chinasi nucleare
attivata da rotture a doppio
filamento
➣ Fosforila e attiva numerosi
substrati che si accumulano
nei “foci” di riparo (quindi
associati alla riparazione
del danno) o bloccano il
ciclo cellulare: p53, BRCA1.
FANC D2, NBS1, istone H2AX
IF Ab Contro H2AX
• Devono essere percepiti da una molecola che è in grado di
riconoscere le lesioni sul DNA o le alterazioni cromatiniche che
derivano dalla rottura del DNA
• Le estremità rotte devono essere quindi processate
• Vengono attivati i fattori di trasduzione del segnale,
probabilmente attraverso modificazioni post-trasduzionali
• Il segnale viene trasmesso ai fattori “effettori”
• Il trasduttore iniziale e principale è ATM
25
Model of the double-stranded-break response cycle.
(A) Undamaged section of a chromosome,
showing two chromatin loops and inactive
ATM dimer.
(B,C) Induction of a DNA doublestranded- break (DSB), modification of
ATM and recruitment of both ATM and
MRE1/RAD50/NBS1 (MRN).
The possibility that MRN binds before
ATM is shown, but the exact order is
unknown. The thin black line indicates
modified chromatin.
(D,E) A wave of H2AX phosphorylation is
followed by recruitment of mediators
(mediator of DNA damage
checkpoint protein 1 (MDC1), p53-binding
protein 1 (53BP1) and breast-cancerassociated protein 1 (BRCA1)) to the
The molecular architecture of the focus
is unknown.
(F) Disassembly of the focus, ATM inactivation and chromatin remodelling.
The model suggests at least two distinct forms of soluble ATM: an inactive oligomer and an active monomer, and at least
two distinct active insoluble forms: one directly at the lesion and another integral to the growing focus.
Note that the MRN complex is also a component of the growing focus but, for clarity, has been omitted here.
Complex, persistent lesions are thought to be more difficult to repair, and this is reflected in the size attained by
26
the growing focus. The insect contains a colour-coded key to the molecules shown
MRN ha un ruolo fondamentale come sensore del DSBs
e nel processamento e riparazione del danno
Principali ruoli del complesso MRN
Stimola l’attività di DNA ligasi
(MRN): MRE1/RAD50/NBS1
Facilita la ricerca di brevi o lunghe
regioni di omologia
Mantiene vicine le
estremità del DNA
Media la coesione tra un cromatidio
rotto e il cromatidio fratello, per
impedirne la separazione
Rad50 modula l’attività
enzimatica di Mre11
27
Sindromi umane associate a difetti
nella riparazione del DNA
MALATTIA
L’attivazione dei checkpoints è necessaria per la
percezione del danno e l’amplificazione del segnale in
modo da attivare un’appropriata risposta biologica.
SINTOMI
FUNZIONI
ALTERATE
GENE MUTATO
Atassia
Telangiecta
sia (AT)
Difetti coordinazione
Riparazione
muscolare; progress.
replicativa del
atrofia muscolare spinale; DNA
ipersensibilità alle IR e a
agenti chimici; predisp. al
cancro, ritardo mentale,
immunodeficienza
ATM
Anemia di
Fanconi (FA)
Anemia aplastica,
malformazione del cuore,
rene ed arti; leucemia;
rotture cromosomiche
Riparazione
replicativa, dimeri
di pirimidina
indotti da UV;
attività ligasica,
esonucleasica e
trasporto enzimi
della riparazione.
FAA,
FAC
Sindrome di
Bloom (BS)
Difetti di crescita;
malattie dovute a
fotosensibilità;
instabilità cromosomica,
alta incidenza di tumori
Allungamento
delle catene di
DNA nella
replicazione.
BLM
Sindrome di
Werner
(WR)
Progeria, ridardo nella
crescita
Allungamento
delle catene di
DNA nella
replicazione.
(BER)
WRN
Cancro del
colon non
poliposico
(HNPCC)
Tendenza allo sviluppo del Mismatch repair
cancro al colon non
poliposico
hMSH2, hMLH1,
hPMS1, hPMS2
28
Clinical phenotype
✔
✔
✔
✔
✔
✔
✔
✔
✔
Clinical phenotypes of ataxia-telangiectasia (ATM gene mutated)
Cerebellar ataxia: progressive neuronal degeneration
Immunological dysfunction: low Ig and IgE; normal V(D)J recombination
Cancer predisposition: lymphoma and leukaemia; chromosomal translocation
Hypogonadism
Sensitivity to ionizing radiation
Premature aging
Increased alpha-fetoprotein
Small stature
Insulin resistance
Cellular phenotype
DNA damage and repair: chromosomal instability; cell-cycle checkpoint
defects in G1,S (radio-resistant DNA synthesis) and G2/M; sensitivity to
ionizing radiation, increased chromosomal breakages, telomere end-to-end
fusions; DNA repair; subtle, but normal kinetics
✔ Other abnormalities: cytoskeletal defects, abnormalities in the plasma membrane,
an increased number of lysosomes; high levels of trophic factors needed for growth;
defects in intracellular signalling.
✔
Nijmegen Breakage Syndrome (NBS)
Rara malattia autosomica recessiva descritta inizialmente
in pazienti olandesi di origine slava
Mutazione fondatrice 657delta5 nel gene NBS1
Frequenza 1/100000
Frequenza eterozigosi in individui slavi 1/155
(asintomatici)
Caratteristiche fenotipiche:
Fronte ristretta
Viso allungato
Mandibola rientrata
Microcefalia
Orecchie pronunciate
29
Cross-talk tra le sindromi della Riparazione del DNA
Proteine che sono difettive in sindromi simili, presentano interazioni funzionali.
• ATM fosforila NBS, difettivo nella Nijmengen Breakage Sindrome
• ATM fosforila BLM e la fosforilazione di BLM dopo l’arresto della replicazione dipende da ATR
Link funzionali tra pathway di signalling apparentemente separate che possono convergere tutte verso una
unica risposta globale al danno del DNA
LESIONI AL DNA
CHECKPOINT
DA DANNO AL DNA
RIPARAZIONE
ARRESTO DEL
CICLO
CELLULARE
REVERSIONE
EXCISIONE
LETALITÀ
ARRESTO DELLA REPLICAZIONE
DEL DNA
RICOMBINAZIONE
NON
OMOLOGA OMOLOGA
MECCANISMI DI
TOLLERANZA
ALLA LESIONE
RICOMBINAZIONE SINTESI
DEL DNA
OMOLOGA
TRANSLESIONE
MUTAGENESI
MANTENIMENTO
DEI TELOMERI
STABILITÀ GENETICA
30
Risposta SOS
Il danno porta alla distruzione proteolitica di un repressore trascrizionale
b) Stato indotto
a) Stato non indotto
Gene RecA
Gene lexA
Gene lexA
Gene RecA
Proteina Lex A
Proteina RecA
(inattiva)
Repressione della
trascrizione dei geni coinvolti
in diversi processi di
riparazione del DNA
Proteina Lex A
Rec A attivo stimola
la scissione di Lex A
Proteina Lex A scissa
Proteina RecA
attivata dal danno al
DNA
Nessuna repressione, i geni
per la riparazione vengono
trascritti
The SOS response is a
global response to DNA
damage in which the cell
cycle is arrested and DNA
repair and mutagenesis
are induced.
RecA protein is stimulated by ssDNA
31
Riparazione post replicativa mediante
Sintesi translesione
Quando la polimerasi III s’imbatte nella lesione presente sullo
stampo, si dissocia dal DNA, al suo posto subentra la DNA
polimerasi translesione che prosegue la sintesi attraverso il dimero
di timina in modo indipendente dall’appaiamento delle basi, e che
viene poi risostituita della DNA pol III.
Riparazione Post Replicativa mediante
Strand exchange
3’
5’
3’
5’
“Stalling” indotto
dal danno
Gap-formation
mediante “re-priming”
Ricombinazione
omologa
Taglio della
giunzione di Holliday
Dopo la lesione, la replicazione del DNA può riprendere mediante il “re-priming” della sintesi del DNA a valle del sito
di danno, generando un gap nel filamento fratello che può essere ripristinato attraverso la pathway di ricombinazione
omologa.
32
La ricombinazione tra il filamento neosintetizzato nella molecola di DNA contenente il danno e il filamento
complementare della molecola di DNA omologa permette al complesso replicativo di superare il danno.
Deans & West, DNA interstrand crosslink repair and cancer. Nature Reviews Cancer 11, 467-480 (July 2011). doi:10.1038/nrc3088
Suppression of ICL sensitivity by inhibitionof non-homologous end joining.
In wild-type cells, interstrand crosslinks
(ICLs) can be repaired by homologous
recombination (HR) or non-homologous
end joining (NHEJ).
The Fanconi anaemia (FA) pathway,
however, promotes HR‑dependent
stabilization of the replication fork and
DNA repair.
In FA cells, double-strand breaks (DSBs)
are frequently created that can be
repaired by either HR or NHEJ.
Because the DSB is one ended, NHEJ
does not have a natural substrate to rejoin
so these breaks can remain unrepaired
(generating chromatid breaks) or
may ligate with a DSB in a different
chromosome (generating radial
chromosomes).
Suppression of NHEJ by deleting a
component of this pathway or by using
DNA-dependent protein kinase catalytic
subunit (DNA-PKcs) inhibitors promotes
the repair of the DSBs by FA‑independent
pathways of HR (dashed arrow).
33
Deans & West, DNA interstrand crosslink repair and cancer. Nature Reviews Cancer 11, 467-480 (July 2011). doi:10.1038/nrc3088
How Interstrand CrossLinks (ICLs) kill tumour cells.
Interstrand crosslinks (ICLs) can block the progression of the replication fork by inhibiting the
progression of the replisome. ICLs can stall transcription. ICLs may distort the structure of
chromatin and prevent the access of DNA-interacting proteins. Tumour cell death can be
induced by p53- and FAS ligand-dependent apoptosis (programmed cell death) or p53independent mitotic catastrophe (death when apoptosis is absent but vital DNA integrity is lost).
34

![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)




![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)



