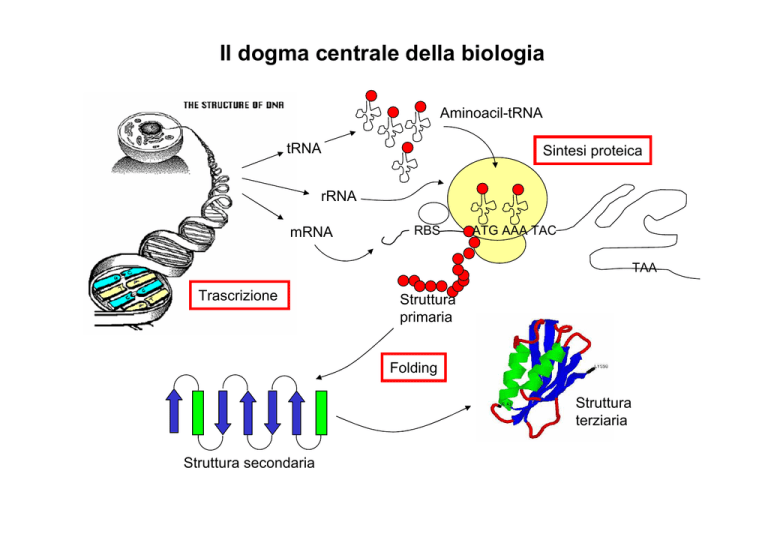
Il dogma centrale della biologia
Aminoacil-tRNA
tRNA
Sintesi proteica
rRNA
mRNA
RBS
ATG AAA TAC
TAA
Trascrizione
Struttura
primaria
Folding
Struttura
terziaria
Struttura secondaria
Il dogma centrale della biologia (rivisto)
Il DNA, avvolto sugli istoni, deve essere reso
accessibile alla polimerasi prima di essere trascritto.
L’mRNA deve essere correttamente modificato (polyA,
CAP) perché sia poi possibile eseguire lo
Splicing, che in tessuti diversi o in momenti diversi può
produrre diverse “varianti” dello steso gene.
La traduzione è un processo “assistito” da molte
proteine che si occupano di proteggere le catene
nascenti.
Solo proteine con la giusta
strutturazione (folding +
modificazioni
post
traduzionali)
sono
funzionanti e non vengono
rimosse.
Ogni proteina è sottoposta
ad un turnover mediato da
apposite strutture cellulari.
Il gene procariotico
Nei procarioti non c’è il nucleo, quindi la
trascrizione e la traduzione possono
avvenire quasi contemporanamente:
questa
caratteristica
influenza
l’
organizzazione del gene procariotico.
Si possono riconoscere 3 regioni importanti:
1. Promotore: regioni a monte delle sequenza codificante su cui si legano fattori di
trascrizione e di repressione e determinano l’arrivo della RNA polimerasi.
2. Regioni non codificanti (UTR): posti a monte (5’) e a valle (3’) del gene, lo delimitano e
contengono i siti di attacco dei ribosomi (RBS) e i segnali per terminare la trascrizione.
3. Regione codificante, dove è contenuta l’informazione per la proteina.
Il gene eucariotico
Diversamente dai procarioti, l’organizzazione di un gene eucariotico è molto più complessa:
1. Esoni: porzioni del gene che contengono la sequenza codificante per la proteina, sono
molto piccoli rispetto al gene intero.
2. Introni: porzioni del gene che interrompono gli esoni e che non contengono
informazioni per la codifica. Costituiscono la maggior parte del gene.
3. TATA box: quattro basi canonioche a cui si lega la RNA polimerasi II e da cui inizia a
svolgere il DNA per trascriverlo.
4. Promotori: regioni a monte delle sequenza codificante a cui si legano i fattori di
trascrizione, che a loro volta chiamano la RNA polimerasi.
5. Enhancers: posti a monte e/o a valle del gene, potenziano/favoriscono l’azione dei
promotori guidando dei particolari ripiegamenti nel DNA.
Trascritto primario e splicing
Lo splicing avviene dopo che un gene è stato trascritto. Il trascritto primario è molto più lungo di
quanto serve perché contiene regioni non codificanti (introni) che vanno rimosse in modo da
mettere insieme quelle codificanti (esoni). Il complesso detto spliceasoma si occupa di questo
processo “taglia e cuci” riconoscendo sequenze specifiche sull’RNA (GT al 5’ e AG al 3’).
Strutture degli RNA
L’RNA, essendo composto da 2 copie di basi complementari come il DNA, non si trova quasi
mai nello stato di singolo filamento ma forma strutture secondarie con funzioni biologiche
basilari.
l’RNA ribosomiale assume una struttura
che guida l’assemblaggio del ribosoma,
chiama a sé le proteine ribosomiali e
costituisce il punto di aggancio per la
formazione del complesso di inizio
traduzione.
l’RNA transfer si struttura a trifoglio e in
base alla struttura assunta viene
riconosciuto da diverse aminoacil-tRNA
sintetasi che guidano la formazione del
legame ad alta energia tra Aminoacido e
RNA, fondamentale per la sintesi
proteica.
Il codice genetico
Si caratterizza come
- UNIVERSALE (…)
- DEGENERATO
- RIDONDANTE
E’ composto da 64 diversi
codoni che codificano i 20
amino acidi.
La tebella accanto mostra
il sistema di decodifica guidato
dal dal ribosoma.
Sono evidenti le
degenerazioni a carico delle
basi 2 e 3 e la ridondanza
complessiva.
Alanine
Ala
A
GC[CATG]
Cysteine
Cys
C
TG[CT]
Aspartic AciD
Asp
D
GA[CT]
Glutamic Acid
Glu
E
GA[AG]
Phenylalanine
Phe
F
TT[CT]
Glycine
Gly
G
GG[CATG]
Histidine
His
H
CA[CT]
Isoleucine
Ile
I
AT[CAT]
Lysine
Lys
K
AA[AG]
Leucine
Leu
L
CT[CATG], TT[AG]
Methionine
Met
M
ATG
AsparagiNe
Asn
N
AA[CT]
Proline
Pro
P
CC[CATG]
Glutamine
Gln
Q
CA[AG]
ARginine
Arg
R
CG[CATG], AG[AG]
Serine
Ser
S
TC[CTAG], AG[CT]
Threonine
Thr
T
AC[CATG]
Valine
Val
V
GT[CATG]
Tryptophan
Trp
W
TGG
TYrosine
Tyr
Y
TA[CT]
-
-
TA[AG], TGA
STOP
Gli amino acidi e il legame peptidico
Agli amino acidi sono composti organici chirali che
presentano almeno un gruppo carbossilico (-COOH) a
funzione acida e un gruppo aminico (-NH2) a funzione
basica.
Le proteine sono composte soltanto da α-amino acidi,
legati tra loro mediante legami amidici detti PEPTIDICI
che si instaurano tra il gruppo α-aminico e il gruppo αcarbossilico.
Ciò che diversifica i vari amino acidi
è la catena laterale legata al
carbonio α, che può conferire
all’amino
acido
caratteristiche
chimico-fisiche diverse.
Classi di amino acidi
Acidi
Polari, non carichi
Strutturali
Basici
Idrofobici
Aromatici
Folding delle proteine
I polimeri di α-amino acidi (le proteine) sono influenzati dalle caratteristiche chimico-fisiche
delle catene laterali e, in base a principi di interazioni deboli di tipo idrofobico o
elettrostatico, si possono ripiegare, fino a raggiungere la minor energia termodinamica.
Questo processo, denominato FOLDING, è alla base del
funzionamento delle proteine, visto che solo se sono
correttamente strutturate esse assumeranno la loro forma e
soprattutto la loro FUNZIONE definitiva.
Importante
La strutturazione delle proteine dipende principalmente
dalla sequenza dei residui che la compongono, oltre che
dall’ambiente in cui si strutturano
Il backbone delle proteine
Il legame peptidico ha delle caratteristiche di doppio legame e costringe i due atomi adiacenti
C=O e N-H a giacere sullo stesso piano.
La rotazione della molecola può avvenire solo intorno al carbonio α, ma non tutti gli angoli
di rotazione sono permessi a causa degli ingombri sterici delle diverse catene laterali e dello
scheletro stesso.
Struttura secondaria delle proteine
Il legame peptidico genera una polarità negli scheletri proteici per cui
si vengono a formare PONTI IDROGENO tra i gruppi amidici e i
gruppi carbonilici di aminoacidi diversi. Queste interazioni deboli
portano la struttura primaria della proteina (la sequenza dei suoi
residui) a ripiegarsi in una STRUTTURA SECONDARIA in cui sono
riconoscibili due formazioni
Alfa elica
Beta-strand
struttura compatta e avvolta in cui i legami
idrogeno sono disposti parallelamente allo
scheletro, stabilizzando aminoacidi vicini tra
loro.
struttura rilassata in cui i ponti idrogeno si
stabiliscono tra catene adiacenti che
possono essere parallele o antiparallele, a
formare i foglietti β
Il plot di Ramachandran
Nel 1963 GN Ramachandran, utilizzando un modello a sfere, gettò
le basi che permisero di interpretare la stereochimica delle proteine.
Egli propose di descrivere gli aminoacidi di una proteina in termini di
angoli torsionali del backbone: queste rotazioni dei vari angoli φ e ψ
non potevano essere arbitrarie, ma dovevano seguire delle logiche
di ingombro sterico delle catene laterali.
Dimostrò quindi che le strutture secondarie avevano tutte un
inquadramento preciso in termini di φ e ψ.
Elica α:
Foglietto β:
Φ
tra - 40° e - 100°
Ψ
tra
Φ
tra - 80° e - 120°
Ψ
tra 120° e
-40° e
-65°
170°
Regioni ordinate e regioni non ordinate
Per ottenere un ripiegamento corretto le varie
strutture secondarie si collegano tra loro
mediante sequenze in cui non ci sono ponti
idrogeno fissi e spesso nemmeno altri tipi di
interazioni intra-molecolari, definiti “random coil”,
o strutture disordinate.
In certe proteine però esistono adattamenti
strutturali specifici che contribuiscono in modo
decisivo alla strutturazione delle proteina e
quindi alla sua funzione definitiva: sono i
cosiddetti “loop” e sono stati classificati in base
al tipo di struttura secondaria che congiungono
in
alpha-alpha,
alpha-beta,
beta-beta links e beta-hairpins.
beta-alpha,
http://www.bmm.icnet.uk/loop/index.html
Strutturazioni successive
Congiungendosi, le strutture secondarie formano motivi comuni e conservati che
prendono il nome di strutture supersecondarie, dette anche MOTIVI. Tali strutture si
organizzano poi a dare le vere strutture terziarie e quaste, assemblandosi, formano le
strutture quaternarie.
β-turn-β
up-down
greca
jelly-roll
α-turn-α
super-barrel
coiled-coil
β-α-β
fold di
Rossmann
four helix
bundle
Esempi di strutture terziarie
Dominio singolo
Dominio triplo
Dominio doppio
Dominio quadruplo
Strutture quaternarie e simmetrie
Targeting delle proteine
Denaturazione del DNA: la temperatura di melting
3’-tacgacactacgactacagacgaatactacgacatacagacgactag-5’
5’-atgctgtgatgctgatgtctgcttatgatgctgtatgtctgctgatc-3’
c = 13
g = 8
a = 20
t = 7
tot = 48
tot GC = 21 (44.75%)
tot AT = 27 (56.25 %)
Tm = 74.11
Se scaldo una soluzione contenente una
molecola di DNA come quella scritta sopra
fino a 75 °C, metà dei doppi filamenti saranno
aperti. Usando il calore per svolgere il DNA
simulo l’azione delle elicasi e delle
topoisomerasi.
Tm°C = 2 × AT + 4 × GC
Tm°C = 69.3 + (0.41*GC%) – (650/N)
Alcune formule per
calcolare la Tm
I primers - inneschi 3’-OH
Supponiamo di voler amplificare un tratto di DNA con questa sequenza
3’-tacgacactacgactacagacgaatactacgacatacagacgactagtacgacatacagacgactag-5’
5’-atgctgtgatgctgatgtctgcttatgatgctgtatgtctgctgatcatgctgtatgtctgctgatc-3’
Devo creare due sequenze che si “fronteggiano” e che:
1. si leghino allo stampo (quindi devono essere complementari al DNA da copiare)
2. forniscano un 3’-OH libero nella direzione di interesse.
3’-tacgacactacgactacagacgaatactacgacatacagacgactagtacgacatacagacgactag-5’
||||||||||||||
5’-atgctgtgatgctg-3’
chiamiamolo primer diretto
chiamiamolo primer inverso
3’-tacagacgactag-5’
|||||||||||||
5’-atgctgtgatgctgatgtctgcttatgatgctgtatgtctgctgatcatgctgtatgtctgctgatc-3’
Per “disegnare” dei primers bisogna conoscere la sequenza che si vuole amplificare, e per
far questo si guardano le banche dati di DNA, cioè collezioni tratti di DNA ottenuti per
esempio dai sequenziamenti genomici.
Ottenuta la sequenza si procede alla produzione (oggi si acquistano) degli stessi.
Per ogni primer che si disegna bisogna calcolare la temperatura di melting (quindi sapere
lunghezza e composizione in basi): primer diretto e inverso infatti devono avere Tm molto
simili!
Enzimi di restrizione
Esistono tre tipi di enzimi di restrizione:
Tipo I. Riconoscono una sequenza specifica ma tagliano lontano da essa.
Tipo II. Riconoscono una sequenza specifica, spesso palindromica, e tagliano al suo interno.
Tipo III: Proprietà intermedie.
Gli enzimi di tipo II sono i più
usati.
Ne sono stati caratteizzati circa
1000 e sono commercialmente
disponibili.
La nomenclatura si basa su:
EcoRI
EcoRV
Organismo
HindIII
Ordine di
Codice
scoperta
aggiuntivo
Il clonaggio di frammenti di DNA
Il clonaggio di un gene di interesse
rappresenta oggi la tecnica di routine sia
per il suo sequenziamento sia per lo studio
delle sue funzioni.
Gli step da seguire sono:
0. Ottenimento del DNA
1. Taglio del DNA con ER.
2. Taglio di un plasmide con ER.
3. Incubazione dei due tagliati.
4. Aggiunta di ligasi
5. Inserimento in batteri
Se il sito di clonaggio si trova all’interno di una
sequenza codificante (es. il gene LacZ),
l’inserimento del gene provoca la perdita di quella
funzione.
LacZ è in grado, se c’è, di reagire con un composto
aggiunto nel terreno e dare colorazione blu.
=> Posso riconoscere i batteri con inserto.
Librerie di DNA
Sono collezioni di frammenti di DNA ottenute per esempio dalla digestione con un enzima di
restrizione di un intero genoma (librerie genomiche): se taglio anche il plasmide con lo stesso
enzima, i frammenti e il plasmide si richiuderanno uno sull’altro. I plasmidi possono poi essere
introdotti nei batteri e fatti moltiplicare.
Possono essere anche create librerie di cDNA: in questo modo nella libreria avrò i geni già
maturati, quindi più piccoli e più gestibili.
Sequenziamento del DNA
La possibilità di sequenziare il DNA è stata, una delle principali rivoluzioni per l’ingegneria
genetica: fino ad allora si era solo in grado di lavorare sui caratteri fenotipici dei geni, non
avendo modo di conoscere la loro seqerunza.
La tecnica oggi utilizzata, detta “ad interruzione di catena” fu introdotta nel 1975 da Frederick
Sanger e sfrutta la capacità della DNA polimerasi di polimerizzare nucleotidi su DNA
stampo ma solo se presente un gruppo 3’OH libero. Fondamentali sono dei nucleotidi
modificati detti di-deossi:
- nel ribosio i carboni 2 e 3 portano dei gruppi OH
- nel desosiribosio il gruppo OH in posizione 2 è assente
- del didesossiribosio anche il groppo 3-OH è rimosso
=> un dideossi-nucleotide inserito dalla polimerasi impedisce
l’allungamento della catena
ribosio
desossiribosio
didesossiribosio
DNA Microarray
E’ una tecnica relativamente nuova che permette il monitoraggio dell’espressione genica di
tutti i geni di un genoma in un singolo esperimento.
Sintesi in situ di sonde
La fotolitografia permette la costruzione di array con alto contenuto di informazione
permettendo di avere 500,000 probe in uno spazio di 1.28 cm2. Ogni singolo probe è
costituito da milioni di oligonucleotidi identici, e un singolo array di 1.28 cm2 contiene probe
set per circa 40,000 geni umani.








![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)



