LA REPLICAZIONE DEL DNA
Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria
Giovanna Attene
Replicazione semiconservativa del DNA
secondo il modello di Watson e Crick (1953)
Replicazione di cromosoma circolare nei procarioti
Una sola origine della
replicazione (OriC,
complesso di preinnesco)
Dimensione media del
cromosoma procariotico:
3 milioni di paia di basi
Avvio della
denaturazione della
doppia elica
Bolla di replicazione
Forcella di replicazione
Replicazione bidirezionale
Rotazione intorno
all’asse
Nei procarioti anche
meccanismo di
replicazione a sigma (σ)
Meccanismo
di replicazione
a theta (θ)
Replicazione a bolle negli eucarioti
Da alcune centinaia di ‘repliconi’ (500 in S. cerevisiae) a diverse
migliaia (35000 in V. faba)
Repliconi
Velocità di replicazione del DNA in
procarioti ed eucarioti
N. repliconi
Lunghezza
media (kb)
Movimento
forcella (bp/min)
1
4200
50000
3500
40
2600
X. laevis
15000
200
500
M. musculus
25000
150
2200
Organismo
E. coli
D. melanogaster
Esperimento di Kornberg (1957)
sintesi del DNA in vitro
estratto
proteico
dNTP (ATP+nucleosidi)
marcati con 32P
Estratto
acellulare
Catena
polinucleotidica
DNA polimerasi
DNA stampo
Deossinucleosidi trifosfati
marcati con 32P
DNA neosintetizzato
contenente nucleotidi
marcati con 32P in un
solo filamento
Assemblaggio dei nucleotidi nel filamento
di DNA in formazione
Desossinucleoside
5’-trifosfato
libero
Filamento in
crescita
DNA stampo
Desossiribosio
Antiparallelismo delle due eliche e
direzione di avanzamento della DNA polimerasi
-
OH
Estremità 3’
OPO3
Estremità 5’
Estremità 3’
Estremità 5’
Entrambi i filamenti
decorrono in
direzione 5’Æ3’
essi sono antiparalleli
OH
Estremità 3’
OPO3Estremità 5’
Estremità 3’
Estremità 5’
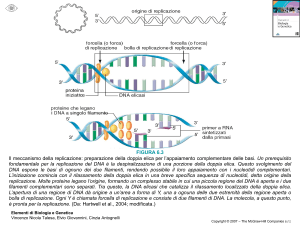
Ipotesi replicazione ad X e a Y
Forma a “X”
Forma a “Y”
Sintesi del primer e avvio della replicazione
Primasi
1 - L’RNA primasi
sintetizza una
corta molecola di
RNA primer.
DNA stampo
RNA iniziatore
2 - La DNA
polimerasi III
si lega al
primer ed inizia a
catalizzare la
sintesi di
nuovo DNA
Polimerasi III
Nuovo DNA
Biochimismo della replicazione nei procarioti (1)
DNA polimerasi III
L’elicasi provvede allo srotolamento della
doppia elica, denaturando il DNA e formando
la forcella di replicazione
Crescita continua
Filamentio anticipato
(leading strand)
Filamento ritardato
(lagging strand)
Crescita
discontinua
Topoisomerasi e girasi
RNA
primer
Proteine ssb, stabilizzatrici
dei singoli filamenti
RNA
primasi
Biochimismo della replicazione nei procarioti (2)
Tempo
1°
2°...
Biochimismo della replicazione nei procarioti (3)
Sintesi e assemblaggio dei frammenti di Okazaki (Lagging strand)
Filamento
ritardato
RNA primer
La primasi sintetizza l’RNA primer
RNA iniziatore
DNA polimerasi III
Frammento di Okazaki
RNA primasi
La DNA polimerasi III aggiunge
nucleotidi al nuovo frammento in
direzione 5’Æ3’ fino ad incontrare il
primer del frammento di Okazaki
precedente
DNA polimerasi I
La DNA polimerasi I rimuove l’RNA
primer sostituendolo con DNA
DNA ligasi
Frammenti di Okazaki:
1000-2000 nucleotidi
nei procarioti
100-200 nucleotidi
negli eucarioti
La DNA ligasi catalizza la
formazione del legame
fosfodiesterico che unisce
due frammenti di Okazaki
Meccanismi di riparazione del DNA
Attività esonucleasica delle DNA polimerasi
DNA polimerasi negli eucarioti
Negli eucarioti le diverse DNA polimerasi sono indicate come α, β, γ, δ, ε,
con localizzazioni (nucleo, citoplasma) diverse e funzioni diverse
DNA polimerasi
(mammiferi)
α
β
γ
δ
ε
Localizzazione
nella cellula
Nucleo
Nucleo
Mitocondri
Nucleo
Nucleo
Attività
esonucleasica
(3’Æ5’)
No
No
Si
Si
Si
Funzione
Replicazione
DNA
Riparazione
DNA
Replicazione
DNA
Replicazione
DNA
Riparazione
DNA




![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)



![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)