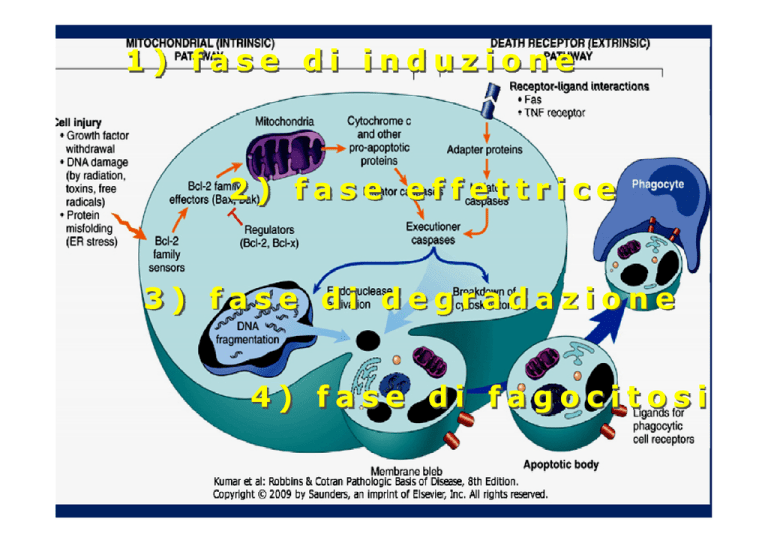
1) fase di induzione
2) fase effettrice
3) fase di degradazione
4) fase di fagocitosi
Annexin V
FITC
3) fase di degradazione
Elettroforesi del DNA di cellule in apoptosi
A- DNA ad alto peso molecolare di
cellule integre
B- DNA di cellule in apoptosi: si può
osservare il ladder(scaletta), formato da
frammenti di peso molecolare discreto
(multipli di nucleosomi)
C- DNA di cellule in necrosi: i frammenti
sono di peso molecolare vario, per lo più
piccolo (striscio continuo)
CROMATINA IN UN NUCLEO INTERFASICO
Matrice
proteica
Fibrille di
cromatina
ciascuna spira
di avvolgimento
è costituita da
6 nucleosomi
la doppia elica
del DNA si
avvolge due
volte attorno
al nucleosoma
Ogni fibrilla di
cromatina è costituita
da un solenoide compatto
il taglio a livello
del DNA “linker”
produce frammenti
multipli di 180 bp
La frammentazione del DNA, prodotta dalle endonucleasi
attivate dalle caspasi durante il processo apoptotico, ha
permesso di mettere a punto una tecnologia basata sul
principio secondo il quale le terminazioni libere 3’-OH che
si vengono a formare in seguito alla frammentazione del
DNA possano servire come substrato per la terminal
deossinucleotidil transferasi (TdT).
Utilizzando come substrato dUTP marcato è possibile
distinguere in una popolazione le cellule apoptotiche.
La TdT catalizza l'addizione di dUTP marcato all'estremità
3'-OH dei frammenti di DNA. Questo metodo che è detto
TUNEL
acronimo di “TdT mediato dUTP nick end
labelling” , ha permesso la visualizzazione in situ della
frammentazione del DNA a livello di singole cellule.
Apoptotic in situ DNA
strand breaks
Microscopio a fluorescenza
Microscopio ottico
citometria di flusso


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)





![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)
