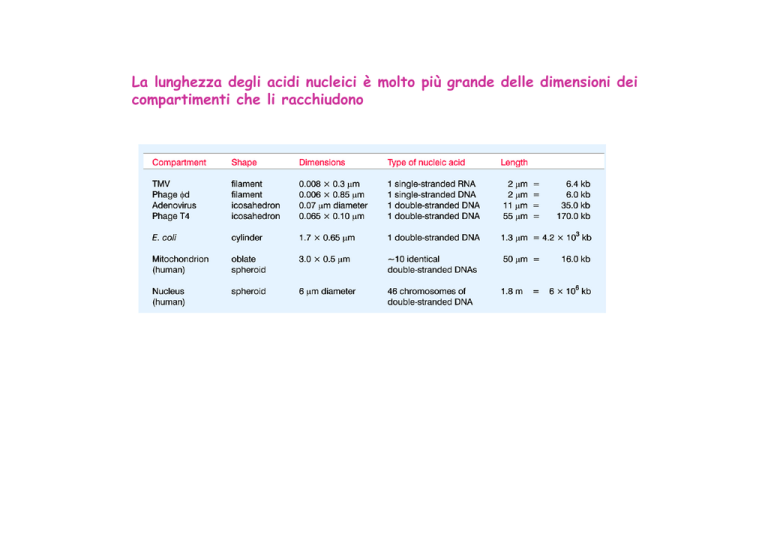
La lunghezza degli acidi nucleici è molto più grande delle dimensioni dei
compartimenti che li racchiudono
ASSEMBLAGGIO DEL VIRUS TMV (Mosaico del tabacco)
Ha 1 molecola di RNA a singolo
filamento della lunghezza di
2mM
La posizione dell’RNA dentro il capside
è determinata dal legame dell’acido
nucleico con le proteine.
Viene assemblato in una forma
filamentosa o a bastoncino
L’acido nucleico viene ad associarsi
progressivamente seguendo un
andamento a spirale con le proteine
del rivestimento.
Parte da una forcina di nucleazione
e prosegue in maniera bidirezionale.
MATURAZIONE DEL FAGO λ
Molto simile al fagoT4
ha un capside icosaedrico
1 molecola di DNA a doppio filamento
della lunghezza di 55µ
µM
Il capside viene prima assemblato
e poi riempito con l’acido nucleico
Traslocazione e condensazione sono
entrambi sfavorevoli dal punto di vista
energetico.
La traslocazione implica l’idrolisi dell’ATP.
Della condensazione si sa molto poco, forse
coinvolge proteine di impalcatura all’interno
del capside.
Il cromosoma dei batteri si chiama
NUCLEOIDE
Questa struttura non presenta un fuso
mitotico e quindi in seguito alla replicazione
la separazione dei due cromosomi si
avvale dell’attacco alla membrana cellulare
Nucleoide di E. coli che fuoriesce
da una cellula lisata
La struttura ad anse del nucleoide
batterico assicura l’efficiente
condensazione del DNA all’interno
della piccola cellula.
La lunghezza media di un’ansa è
di 40 Kb.
Come siano organizzate le proteine
che tengono insieme i domini e
come interagiscono con il DNA
non è stato del tutto chiarito
Proteine che legano il NUCLEOIDE di E. coli
HU: Condensa il DNA
H1: lega sequenze curve
P: ha proprietà di legame al DNA
Compattamento Nucleo-cromosoma
+ 2M NaCl
histones
radial loop
chrosomosome model
Mitosi
chromatid 1µm
mitotic chromosome
10 µm
DNA loops
+ 2M NaCl
istoni
chromatid
compact size
DNA
length
compaction
nucleus (human)
2 x 23 = 46 chromosomes
92 DNA molecules
10 µm ball
12,000 Mbp
4 m DNA
400,000 x
mitotic chromosome
2 chromatids, 1 µm thick
2 DNA molecules
10 µm long X
2x 130 Mbp
2x 43 mm DNA
10,000 x
DNA domain
anchored DNA loop
1 replicon ?
60 nm x 0.5 µm
60 kbp
20 µm DNA
35 x
Compaction by chromosome scaffold / nuclear matrix
Livelli di compattamento della cromatina
10
1
80
6-7
1,200
-40
60,000
680
~1.1x106 1.2x104
18 loops/ 1.2x104
miniband
Ogni ansa è formata da una fibra che
può contenere sia regioni da 10nm
(meno condensate, più accessibili) sia
regioni da 30nm (più condensate, meno
accessibili)
STRUTTURA DEL NUCLEOSOMA
L’unità di base della struttura cromatinica.
Primo Livello: Fibra da 10 nm
ISTONI
H1
Linker histone
proteine piccole,
basiche altamente
conservate,
H2A
H2B
helix
Core histones
variable
H3
H4
conserved
N
Histone acetylation
is a reversible modification
of lysines in the N-termini
of the core histones.
Result:
• reduced binding to DNA
• destabilization of chromatin
Elettroforesi degli Istoni
SDS
AU
-
+
AUT
+
++
+
TTTT
elettrodo:
taglia + carica +
idrofobicità
taglia + carica
Separazione: Taglia
T
TT
T
H3.1
T
135aa
++
+
++
+
T
T
4
135 aa
H3
H4
H4
++
+
H4
H3
0
++
+
4
3
2
1
0
H4
102aa
102 aa
+
H3.2
T
-
H4 TT
T
1
H3.1
T
135aa
135aa 32
H3
elettrodo:
H3.2
TT
T
H3
34
2
10
4
3
12
0
4
3
2
1
0
H4
102aa
-
Figure 13.1
La Nucleasi Micrococcale taglia il DNA su entrambi i filamenti
Questo enzima non taglia sul nucleosoma ma solo
tra particelle adiacenti. Si possono ottenere digesti parziali
Dopo la digestione gli Istoni
vengono rimossi
I Prodotti vengono analizzati
tramite elettroforesi
Digestione spinta con Nucleasi Micrococcale
180-200bp
Valore medio
154bp (fungo)
Oscillazioni tra varie
260bp (riccio di mare) Specie
146bp
Nucleo Centrale - Invariante
8-114bp
DNA Linker - Variabile
Attraverso il dominio histone-fold
gli istoni prendono contatti a
formare il core globulare.
Le code N-terminali protrudono
all’esterno dove prenderanno contatto
con lo scheletro di fosfato del DNA
Caratteristiche generali del
nucleosoma
Assemblaggio del nucleosoma:
Tetramero H3/H4 e dimeri H2A/H2B
si formano indipendentemente
Il tetramero H3/H4 si lega al DNA
I dimeri H2A/H2B si assemblano per ultimi
Modello derivante dalla cristallografia del nucleosoma
H2A: rosso
H2B: giallo
H3: viola
H4: verde
Struttura cristallografica del nucleosoma alla risoluzione di 8Å
Luger, Mader, Richmond, Sargent & Richmond
Nature 389, 251-260 (1997)
Rotazione
di
90°
Rotazione
di
90°
Il nucleosoma presenta una simmetria diadica
Le regioni histone-fold del tetramero interagiscono con le 60bp centrali
Le regioni N-terminali interagiscono con i punti di entrata e di uscita del DNA
Ciascun dimero H2A/H2B occupa le 30bp adiacenti alle 60 occupate dal tetramero
Le code istoniche N-terminali prendono contatto con lo scheletro di fosfato
protrudendo in posizioni specifiche
Il DNA ricco in AT ha tendenza a curvarsi meglio in direzione del solco minore
Invece le sequenza ricche in GC tendono a esporre il solco minore verso l’esterno
Posizionamento Traduzionale
Posizionamento rispetto ad un
elemento di sequenza.
La particolare sequenza può
essere inclusa o esclusa dal
nucleosoma
Posizionamento Traslazionale
Posizionamento rispetto alla
fase dell’elica.
La sequenza può trovarsi rivolta
verso l’interno o verso l‘esterno
sulla superficie del nucleosoma
Il Posizionamento dei nucleosomi presenta risvolti importanti per quanto
riguarda la trascrizione genica
Proteine che posizionano i nucleosomi
possono formare regioni nucleosome
free.
Proteine che legano fortemente il
nucleosoma possono posizionarlo
preferenzialmente in un sito
Secondo Livello: Fibra da 30nm
COME SI PASSA DALLA FIBRA DA 10nm ALLA FIBRA DA 30nm?
Figure 13.6
Cromatosoma
C
N
H1
H3
1 mM
H3
5 mM
Il complesso di un nucleosoma con l’istone H1
è chiamato “cromatosoma” e contiene 168bp
di DNA + ottamero istonico + istone linker H1
L’istone linker verosimilmente dirige il
posizionamento relativo di nucleosomi successivi
e il profilo dei contatti nucleosoma-nucleosoma
Zhou, Gerchman, Ramakrishnan, Travers, An, Leuba, van Holde, Zlatanova
Muyldermans Nature 395, 402 (1998)
PNAS 95, 3396 (1998)
Accorciamento della lunghezza del DNA
associato al nucleosoma
L’istone H1 è esterno al nucleosoma e lega il DNA nei punti di entrata e uscita
La parte globulare lega sia un tratto di DNA linker che una porzione centrale associata al core istonico.
L’istone H1 contribuisce anche alla formazione delle strutture di ordine superiore:
la fibra da 30 nm.
Emergono due possibili classi di modelli per la fibra da 30nm:
1) Elica solenoidale (tecnicamente definita one-start), in cui i nucleosomi
sono avvolti in maniera lineare.
Evidenze a favore di questo modello:
a) Diametro invariante rispetto alla lunghezza del DNA linker
b) Aumentato compattamento per fibre con 6 o più nucleosomi
c) Necessità di un DNA linker superavvolto
(Robinson et al., 2006)
2) Nastro a zig-zag (definito two-start), in cui il nastro si piega e si
superavvolge.
Evidenze a favore di questo modello:
a) Variazione del diametro della fibra al variare del linker
b) Percorso a zig-zag dei nucleosomi
c) DNA linker non strettamente piegato
(Schalch et al., 2005)
One-start: modello solenoidale della fibra da 30nm
Polinucleosomi ricostituiti con lunghezze
variabili del DNA linker
Condizioni saline fisiologiche
Presenza dell’istone H1
Tecnica della microscopia elettronica
L’approccio non fornisce informazioni precise
sul percorso del DNA linker ma la costanza
delle dimensioni suggerisce che esso sia
ripiegato all’interno della fibra in maniera
continua e regolare.
La compattazione è migliore in presenza
dell’istone linker
Two-start helix: modello a zig-zag della fibra da 30nm
Tetranucleosoma ricostituito analizzato con la
diffrazone alla risoluzione di 9Å
LS: linker dritto
LB: linker piegato
La presenza di due differenti conformazioni
del linker suggerisce che il dinucleosoma
possa rappresentare meglio l’unità
ripetitiva della struttura di ordine superiore
Le analisi su questa struttura favoriscono
il modello a zig-zag per il DNA linker.
Non definisce chiaramente il ruolo
dell’istone linker.
Stabilizzazione della fibra da 30nm attraverso le code istoniche N-terminali
Effetto barriera del nucleosoma sull’interazione
DNA-proteine
Esiste un meccanismo di competizione
tra la formazione del nucleosoma e l’attacco
di proteine che legano il DNA
DNA 5S + TFIIIA = Trascrizione
Istoni aggiunti dopo NON formano il nucleosoma
DNA 5S + istoni = formazione del nucleosoma
TFIIIA aggiunto dopo = Niente trascrizione
Il Nucleosoma può bloccare l’accesso all’apparato trascrizionale
Esperimenti di Roeder mostrano che il nucleosoma può bloccare TBP,
fattori e polimerasi II. DNA usato è quello di Adenovirus.
TATA
TATA
Nucleosomi aggiunti prima
di TFIID, fattori e pol II
TATA
+
II
Fattori e pol II aggiunti
prima, poi gli istoni
+
TATA
Nessuna trascrizione
Trascrizione
Questo può avvenire in seguito alla replicazione
II
Dinamicità della cromatina
Nonostante la stabilità del nucleosoma e dell’alto grado di compattezza nel nucleo
la cromatina è sorprendentemente dinamica.
Esistono vari modi di modificare la cromatina e rispondere alle necessità della
cellula di regolare i processi biologici dall’espressione genica, alla riparazione
del DNA, alla replicazione e ricombinazione.
1) Uso di varianti istoniche, in particolare varianti di H2A e H3.
2) Modificazioni epigenetiche degli istoni: acetilazione, metilazione ed altre.
3) Uso di sistemi di rimodellamento che cambiano la stabilità e/o la posizione
del nucleosoma.
VARIANTI ISTONICHE
S viene fosforilata in seguito a
danni al DNA
§: differenze in questo loop
prevengono l’assemblaggio di un nuc.
contenente H2A e H2A.Z
§
#
#: questo dominio contatta (H3-H4)2
e variazioni qui influenzano la
stabilità del nucleosoma.
Le varianti istoniche sono coinvolte in vari processi, sia nella espressione genica, sia
nella riparazione del DNA, sia nella definizione di strutture eterocromatiche.
Modificazioni delle code Istoniche N-terminali
RUOLO DELL’ACETILAZIONE ISTONICA
HAT
HDAC
•Decondensa la fibra da 30nm
•Favorisce l’accesso dei fattori di trascrizione sulla cromatina
•Agisce come segnale per il legame di proteine non istoniche
Residui acetilati
interagiscono con
il Bromodominio
Residui metilati
interagiscono con
il Cromodominio
Modificazioni covalenti delle code ammino-terminali
H3
R2
H4
S1
R3
H2A
H2B
K4
K9 S10
K5
K5
S1 E3
Metilazione
K8
K14
K12
K9
R17 K18
K16
K23
R26K27 S28 K79
K20
K119
K12
K15
K20
Acetilazione
Poly-ADP ribosilazione
K24
K120
Fosforilazione
Ubiquitinazione
IL CODICE ISTONICO
Quasi sempre le modificazioni istoniche sono evidenziabili in combinazione.
La natura combinatoriale delle modificazioni istoniche rivela l’esistenza di una
sorta di CODICE ISTONICO (“histone code”) che impone le caratteristiche
regolative di un gene, estendendo l’informazione potenziale del codice genetico.
Questi
stati epigenetici devono essere stabiliti, mantenuti ed ereditati
attraverso mitosi e meiosi (MEMORIA CELLULARE).
Il fenotipo è però sempre VARIEGATO, poiché possono avvenire repentini
cambiamenti di stato.
Le regole del codice istonico
•Modificazioni sullo stesso e/o su differenti istoni possono essere interdipendenti
e generare varie combinazioni su qualunque nucleosoma
•Distinte modificazioni delle code istoniche inducono interazioni con diverse
proteine associate alla cromatina:
•Diversi tipi di struttura di ordine superiore dipendono dalla
concentrazione locale e dalla combinazione di nucleosomi modificati
differenzialmente
In pratica le modificazioni
istoniche servono a
definire particolari stati
della cromatina.


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)





![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)


