
Corso di Lingua Inglese Si ricorda a tutti gli studenti del 3° anno (immatricolati nell aa 2014/2015) che
per potersi laureare dovranno superare la prova di idoneità in Lingua Inglese. Il Corso di Lingua inglese è tenuto dalla Prof.ssa Maria Cristina Grande e si
svolgerà il Venerdi dalle 15-17 in Aula Pasquini Edificio di Zoologia.
[email protected]
Il CLONAGGIO consiste nella moltiplicazione
frammento di DNA appartenente ad un dato genoma.
di un
Ciò è possibile grazie a delle endonucleasi che provocano delle
rotture interne a doppio filamento sul DNA in corrispondenza
di specifiche sequenze nucleotidiche (Enzimi di Restrizione) e a
una piccola molecola di DNA che serve da carrier (Vettore di
Clonaggio).
Successivamente si avrà la replicazione di questo DNA
ricombinante (Clone) milioni di volte fornendo un gran numero
di copie del clone stesso.
Il risultato è l amplificazione selettiva di quel determinato
frammento di DNA
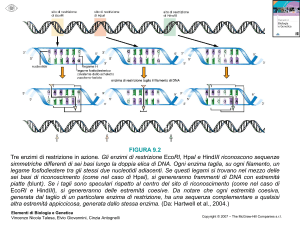
ER di classe II
Esistono tre classi principali di ER: quelli di tipo I, di tipo II e di tipo
III.
tipo I: attività di restrizione e metilazione sulla stessa molecola.
Tagliano il DNA in modo casuale lontano dal sito di riconoscimento.
tipo III: attività di restrizione e metilazione su sub-unità diverse. Non
garantiscono sufficiente specificità di taglio.
Nessuna delle due classi viene utilizzata in biologia molecolare a
causa della loro aspecificità di taglio.
Gli enzimi di classe II, invece, portano le due attività su molecole
distinte e sono caratterizzati da una elevata specificità di taglio.
Enzimi di restrizione
La scoperta degli enzimi di restrizione, avvenuta negli anni 70,
è valsa ai suoi scopritori, Arber, Nathans e Smith, il
conferimento del premio Nobel per la Fisiologia e Medicina.
Gli ER riconoscono di solito sequenze palindromiche,
tagliandole in posizioni specifiche. Una palindrome è una parola
che si legge allo stesso modo sia da destra che da sinistra, per es.
la parola radar oppure ala. Un sito di riconoscimento
palindromico è una sequenza in cui il filamento superiore e
inferiore, letti in direzione 5 -3 , sono uguali. Per es. la
sequenza:
5 -GAATTC-3
3 -CTTAAG-5
Gli ER furono scoperti studiando il fenomeno della restrizione-modificazione.
L introduzione in E.coli di DNA esogeno risultava nella sua rapida
frammentazione in piccoli frammenti (restrizione). L analisi di un DNA virale
rivelatosi capace di resistere alla degradazione, rivelò la presenza di alcune
basi metilate. Si scoprì, quindi, l esistenza in E.coli di sistemi di restrizione/
modificazione capaci di metilare specifiche basi e, contemporaneamente, di
tagliare le stesse basi quando non metilate. Con questo sistema E.coli è capace
di degradare DNA esogeno tagliandolo in specifici siti di riconoscimento,
mentre gli stessi siti presenti sul DNA endogeno non sono tagliati perché
preventivamente metilati.
Alcuni ER, isolati da batteri differenti, riconoscono sequenze di taglio
Estremità
blunt
diverse, ma
che producono estremità compatibili, come EcoRI e MefI
Il risultato però……..
Isoschizomeri
Alcuni ER, detti isoschizomeri, isolati da batteri differenti,
riconoscono la stessa sequenza di taglio. Alcuni di essi
tagliano la
sequenza nello stesse posizioni, mentre altri tagliano in posizioni
differenti (Acc65I e KpnI)
estremità
5 protruding
estremità
3 protruding
Specificità e frequenza di taglio
Il numero di basi riconosciute determina la frequenza media di taglio e la
dimensione media dei frammenti generati. Enzimi che riconoscono sequenze
più corte tagliano più frequentemente e quindi producono frammenti più
numerosi e mediamente più corti, degli enzimi che riconoscono sequenze più
lunghe (8 bp rare cutter).
Ad es. l enzima AluI riconosce la sequenza AGCT. Assumendo che la
distribuzione delle 4 basi sia casuale, in ogni posizione del DNA ci sarà 1
probabilità su 4 (25%) di trovare una A. Analogamente 1 probabilità su 4 di
trovare una G, quindi la probabilità di trovare la sequenza AGCT sarà uguale a
1/4 n , dove n = numero di nucleotidi del sito di riconoscimento.
1/44 =1/256 basi,
1/46= 1/4096,
1/48= 1/55536
La maggior parte funziona in tamponi a pH 7/8, a 37°C.
Un unità di un enzima di restrizione è la quantità di enzima richiesta per
digerire completatamente 1 µg di DNA in un ora.
Estrazione di frammenti di DNA da gel
Dopo aver digerito un DNA con enzimi di restrizione ed averne separato i
frammenti risultanti su gel di agarosio, è possibile excidere dal gel, con un
bisturi, specifiche bande di interesse e purificarle da gel.
Esistono molti sistemi per purificare bande da gel, tra cui:
•
elettroeluizione
•
colonne a scambio ionico
•
gel-filtration
•
agarosio a basso punto di fusione
La Ligasi
La ligazione
La ligazione va ottimizzata rispetto a:
• temperatura e tempo di reazione
• concentrazione del DNA: totale, dell inserto e del vettore.
Temperatura
Calcolata tenendo in considerazione che la stabilizzazione
dell appaiamento tra estremità coesive è ottimale a basse temperature,
mentre l attività enzimatica della ligasi è massima a 37°C. Si utilizza
spesso una temperatura di 16°C per 12 ore.
Concentrazione
Basse concentrazioni di DNA totale favoriscono le reazioni di primo
ordine (intramolecolare) come la ricircolarizzazione del vettore.
Aumentare la concentrazione totale incrementando la concentrazione
di vettore, peggiora la situazione, ma aumentare la concentrazione
dell inserto aumenta la probabilità di avere vettori con inserti
multipli.
In genere
[DNA Tot] ≥ 10-20 ng/µl
rapporti molari I:V da 3:1 a 1:1
Vol. fin.= 10-20 µl.
N.B. si devono utilizzare i rapporti molari
moli = g/PM
Normalmente si considera statisticamente uguale il PM di ogni singolo
paio di basi il cui peso medio è 660 Dalton, quindi
moli = g/ 660 x bp
Metafora delle pere da 100gr o da 250gr
Esempio
Vogliamo ligare 50 ng di un vettore di 10 Kb con un inserto di 1 Kb,
utilizzando rapporti molari I/V 1:1 e 3:1, in due reazioni separate.
Rapporto I/V= 1:1
Rapporto I/V= 3:1
x = 15 ng
50 ng/10 kb = x ng/1 kb
x ng = 50/10
x = 5ng
Fosfatasi alcalina
Quando vettore ed inserto sono tagliati con lo stesso enzima di restrizione,
non si riesce ad evitare un elevata frequenza di ricircolarizzazione del
vettore.
P-5
vettore
AATTC
HO-3
3 OH
TTAAG -5 P
inserto
3 OH
TTAAG -5 P
P-5
AATTC
HO-3
vettore
Una strategia consiste nell utilizzo di una fosfatasi, come ad esempio la
fosfatasi alcalina (BAP), un enzima che rimuove il gruppo fosfato al 5
impedendo così l azione della ligasi. La defosforilazione del vettore con una
fosfatasi impedisce la ricircolarizzazione del vettore, abbassando
sensibilmente il background.
HO- AATTC
HO-3
P-5
vettore
AATTC
HO-3
3 OH
TTAAG
inserto
3 OH
TTAAG
vettore
-5 P
-OH
+ ligasi
vettore
OH HO
inserto
OH HO
vettore
• Clonaggio di un frammento con estremità blunt in un vettore con
estremità sticky 5 protruding (es.EcoRI)
FILLING IN
Consiste nel riempire un estremità sporgente al 5 con la Klenow, una
DNA polimerasi I modificata, in quanto priva dell attività 5 -3
esonucleasica
dTTP
5 P
3 OH
G 3 OH
C AATTC
dATP
dGTP
5 P
5 P
3 OH
G TTAAG
C AATTC
3 OH
5 P
• Clonaggio di un frammento con estremità blunt in un vettore con
estremità sticky 3 protruding (es. PstI)
TRIMMING
Non è possibile adoperare Klenow che, come tutte le polimerasi, sintetizza solo
in direzione 5 -3 . Useremo, quindi l attività 3 -5 esonucleasica di un enzima
come la DNA polimerasi del fago T4.
5 P
3 OH
T4
DNA pol.
CTGCA
G
5 P
3 OH
5 P
3 OH
C 3 OH
G
5 P
Clonaggio di un frammento con estremità blunt in un vettore con estremità
sticky (BamHI), oppure con estremità blunt
N.B. L efficienzà delle ligazioni blunt è abbastanza più bassa di quella di ligazioni
con estremità sticky
LINKERS
LINKER: corti oligonucleotidi autocomplementari con un sito di restrizione
5 -CCGGATCCGG-3
Questo corto oligo ds va ligato con il frammento con blunt end
Frammento SmaI
Frammento SmaI
+
CCGGATCCGG-3
GGCCTAGGCC-5
CCGGATCCGG-3
GGCCTAGGCC-5
+ BamHI
Frammento SmaI
CCGGATCCGG-3
GGCCTAGGCC-5
Frammento SmaI
CCG-3’
GGCCTAGGCC-5
Clonaggio di un frammento con estremità sticky BamHI in un vettore con
estremità sticky EcoRI
ADAPTERS
coppie di brevi oligonucleotidi parzialmente complementari con estremità coesive differenti
5 -GATCCCCGGG-3
3 -GGGCCCTTAA-5
5 GATC
3
inserto
3
CTAG 5’
5 -GATCCCCGGG-3
3 GGGCCCTTAA-5
5’ AATT
3
vettore
Annealing
& ligazione
GATC
inserto
GATCCCCGGGAATT
CTAGGGGCCCTTAA
vettore
3
AATT 5’
3
AATT 5’
Esercizio:
1. Dire che tipo di estremità producono i seguenti enzimi
2. Disegnare le estremità prodotte
3.
4.
Disegnare un linker EcoRI
Disegnare un adapter PstI-EcoRI
Risposta 3
5 -GGATCC-3
3 -CCTAGG-5
BamHI
5 -AAAGAATTCTTT-3
5 -AAAGAATTCTTT-3
3 -TTTCTTAAGAAA-5
5 -GAATTC-3
3 -CTTAAG-5
EcoRI
Risposta 4
5 -CTGCAG-3
3 -GACGTC-5
PstI
5 -GTTAAC-3
3 -CAATTG-5
SmaI
5 -AATTCTTTCCCCTGCA-3
3 -GAAAGGGG-5
5 -AATTCTTTCCCCTGCA-3
3 -GAAAGGGG-5






![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)


![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)
