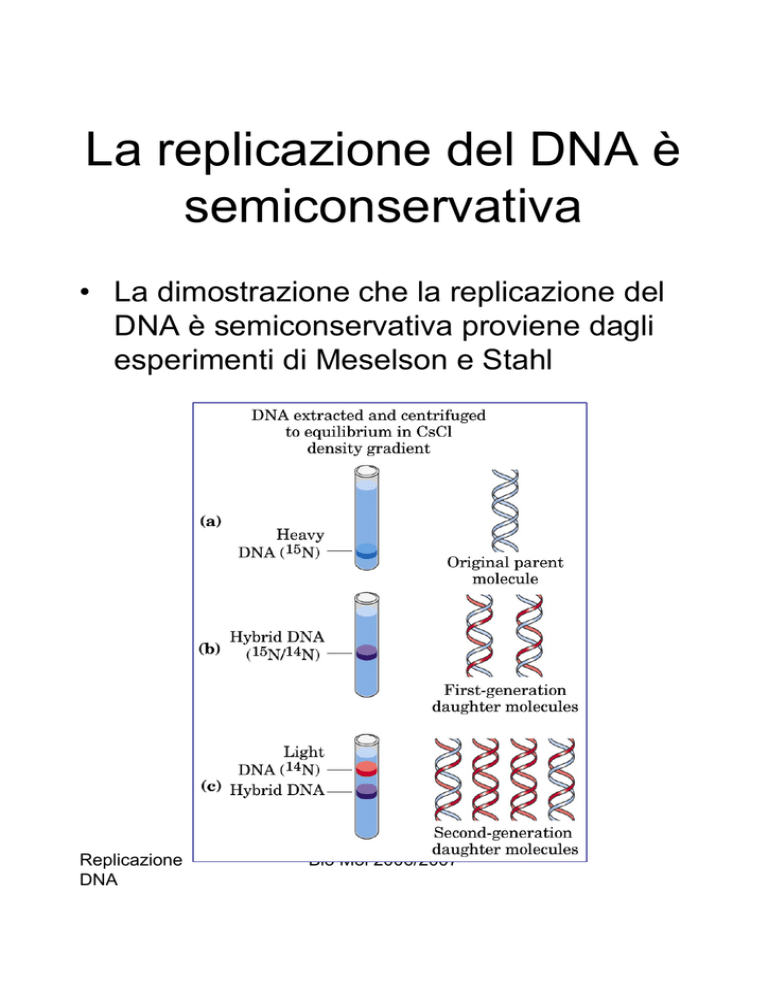
La replicazione del DNA è
semiconservativa
• La dimostrazione che la replicazione del
DNA è semiconservativa proviene dagli
esperimenti di Meselson e Stahl
Replicazione
DNA
Bio Mol 2006/2007
Modalità di replicazione:
bidirezionale o
unidirezionale
Esperimenti di John Cairns con Timidina triziata
Replicazione
DNA
Bio Mol 2006/2007
Replicazione
DNA
Bio Mol 2006/2007
Autoradiografia del cromosoma batterico dopo marcatura
per tempo breve con timidina triziata.
Si distingue una densità maggiore di granuli nei punti
di crescita della bolla replicativa (il resto del DNA non è visibile
perchè non è radioattivo):.
Replicazione
DNA
Bio Mol 2006/2007
Proprietà universali delle DNA
polimerasi
1. Necessitano di una catena polinucleotidica appaiata
allo stampo che presenta all’estremità un 3’-OH. ovvero
una giunzione innesco: stampo (primer-template)
Replicazione
DNA
Bio Mol 2006/2007
2. Richiedono come substrato i deossi-ribonucleosidi 5’trifosfato (dNTPs)
Replicazione
DNA
Bio Mol 2006/2007
3. La reazione di polimerizzazione procede in direzione 5’ 3’.
La polarità della catena sintetizzata è opposta a quella
del templato.
(NMP)n + dNTP → (NMP)n+1 + 2 Pi
Replicazione
DNA
Bio Mol 2006/2007
Bilancio della reazione
(NMP)n + dNTP ↔ (NMP)n+1 + PPi
------------------------------------------------------------------------Bilancio parziale - 5 kJ/mole
PPi → 2 Pi
ΔG0’ -30 KJ/mole
___________________________________
dNTP + (NMP)n → (NMP)n+1 + 2 Pi
Bilancio netto ~ - 35 kJ/mole
Keq = 105 M-1
Partendo da
dNMP
dNDP
-30 kJ/mole
Bilancio netto
+ 25 kJ/mole
Reaz sfavorita
– 5 kJ/mole
Reaz. debolmente Favorita
dNTP - 30 kJ/mole
a PPi + dNMP
PPi 2 Pi - 30 kJ/mole
Replicazione
DNA
-35kJ/mole
Reazione irreversibile
Bio Mol 2006/2007
Reazione inversa: pirofosforolisi. Avviene in vitro in
presenza di elevate concentrazioni di pirofosfato
(NMP)n + dNTP ↔ (NMP)n+1 + PPi
PPi + (NMP)n+1
Replicazione
DNA
dNTP + (NMP)n
Bio Mol 2006/2007
Struttura della DNA
polimerasi
Replicazione
DNA
Bio Mol 2006/2007
Struttura a palmo di mano delle DNA polimerasi
4 differenti substrati: dATP, dGTP, dTTP, dCTP
Replicazione
DNA
Bio Mol 2006/2007
Il problema della fedeltà
di replicazione
1. Selettività cinetica
Velocità di formazione del legame dipende dall’instaurarsi
della corretta geometria dll’appaiamento
Replicazione
DNA
Bio Mol 2006/2007
Esclusione sterica dei ribonucleotidii
Replicazione
DNA
Bio Mol 2006/2007
Correzione di un errore di incorporazione
Replicazione
DNA
Bio Mol 2006/2007
Secondo controllo: attività di
proof-reading
Le DNA polimerasi sono dotate
di attività esonucleasica 3’
5’ .
Questa attività consente
all’enzima di “tornare indietro” e
correggerre l’errore.
Terzo controllo: livelli intracellulari
di dNTPs mantenuti bilanciati
Tasso di errore DNA pol: 1 nucleotide/105
Dopo correzione tramite attività exonucleasica: 1 /107
Tasso di errore
(meccanismi riparazione post-sintesi: 1 nucleotide/1010
Replicazione
DNA
Bio Mol 2006/2007
La processività: una
proprietà fondamentale
delle DNA polimerasi
Replicazione
DNA
Bio Mol 2006/2007
Filmato
Replicazione
DNA
Bio Mol 2006/2007
La replicazione del
DNA nei procarioti
In E.coli esistono almeno cinque DNA polimerasi:
DNA polimerasi I (enzima di Kornberg)
•
Singolo monomero di circa 100 kDa (prodotto del gene polA)
1.
Possiede attività polimerasica 5’ 3’
2.
Attività esonucleasica 3’- 5’ (proof-reading)
3.
Attività esonucleasica 5’- 3’ (produce NMP e oligonucleotidi)
4.
E’ poco processivo ma presente in alto numero di molecole
(400/cellula)
Utilizza i nick nel DNA, gap e primer appaiati a un ssDNA .
Gap
5’
3’
3’OH
DNA fagico a singolo filamento
M13 o φX 174 o DNA plasmidico
Replicazione
DNA
Bio Mol 2006/2007
Nick
3’OH
Attività di Nick-translation (spostamento del nick)
della DNA pol I
Replicazione
DNA
Bio Mol 2006/2007
Tramite trattamento con proteasi si sono ottenuti due
frammenti della pol I: uno possiede l ’attività di
polimerizzazione e l’ attività 3’ 5’ esonucleasica e l’altro
ha solo l’attività 5’ 3’ esonucleasica.
Taglio proteolitico
C
Frammento di
Klenow
Pol 5’ 3’
Exo 3’-5’
N
Exo 5’ 3’
Funzioni della DNA pol I:
• E’ un enzima essenziale: durante la replicazione del
DNA effettua la sintesi del DNA dopo che il primer è
stato rimosso (mai isolati mutanti completamente privi di
tale attività).
• Inoltre, la DNA pol I è’ coinvolta nei meccanismi di
riparazione del DNA.
Mutanti PolA1 sono infatti ipersensibili a UV e mutageni
Replicazione
DNA
Bio Mol 2006/2007
•
DNA polimerasi II (gene PolB )
Singolo monomero (120 kDa)
Non possiede l’attività esonucleasica
5’ 3’.
Ha attività di proof-reading.
Non è essenziale per la replicazione
Mutanti polB non sono ipersensibili agli UV
Mutanti polA polB sono ancora capaci di
replicare
•
DNA polimerasi III (è il vero enzima
responsabile della replicazione del
cromosoma di E.coli – è presente in
10-20 molecole/cellula. Mutanti privi di
tale attività non sono vitali)
•
•
DNA polimerasi IV
DNA polimerasi V
Queste ultime due DNA polimerasi sono coinvolte in
particolari meccanismi di riparazione del DNA.
Replicazione
DNA
Bio Mol 2006/2007
La DNA polimerasi III è
costituita da molte
subunità
• DNA polimerasi “core” (3 subunità
α,ε,θ)
α:polimerasi 5’ 3’
ε: esonucleasi 3’ 5’
• Proteina τ (fattore di
dimerizzazione del “core”)
• Complesso γ (caricatore della
pinza)
• Subunità β (pinza scorrevole)
• Polimerasi III* + β
Replicazione
DNA
Oloenzima
Bio Mol 2006/2007
Subunità β o fattore di
processività
Replicazione
DNA
Bio Mol 2006/2007
DNA polimerasi III oloenzima
Replicazione
DNA
Bio Mol 2006/2007
Ipotesi sui meccanismi di replicazione
La replicazione del DNA è semidiscontinua: Leading
strand (filamento guida)
e lagging strand (filamento ritardato
FRAMMENTI DI OKAZAKI:
1000 nu in batteri
200 nu in eucarioti
Replicazione
DNA
Bio Mol 2006/2007
L’enzimologia della replicazione:
proteine richieste per la
replicazione del DNA: l’ elicasi
(DnaB). Esamero di subunità
identiche.
Esperimento per dimostrare un’attività elicasica
Replicazione
DNA
Bio Mol 2006/2007
Altre proteine richieste per la
replicazione del DNA:
le single strand binding
proteins (SSB)
Replicazione
DNA
Bio Mol 2006/2007
L’inizio di un nuovo filamento
di DNA richiede un innesco a
RNA:
la primasi (DnaG)
La primasi è una particolare RNA polimerasi che
interviene nella replicazione del DNA.
Replicazione
DNA
Bio Mol 2006/2007
Come si volge l’unione dei frammenti di Okazaki?
Enzimi coinvolti
L’RNasi H rimuove il primer a RNA (tranne ultimo ribonucleotide
che viene rimosso da una 5’ exo-nucleasi)
Interviene successivamente la DNasi I che completa i gap con
sintesi di nuovo DNA
Interviene infine la ligasi a unire tramite legami covalenti i frammenti
Replicazione
DNA
Bio Mol 2006/2007
La DNA ligasi
ricongiunge un 3’-OH con
un 5’-fosfato
Ligasi di E.coli richiede NAD
Ligasi del fago T4 richiede ATP
Replicazione
DNA
Bio Mol 2006/2007
Forca replicativa in
movimento
filmato
Replicazione
DNA
Bio Mol 2006/2007
Replicazione
DNA
Bio Mol 2006/2007
Per semplicità le SSB non sono riportate
Replicazione
DNA
Bio Mol 2006/2007
La DNA polimerasi ha finito di sintetizzare un frammento
e si stacca
Replicazione
DNA
Bio Mol 2006/2007
Il complesso γ carica la subunità β a livello del primer
più vicino
Replicazione
DNA
Bio Mol 2006/2007
La DNA polimerasi si lega al primer più vicino, si
forma una nuova ansa (loop) e riprende la
sintesi di un nuovo frammento
Replicazione
DNA
Bio Mol 2006/2007
Proteine richieste per la
replicazione in E.coli
•
•
•
•
•
•
•
•
DNA elicasi
SSB
primasi
DNA polimerasi III
RNasi H
DNA polimerasi I
DNA ligasi
Topoisomerasi
Replicazione
DNA
Bio Mol 2006/2007
Problemi topologici legati alla replicazione
del DNA
Replicazione
DNA
Bio Mol 2006/2007
DNA polimerasi
eucariotiche
Attività
Ruolo
Polimerasi α
DNA pol/
primasi
replicazione
Polimerasi δ
DNA pol/3’ exo
replicazione
Polimerasi ε
DNA pol/3’exo
replicazione
riparazione
Polimerasi γ
DNA pol/3’ exo
repl.DNA mit
Polimerasi β
DNA pol
riparazione
e altre (in totale circa 15 DNA polimerasi)
Replicazione
DNA
Bio Mol 2006/2007
Switching delle DNA polimerasi in eucarioti
Replicazione
DNA
Bio Mol 2006/2007
Enzimi della
replicazione in eucarioti
• La replicazione in eucarioti molti fattori:
•
•
•
•
SSB
Innesco
Sintesi DNA
Fattore di
processività
• Caricatore PCNA
• Rimozione RNA
• Saldatura gap
Replicazione
DNA
RP-A
DNApolα
DNApolδ o ε
PCNA (proliferation cell
nuclear antigen)
RF-C
FEN1 (Flap-endonucleasi1)
e RNasi H
DNA ligasi I
Bio Mol 2006/2007
Sliding clamps, o pinze scorrevoli, da vari organismi
PCNA = proliferating cell nuclear antigen
Replicazione
DNA
Bio Mol 2006/2007
Modello d’inizio della
replicazione
Replicazione
DNA
Bio Mol 2006/2007
Come ha origine la
replicazione del DNA
cromosomale di E.coli?
• Il cromosoma di E.coli è
costituito da un unico
REPLICONE
• La replicazione inizia da
un’unica origine detta oriC (il
replicatore)
• Vi è una proteina detta
iniziatrice che avvia la
replicazione a livello di oriC
(proteina DnaA)
Replicazione
DNA
Bio Mol 2006/2007
Come è stata
identificata oriC?
Plasmide
privato della
propria origine
di replicazione
Replicazione
DNA
Bio Mol 2006/2007
Caratteristiche
dell’’OriC di E.coli
oriC
Segmento di 245 bp
Replicazione
DNA
Bio Mol 2006/2007
Struttura dei replicatori
Virus SV40
lievito
Verde : siti di legame per l’iniziatore
Blue: segmenti dove il DNA inizia a separarsi
Rosso: sito dove inizia la sintesi del DNA
Replicazione
DNA
Bio Mol 2006/2007
La proteina SeqA impedisce il re-inizio
Replicazione
DNA
Bio Mol 2006/2007
Terminazione della
replicazione in E.coli
Replicazione
DNA
Bio Mol 2006/2007
• In eucarioti il DNA è costituito da
molteplici repliconi
• Ogni replicone ha un replicatore e una
propria origine di replicazione
replicone
replicone
• In ogni replicone la replicazione è di tipo
bidirezionale.
• Negli eucarioti il DNA viene replicato
durante la fase S e i replicatori vengono
attivati in tempi diversi e una sola volta
per ciclo cellulare
Replicazione
DNA
Bio Mol 2006/2007
• In lievito vi sono circa 400 repliconi,
ciascuna ha un proprio replicatore detto
ARS (autonomous replicating
sequence).
(isolamento ARS per capacità di conferire replicazione autunoma a
plasmidi integrativi)
-L’iniziatore in eucarioti è una proteina
esamerica chiamata ORC: (Origin
Recognition Complex).
ORC lega il replicatore e recluta altre
proteine formando il Pre-RC.
All’inizio della Fase S, sotto controllo
delle proteine chinasi ciclina-dipendenti,
inizia la replicazione.
Replicazione
DNA
Bio Mol 2006/2007
Replicazione
DNA
Bio Mol 2006/2007
Nell’uomo esistono 30.000
repliconi.
I replicatori sono più lunghi e
ancora poco caratterizzati
Velocità nell’uomo: 200 nt/s
Replicazione
DNA
Bio Mol 2006/2007
Il problema della replicazione
all’estremità dei cromosomi
lineari
Molecola della
generazione
successiva più
corta
Perdita di informazione genica
Replicazione
DNA
Bio Mol 2006/2007
Telomeri e telomerasi: una soluzione adottata
dagli eucarioti
Telomeri: estremità dei cromosomi formate da ripetizioni
di sequenze ricche in TG e disposte testa-coda
Si estendono anche per un tratto a singolo filamento
Responsabili del reclutamento di un enzima detto telomerasi
La telomerasi: una DNA polimerasi - RNA dipendente
E’ costituita da una componente
proteica e da una ad RNA (ribonucleoproteina)
FUNZIONE: allunga il terminale 3’ dei cromosomi
Utilizzando come stampo il proprio RNA
Replicazione
DNA
Bio Mol 2006/2007
Sequenza
complementare
ai telomeri umani:
5’-TTAGGG-3’
(MINISATELLITE)
Replicazione
DNA
Bio Mol 2006/2007
Allunagamento del terminale 3’ del telomero da parte
della telomerasi
Replicazione
DNA
Bio Mol 2006/2007
Filmato sull’attività della telomerasi
Replicazione
DNA
Bio Mol 2006/2007
Le telomerasi sono molto attive durante lo
sviluppo embrionale, in cellule staminali o in
riproduzione
In cellule in coltura con l’insorgenza della
senescenza le telomerasi non sono più attive e
i cromosomi si accorciano
Al contrario in cellule tumorali le telomerasi
sono attive o iperattive (causa o effetto?)
Le telomerasi rappresentanoun bersaglio per
la terapia antitumorale.
Replicazione
DNA
Bio Mol 2006/2007



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)


![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)




![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)