MODULO 3 – B
LA DUPLICAZIONE DEL DNA BATTERICO
Per capire il processo di replicazione, trascrizione e traslazione dell’ informazione
genetica è necessario descrivere il DNA.
Il DNA nei batteri è formato da due catene polinucleotidiche che sono
avvolte l’uno sull’altra in una struttura elicoidale.
I nucleotidi sono in genere quattro, ripetute in sequenze specifiche. Ciascun
nucleotide è formato da una base purinica legata tramite il legame βglicosidico ad uno zucchero a 5 atomi di carbonio, cui è legato a sua volta un
radicale fosforico; si legano al carbonio in posizione 3’ del deossiribosio di un
nucleotide e al carbonio in posizione 5’ di quello adiacente (gli atomi C 3’ e 5’
sono gli unici ad avere una funzione ossidrilica che consente l’esterificazione
con il radicale fosforico), mediante un legame fosfodiesterico.
Le due catene polinucleotidiche che formano una molecola di DNA sono
tenute insieme da legami idrogeno tra le basi azotate delle due catene
adiacenti (adenina-timina; guanina-citosina: catena complementare).
A causa dell’ angolatura del legame covalente e delle forze non covalenti, il
DNA nativo presenta una conformazione ad elica destrofica, che può
assumere configurazioni differente a seconda dell’ambiente:
A) Ambiente acquoso : B-DNA;
B) Ambiente disidratato : A-DNA;
C) Ambiente con elevata presenza di cationi : Z-DNA.
Vi sono due proteine enzimatiche che agiscono sul numero di legame del
DNA (numero di legame: numero di volte che un filamento di una molecola
di DNA incrocia l’altro filamento) e sono le TOPOISOMERASI.
1. La DNA-TOPOISOMERAI I scinde un filamento e fa variare di
unità il numero di legame.
2. La DNA-TOPOISOMERASI II scinde entrambi i filamenti e fa
variare di due unità il numero di legame.
La DUPLICAZIONE DEL DNA batterico avviene attraverso un processo
asessuato, detto SCISSIONE BINARIA, durante la quale la cellula si divide
in due metà più o meno uguali dalla progenitrice iniziale.
Avviene, quindi, con un meccanismo semiconservativo, ossia da una
molecola di DNA si originano due nuove molecole nelle quali una è
parentale, FILAMENTO GUIDA, mentre l’altra è un filamento
complementare di nuova sintesi, FILAMENTO LENTO.
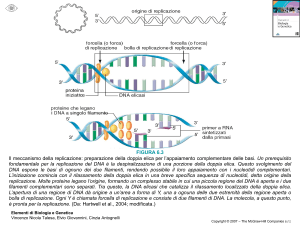
La replicazione ha inizio in un sito specifico del cromosoma batterico, detto
oriC per ORIGINE DI REPLICAZIONE.
Durante la replicazione, la molecola di DNA si srotola: un gruppo proteico,
un esomero della proteina DnaB, si lega, unito da un’altra proteina, detta
DnaC, alle due FORCHE REPLICATIVE, i due siti di sintesi dei nuovi
filamenti, formando un anello intorno al DNA e completando la costruzione
del complesso iniziale.
La proteina DnaB, detta ELICASI, ha la capacità di svolgere la doppia elica
e, assistita dalla DNA-girasi, di promuovere la separazione dei due filamenti.
Le forche replicative sono formati da un complesso di proteine che hanno la
funzione di incorporare i nucleotidi complementari nelle eliche, copiando
quelle parentali.
Il complesso di inizio, oltre a comprendere oriC, contiene un certo numero di
proteine. OriC è composto da 245 nucleotidi.
Per iniziare il processo sono richieste 12 proteine. I complessi ATP-proteina
dnaA si legano ai 5 siti composti da nove paia di basi posti in oriC.
L’ esamero DnaB si lega a 6 molecole di DnaC per formare un complesso di
12 polipeptidi. Lo srotolamento dell’ elica fa spostare la dnaA, ed espone le
basi nucleotidiche perché possa aver luogo l’ appaiamento complementare.
La separazione delle due eliche richiede l’ intervento della proteina SSB o
SINGLE STAND BINDING, proteina che stabilizza l’ elica; in questa fase
possono intervenire proteine prodotte da geni rep, dnaP e dnaS.
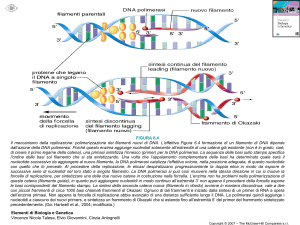
L’ enzima primasi, la componente più importante di questo complesso,
sintetizza un filamento già esistente, detto PRIMER di RNA, indispensabile
per la sintesi e allungamento del DNA.
La polimerizzazione dei singoli desossinucleotidi-3-fosfato avviene ad opera
dell’enzima DNA polimerasi, presente nei batteri in 3 varianti:
· DNA-pol-I : con la DNA-ligasi, svolge un ruolo cruciale nella rimozione
dei primer di RNA e nel colmare gli spazi vuoti della sequenza;
· DNA-pol-II : il suo ruolo non è stato del tutto chiarito;
· DNA-pol-III : è responsabile dell’ allungamento dei filamenti nascenti.
La polimerizzazione avviene in direzione 5’-3’. L’accuratezza della
replicazione del filamento parentale è assicurata dalla formazione dei legami
idrogeno delle basi: guanina (G) – citosina (C); timina (T) – adenina (A).
I frammenti di dna sintetizzati sono detti FRAMMENTI DI OKATAKI; essi
devono essere completati tramite la rimozione degli inneschi da parte di
ENDONUCLEASI, enzimi che scivolano un polimerasi di riparazione, cosi
il frammento di RNA è sostituito con DNA.
Lo stadio finale nella maturazione di DNA del filamento sintetizzato è la
saldatura dei frammenti di Okataki da parte della DNA-ligasi: esso catalizza la
formazione di un legame fosfodiestere tra il gruppo ossidrilico 3’ di un
frammento e il gruppo fosfato 5’ del frammento adiacente.
Durante la replicazione, possono verificarsi errori, creando un difetto di
appaiamento delle basi (genoma diverso da quello parentale).
Il tasso di errore in Escherichia Coli è inferiore a 10 per paio di basi
duplicate. Il tasso di errore è basso a causa dell’attività di riparazione o correzione
di bozze o PROOF-READING della DNA-polimerasi.
Il complesso di proteine MUTS, MUTH, MUTL è in grado di identificare e
rimuovere una base non correttamente appaiata.
Il filamento parentale di una doppia elica è metilato (ovvero viene sostituito
un gruppo metile su vari substrati). La proteina MutS riconosce e lega una
coppia di basi errate, il MutH scorre lungo il filamento fino alla prima base
metilata; la MutL si lega alla oltre due proteine e permette il taglio del
filamento non metilato e la rimozione della porzione contenente l’errore. Il
passaggio finale è la sintesi del frammento di DNA mancante.
Purine
Pirimidine



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)