Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
1
2
AMPLIFICAZIONE DEL DNA MEDIANTE REAZIONE DI PCR
(Polymerase Chain Reaction)
Progetto Lauree Scientifiche 2009-2010
Esercitazione di
Antropologia Molecolare
1. Introduzione
1.1 Che cosa studia l’Antropologia Molecolare
L’Antropologia Molecolare, una disciplina relativamente recente nell’ambito di quelle dedicate allo
studio dell’Uomo, è incentrata sull’analisi della variabilità biologica delle sue popolazioni.
Lo studio di questa variabilità, non necessariamente corrispondente a quella osservabile da un punto
di vista culturale e/o linguistico, permette di ricostruire l’origine e l’eventuale mescolamento delle
popolazioni umane, nonchè la storia evolutiva dell’intera specie (Homo sapiens), dalla sua nascita
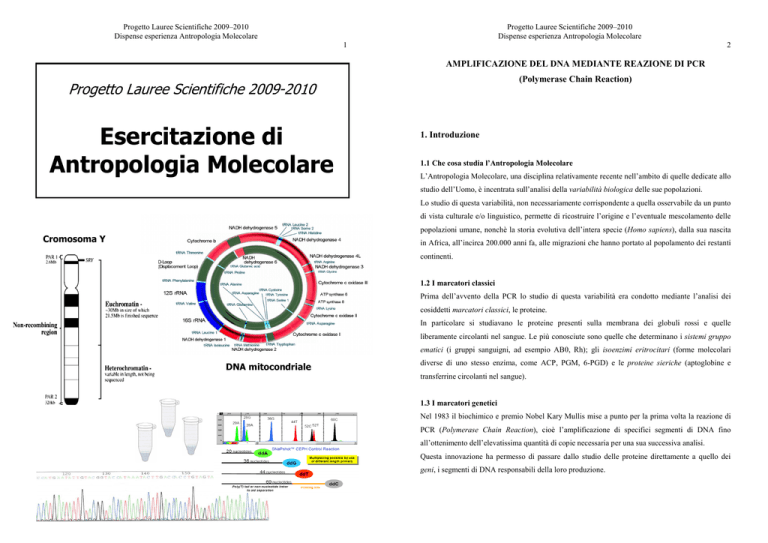
Cromosoma Y
in Africa, all’incirca 200.000 anni fa, alle migrazioni che hanno portato al popolamento dei restanti
continenti.
1.2 I marcatori classici
Prima dell’avvento della PCR lo studio di questa variabilità era condotto mediante l’analisi dei
cosiddetti marcatori classici, le proteine.
In particolare si studiavano le proteine presenti sulla membrana dei globuli rossi e quelle
liberamente circolanti nel sangue. Le più conosciute sono quelle che determinano i sistemi gruppo
ematici (i gruppi sanguigni, ad esempio AB0, Rh); gli isoenzimi eritrocitari (forme molecolari
DNA mitocondriale
diverse di uno stesso enzima, come ACP, PGM, 6-PGD) e le proteine sieriche (aptoglobine e
transferrine circolanti nel sangue).
1.3 I marcatori genetici
Nel 1983 il biochimico e premio Nobel Kary Mullis mise a punto per la prima volta la reazione di
PCR (Polymerase Chain Reaction), cioè l’amplificazione di specifici segmenti di DNA fino
all’ottenimento dell’elevatissima quantità di copie necessaria per una sua successiva analisi.
Questa innovazione ha permesso di passare dallo studio delle proteine direttamente a quello dei
geni, i segmenti di DNA responsabili della loro produzione.
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
4
3
La variabilità delle popolazioni umane osservata a livello proteico è, infatti, il risultato di una ben
maggiore variabilità genetica, che consiste in differenze, da individuo a individuo, nella sequenza
Polimorfismi unici
(M91, M31, YAP…)
nucleotidica dei geni che portano a differenze nella sequenza amminoacidica delle relative proteine.
Un polimorfismo genetico è quindi una determinata posizione nella sequenza del DNA che risulta
variabile fra gli individui di una stessa specie ed è tale quando una sua variante presenta una
frequenza di almeno 1% in una data popolazione.
1.4 I marcatori genetici uniparentali
I principali marcatori genetici studiati nell’ambito dell’Antropologia Molecolare sono i
polimorfismi presenti sul cromosoma Y e sul DNA mitocondriale.
Rispetto ai cromosomi autosomici (la stragrande maggioranza del DNA contenuto nel nucleo delle
cellule), questo materiale genetico ha una trasmissione uniparentale, esclusivamente da padre a
figlio (il cromosoma Y) e da madre a figlio/a (il DNA mitocondriale), senza cioè che avvenga
ricombinazione, ovvero il rimescolamento di parti del DNA materno e paterno nei figli.
E’ quindi possibile determinare la discendenza e l’ancestralità degli individui nella linea materna e
in quella paterna, poiché individui con un’origine comune condivideranno gli stessi polimorfismi.
1.5 Il cromosoma Y
Il cromosoma Y è uno dei due cromosomi sessuali della nostra specie, in particolare quello che
determina il sesso maschile e si trova quindi solamente nei maschi.
Poiché il 95% della sua sequenza è non ricombinante, nel corso della nostra storia evolutiva è stato
trasmesso pressoché inalterato nel corso delle generazioni dai progenitori ancestrali maschi.
Pertanto le attuali differenze fra individui sono dovute esclusivamente all’accumulo di mutazioni
nel tempo.
Questo permette di individuare polimorfismi che avendo un basso tasso di mutazione, non essendo
quindi ricorrenti ma unici, risultano spesso specifici di determinate popolazioni o aree geografiche e
vengono pertanto utilizzati per costruire la filogenesi del cromosoma Y rappresentata sotto forma di
albero.
Percorrendo i rami evolutivi di questo albero, alla cui radice si trova l’ipotetico antenato comune, si
arriva fino alle diramazioni recenti, che testimoniano le ultime mutazioni avvenute. Inoltre,
seguendo un ordine gerarchico i cromosomi Y analizzati possono essere raggruppati in linee dette
aplogruppi, definiti dalla presenza di determinati polimorfismi.
Albero filogenetico del cromosoma Y
Aplogruppi
(A, B, C…)
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
5
6
2. Il polimorfismo YAP
filamenti che ne costituiscono la doppia elica. La Taq polimerasi viene infatti estratta dal betterio
Lo YAP appartiene alla famiglia dei polimorfismi Alu ed è costituito da un’inserzione di 280 paia
termofilo Thermus acquaticus, che vive in natura a temperature prossime agli 80°C.
basi nel sito nucleotidico DYS287, situato sul braccio lungo del cromosoma Y.
L’utilizzo di questo enzima permette quindi di riprodurre in vitro quanto avviene all’interno delle
Le due forme (alleli), YAP- (assenza di inserzione) e YAP+ (presenza di inserzione), indicano
cellule quando il DNA viene copiato, garantendo la selezione, l’isolamento e l’amplificazione di
rispettivamente la forma ancestrale e quella derivata, che sembra essere comparsa, circa 141.000,
qualsiasi regione del DNA di cui si conoscano le estremità fiancheggianti a monte e a valle.
anni fa, anche se non è ancora chiaro se si sia originata in popolazioni del continente africano o
Perché la polimerasi possa svolgere la sua funzione è tuttavia necessario fornirgli i reagenti che
asiatico.
essa utilizzerebbe all’interno delle cellule:
Gli individui che presentano l’inserzione YAP sul proprio cromosoma Y possono appartenere a due
soli aplogruppi: D o E. Per questo motivo tale polimorfismo viene generalmente testato sull’intero
dNTPs = deossinucleotidi trofosfati (dATP, dTTP, dCTP, dGTP). Sono costituiti da una
campione come screening iniziale per indirizzare le analisi successive in quanto permettere di
base azotata (adenina, timina, citosina, guanina) legata ad un deossiribosio, uno
distinguere due grossi gruppi di linee del cromosoma Y (D e E / tutti gli altri aplogruppi).
zucchero privo del gruppo OH in posizione 2’ e con 3 gruppi fosfato in posizione 5’.
Nella miscela di reazione i 4 deossinucleotidi devono essere presenti in eguale quantità
in modo che la polimerasi abbia la stessa probabilità di utilizzarli, a seconda di quello
O A
D
D
B
Q F
E
necessario ad ogni inserzione. La polimerasi aggiunge un deossinucleotide alla volta,
G
K
legandone il gruppo fosfato in posizione 5’, al gruppo OH in posizione 3’ dell’ultimo
nucleotide del primer. In tal modo ogni nucleotide viene appaiato al nucleotide
C
E
D
D
L
P
formano fra le rispettive basi azotate.
D
E
H
YAP+
N
I
R
E
complementare presente sul filamento di DNA stampo, grazie ai legami idrogeni che si
J
M
YAP-
Cromosomi Y appartenenti agli aplogruppi D, E (YAP+) e ai restanti aplogruppi (YAP-)
MgCl2 = cloruro di magnesio. Fornisce gli ioni Mg++ che fungono da catalizzatori necessari
3. La reazione di PCR (Polymerase Chain Reaction)
La PCR è una reazione che sfrutta la capacità di una polimerasi batterica, l’enzima che nelle cellule
batteriche si occupa di copiare il DNA quando il batterio si moltiplica, di resistere a temperature
sufficientemente alte da permettere la denaturazione del DNA, ovvero la separazione dei due
per il funzionamento della polimerasi.
Buffer = tampone di reazione. Soluzione salina che garantisce le condizioni chimico-fisiche
idonee per un’ottimale attività della polimerasi.
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
8
7
Primers = oligonucleotidi. Piccole sequenze di DNA, lunghe 18-25 basi, complementari alle
sequenze fiancheggianti la regione che si vuole amplificare e alle quali si appaiano. Dal loro
ultimo nucleotide la polimerasi inizia ad aggiungere i nucleotidi che costituiranno il nuovo
filamento.
Una volta miscelati i reagenti in una mix di reazione e aggiunto il DNA che si vuole amplificare, la
reazione di PCR avviene automaticamente all’interno di un termociclatore, un’apparecchiatura che
compie gli innalzamenti ed abbassamenti di temperatura necessari allo svolgimento della reazione.
Il ripetersi ciclico delle 3 fasi della reazione, generalmente per un numero totale di 30-35 cicli, con
il raddoppio del numero di frammenti di DNA ad ogni ciclo, poiché entrambi i filamenti di ogni
doppia elica di DNA sono utilizzati come stampo, garantisce un incremento esponenziale delle
copie di DNA, fino ad alcuni milioni.
Ogni ciclo di reazione è costituito da tre fasi:
Estensione = la temperatura è fatta risalire fino a raggiungere i 72°C, temperatura che
garantisce la massima efficienza della Taq polimerasi. Si realizza così la rapida sintesi di un
nuovo filamento di DNA per ciascun filamento stampo, con l’aggiunta di 35-100 nucleotidi
al secondo. La polimerasi procede nella sintesi in direzione 5’-3’, attaccando il gruppo
fosfato in posizione 5’ del nuovo dNTP al gruppo OH in posizione 3’ dell’ultimo nucleotide
del primer o, successivamente, dell’ultimo nucleotide del filamento in costruzione.
Denaturazione = la temperatura è portata rapidamente a 94°C con conseguente rottura dei
legami idrogeno fra le basi complementari dei due filamenti che compongono la doppia
elica del DNA e loro separazione.
Al termine dei cicli previsti, il cui numero varia in base alle dimensioni del frammento di DNA che
si vuole amplificare, avviene una fase di estensione finale, a 72°C per alcuni minuti, che assicura il
completamento dell’allungamento dei filamenti di DNA rimasti incompleti.
Allineamento dei primers = la temperatura viene diminuita rapidamente fino alla
temperatura di melting (TM), specifica di ogni tipo di primer, e alla quale i primers stessi si
3.1 Protocollo di amplificazione del marcatore YAP
allineano alle regioni complementari presenti sui due filamenti di DNA utilizzati come
Di seguito sono riportate le sequenze dei primers necessari per l’amplificazione del frammento di
stampo. La formazione di legami idrogeno fra le basi azotate del primer e del filamento
DNA di 150 paia basi contenente il locus DYS287 sul quale può avvenire l’inserzione YAP.
stampo ne garantiscono un appaiamento stabile. La polimerasi si lega a questo doppio
filamento (DNA stampo + primer) ed è così pronta ad iniziare la sua attività.
YAP.F (forward)
5' CAG GGG AAG ATA AAG AAA TA 3'
YAP.R (reverse)
5' ACT GCT AAA AGG GGA TGG AT 3'
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
9
L’amplificazione di ciascun campione di DNA è eseguita in un volume finale (Vf) di 25 µl di una
10
4. Visualizzazione dei risultati
miscela di reazione contenente i reagenti. Di ciascuno di questi sono riportate in tabella la
concentrazione iniziale di stoccaggio (Ci) e quella finale nella miscela di reazione (Cf).
4.1 Elettroforesi su gel d’agarosio
Per calcolare la quantità unitaria di ciascun reagente (Vi), necessaria per l’amplificazione di un solo
Il successo della reazione di amplificazione e la presenza/assenza dell’inserzione YAP vengono
campione di DNA, è necessario applicare la regola delle diluizioni di seguito riportata:
verificate mediante corsa elettroforetica dei frammenti di DNA ottenuti su gel d’agarosio.
L’elettroforesi su gel permette la separazione di frammenti di DNA di dimensioni e peso differenti
regola delle diluizioni Vi x Ci = Vf x Cf
grazie all’applicazione di un campo elettrico.
Il DNA, posto in una soluzione tampone con pH e salinità adeguate, mantiene una carica netta
negativa, per la presenza dei numerosi gruppi fosfato che legano i nucleotidi all’interno di ciascun
Miscela di reazione
Reagenti (Ci)
H2O
CONC.
Finale
(Cf)
QUANTITA'
UNITARIA
(µ
µl)
QUANTITA'
x n° tot campioni
(µ
µl)
POPOLAZIONE:
filamento, e pertanto migrerà nel campo elettrico in direzione del polo positivo.
LISTA CAMPIONI
Durante questa migrazione i frammenti di maggiori dimensioni si muoveranno con maggior
difficoltà e lentezza attraverso i pori del gel rispetto ai frammenti più piccoli e leggeri, rimanendo
quindi più vicini ai pozzetti nei quali sono stati caricati.
/
DNTPs Mix
(10 mM)
0,4 mM
PRIMER F
(10 µM)
0,2 µM
K-
PRIMER R
(10 µM)
0,2 µM
TOT
MgCl2
(25mM)
2,5 mM
(5X)
1X
BUFFER
Taq
(5 U/µ
µl)
lasciarne sulle pareti.
d’alluminio.
24
(20 ng/µ
µl)
3. Trasferire la beuta sul becco bunsen (o nel forno a microonde ma rimuovendo la carta
1
d’alluminio) in modo da sciogliere l’agarosio e mandarlo in soluzione.
25
Volume finale (Vf)
4. Attendere qualche minuto che si raffreddi, aggiungere 1.5 µl di Bromuro di Etidio che funge
CICLI DI PCR
TEMP (°C)
STEP
2 min
94
1
DENATURAZIONE
1 min
94
2
ANNEALING
1 min
52
3
ESTENSIONE
90 sec
72
4
5 min
72
5
∞
4
6
ESTENSIONE FINALE
STOP
da intercalante delle basi azotate del DNA permettendone la visualizzazione ai raggi
DURATA
ATTIVAZIONE Taq
1. Pesare 0.6 g di agarosio in polvere e trasferirlo in una beuta facendo attenzione a non
2. Aggiungere 30 ml di TBE 1X (tris-buffer-EDTA), chiudere la beuta con un po’ di carta
1U
Tot mix
DNA
4.2 Preparazione di un gel d’agarosio al 2%
ultravioletti.
5. Versare la soluzione nel vassoio dopo aver aggiunto i pettini ad una delle due estremità e al
35 cicli
centro.
6. Attendere che il gel sia solidificato, togliere i pettini e sistemare il gel nella vasca di corsa,
quindi aggiungere il tampone (TBE 1X) fino a coprire il gel con un sottile strato.
Progetto Lauree Scientifiche 2009–2010
Dispense esperienza Antropologia Molecolare
11
4.3 Caricamento degli amplificati
Preparare su parafilm tante gocce da 1-2 µl di Loading Buffer 6X e a ciascuna aggiungere 8 µl di
DNA amplificato, miscelare bene e caricare nei pozzetti del gel. Il buffer di caricamento ha la
funzione di rendere visibile il campione durante la fase di caricamento ed appesantirlo affinché si
depositi sul fondo del pozzetto.
La corsa elettroforetica viene effettuata a 95 volt per un tempo di almeno 15 minuti.
Infine la visualizzazione delle bande di DNA viene effettuata mediante l’esposizione ai raggi
ultravioletti di un transilluminatore.
M
Maarrccaattoorree m
moolleeccoollaarree
430 bp (YAP+)
150 bp (YAP-)
430 bp (YAP+)
150 bp (YAP-)



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)





![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)

