ECTS - EUROPEAN CREDIT TRANSFER SYSTEM
PROGRAMMA SOCRATES/ERASMUS I ROMA01
CONSIGLIO DI AREA DIDATTICA IN SCIENZE FISICHE E SCIENZE DELL’UNIVERSO
DIPARTIMENTO DI FISICA
ANNO ACCADEMICO 2007-2008
CORSO DI LAUREA
(CdLS) SPECIALISTICA
Denominazione CdLS:
Sede
"Fisica"
Dipartimento di Fisica – P.le A. Moro, 2 – 00185 Roma
Sito Web
Codice CdL
http://www.phys.uniroma1.it
12383
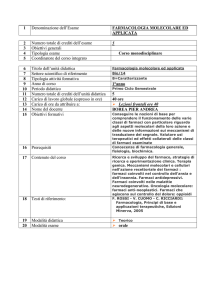
Denominazione
INSEGNAMENTO
Simulazione atomistica
Settore Scientifico-disciplinare (SSD)
Docente(i) titolare (i)
dell’insegnamento
Pre-requisiti
Crediti ECTS
6
Anno di corso
2
Codice esame
FIS/02
1003427
Tipo attività formativa
Semestre
Terzo (ved. Calendario didattico)
Prof. Leonardo Guidoni
Conoscenze di nozioni di base di meccanica statistica. Elementi di base della chimica e della
struttura della materia. Conoscenze di Biofisica strutturale sono utili ma non necessarie.
Il corso si propone di fornire gli strumenti di base della meccanica statistica delle simulazioni
atomistiche al calcolatore di sistemi molecolari. Permetterà di conoscere tecniche ed algoritmi
(conoscenze e competenze) usati correntemente per lo studio delle proprietà di liquidi e macromolecole con particolare
attenzione per i sistemi di interesse biologico.
Le esercitazioni in laboratorio di calcolo sono parte integrale dei corsi e forniranno conoscenze
di base per l’utilizzo pratico di strumenti di pubblico dominio per la manipolazione e la
simulazione di macromolecole (simulazione, visualizzazione, banche dati).
Obiettivi formativi
dell’insegnamento
Programma di massima
Meccanica Statistica e simulazioni al calcolatore. Scale di tempo nella dinamica e nelle
simulazioni di sistemi molecolari. Ergodicità e medie temporali.
Metodi Monte Carlo. Il bilancio dettagliato. L'algoritmo di Metropolis. Differenze tra
Monte Carlo e Dinamica Molecolare. Interazioni molecolari e campi di forza per
biomolecole.
Struttura di un programma di Dinamica molecolare. Formulazione di Liouville della
dinamica. Condizioni al contorno ed iniziali per la Dinamica molecolare. Algoritmo di
Verlet. Velocity-Verlet. Potenziale Lennard-Jones e sistema di unita` ridotte. Dinamica
Browniana. Espansione di Trotter. Multiple time step. Simulazione dell'ensemble NVT.
Le interazioni a lungo raggio. Il troncamento delle interazioni per potenziali a due corpi
tramite l'uso di cut-off. Somme di Ewald. Cenni sul metodo Particle-Mesh-Ewald.
Proprietà statiche e dinamiche. Funzioni di distribuzione.
Rappresentazione di solvente implicito. Il Modello di Born-Onsager. L'equazione di
Poisson-Boltzmann. Calcoli di energia libera con la dinamica molecolare.
Cenni di dinamica Molecolare da principi primi. Dinamica Molecolare Born-Oppenheimer.
Il metodo di Car e Parrinello. Dinamica molecolare mista Quanto/Classica.
Il Protein Data Bank. Rappresentazione grafica di proteine ed acidi nucleici.
Modelli e simulazioni atomistiche in biofisica.
Esercitazioni al calcolarore: Il Protein Data Bank. Visualizzazione grafica della struttura
di proteine ed acidi nucleici. Simulazioni al calcolatore di piccole biomolecole in fase
condensata e delle loro proprietà .
Bibliografia
Understanding Molecular Simulation, D. Frenkel & B. Smit, Academic press 2002. (Varie copie
sono disponibili in biblioteca.) + altro materiale distribuito a lezione
Modalita’ di apprendimento ed insegnamento
Lezioni e
Seminari
Impegno per
l’apprendimento
espresso in ORE
Attivita’
di verifica
Lavori in gruppo –
laboratori
30
Modalità dell’esame e
peso %
Commissione d’esame
Prove in itinere
Prova Scritta
Prova Orale
50
Esercitazioni
Studio
personale
Totale
ore
18
102
150
Tesina o relazione laboratorio
50
L.Guidoni, A. Tebenbaum, G. Ciccotti
Orario delle lezioni
Calendario esami
100 %