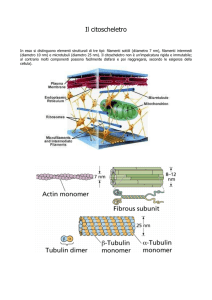
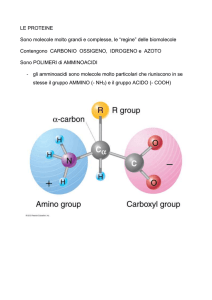
La funzione delle proteine dipende dalla loro
struttura tridimensionale
•
•
•
•
•
La struttura dipende dal ripiegamento di particolari sequenze
aminoacidiche
La sequenza aminoacidica della catena polipeptidica è la struttura
primaria
Regioni diverse della sequenza formano strutture secondarie regolari
(alfa eliche e filamenti beta)
Gli elementi di struttura secondaria si dispongono secondo semplici
motivi strutturali. La struttura terziaria risulta dalla associazione di
più motivi strutturali in una o più unità dette “domini”.
La proteina può essere formata da più catene polipeptidiche disposte in
una struttura quaternaria
Elementi strutturali delle proteine
• L’interno delle molecole proteiche contiene
soprattutto catene laterali idrofobe disposte in
strutture secondarie che permettono di neutralizzare
la polarità dei loro gruppi polari mediante la
formazione di legami idrogeno.
• Esistono 2 tipi principali di struttura secondaria: αeliche e foglietti-β (che costituisocno le regioni del
core), e sono collegate da regioni loop presenti alla
superficie
Gli elementi della costruzione
α-elica
-Presenti nelle proteine quando una stringa di residui consecutivi
presentano tutti coppie di angoli diedri Φ e ψ con valori compresi
tra -60° e -50°.
-Presenta 3,6 residui per spira
-Stabilizzata da legami idrogeno che si formano tra il gruppo C=O
di un residuo n ed il gruppo NH del residuo n+4. In questo modo
tutti i gruppi NH e CO della catena principale sono uniti da
legami idrogeno.
-L’elica è sempre destrorsa
Gli elementi della costruzione
foglietto-β
Rossiossigeno
Bluazoto
Biancoidrogeno
NeroCα cat princ
Violaposizione cat lat
-Costituiti da filamenti β che possono essere tutti
antiparalleli oppure paralleli tra loro.
-Nel foglietto β i filamenti si trovano allineati
giacendo uno accanto all’altro in modo da formare legami
idrogeno tra i gruppi C=O di un filamento β ed i
gruppi NH di un filamento adiacente
I filamenti beta antiparalleli sono tra
gli elementi a più alta stabilità
foglietto β antiparallelo
(direzioni opposte NC CN)
foglietto β parallelo
(medesima direzione biochimica NC)
Gli elementi della costruzione
loop
-tratti di catena apparentemente disorganizzati, di lunghezza variabile
-fanno da collegamento fra α-eliche o filamenti β
-partecipano spesso alla formazione di siti di legame
(come i loop degli anticorpi) o del sito attivo degli enzimi
-si trovano alla superficie della molecola
I diagrammi di topologia
Topologia come gli elementi di struttura secondaria sono connessi tra loro
utili per rappresentare le connessioni tra elementi di struttura secondaria
aspartato
carbamiltransferasi
flavodossina
plastocianina
Gli elementi di struttura secondaria sono
collegati a formare semplici motivi strutturali
• MOTIVO STRUTTURALE semplice
combinazione di pochi elementi
fondamentali di struttura secondaria
• Motivi strutturali semplici possono
combinarsi a formare motivi complessi
• Combinazioni di piccoli motivi strutturali
costituiscono il core dei domini (unità
fondamentali della struttura terziaria)
I 4 motivi strutturali più ricorrenti
Elica-loop-elica
Forcina β
Chiave greca
β-α-β
La combinazione dei motivi strutturali semplici dà
luogo a strutture complesse
Motivo Elica-loop-elica
motivo strutturale specifico
per il legame del DNA
motivo strutturale specifico
per il legame del calcio
(parvalbuina,calmodulina, troponinaC)
Il motivo “EF hand”
-Comprende 2 alfa-eliche:E ed F, che affiancano il loop:
5 residui del loop legano il calcio quindi la loro cat lat
deve contenere un atomo di ossigeno
Elica E direzione che parte dalla base dell’indice
Dito medio regione loop di 12 residui che lega il calcio
Elica F diretta all’estremità del pollice
-Residuo num.6 del loop è sempre glicina
(poco ingombrante)
La mano destra simboleggia
il motivo di legame al calcio
Sequenze consenso dei motivi EF
in 3 proteine diverse
(in arancio aa deputati al legame
con il calcio;
In verdeaa con cat lat idrofobe
destinate a formare core idrofobo
tra le alfa-eliche)
Struttura della troponina C
con i suoi motivi EF
-E’ costituita da 4 motivi EF hand di cui 2
legano il calcio (sfere rosa)
-L’atomo di calcio è legato attraverso
6 atomi di ossigeno, dei quali tre proviengono
dalla cat lat di Asp (D9), Asn (N11) e Asp (D13)
due sono forniti dalla cat lat di Glu (E20),
ed uno dal legame peptidico del residuo 15.
-Al calcio è legata anche una molecola d’acqua.
Quindi questo motivo, per espletare la sua funzione,
necessita sia di una specifica conform della catena principale sia
della presenza di un peculiare raggruppamento di catene laterali.
Il motivo elica-loop-elica fornisce l’impalcatura che ancora i ligandi
degli ioni calcio in posizione corretta per poter legare e liberare questi ultimi
motivo elica-loop-elica legante il DNA
Motivo elica-loop-elica legante il
DNA presente nella proteina Cro del fago λ….
…legato al DNA
Forcine β
forcina β come foglietto isolato
forcina β come parte di un
foglietto più complesso
Motivo a greca
-Quando 4 filamenti beta antiparalleli si dispongono
secondo un modello simile all’unità ripetitiva di un tipico
disegno ornamentale caratteristico dell’arte dell’antica
Grecia, noto come “Greca”.
-Si basa sull’iniziale formazione di una lunga struttura
antiparallela in cui i loop sono presenti al centro di entrambi
i filamenti beta.
-Il motivo a greca si originerebbe in conseguenza di
cambiamenti strutturali nelle regioni loop interposte tra i
filamenti beta 1 e 2 e nelle regioni
interposte tra i filamenti 3 e 4. Per effetto di ciò, la
struttura si ripiega verso il basso ed il filamnto beta 2 va
ad associarsi col filamento 1 formando con questo legami
idrogeno.
Ipotetica modalità di ripiegamento di
struttura a forcina che porta
alla formazione del motivo a greca
Motivo a greca
a) diagramma topologico della greca
b)motivo a greca presente
nell’enzima nucleasi Staphylococcus
.
Il motivo a greca fornisce un
semplice modo di collegare filamenti beta
antiparalleli che si trovino su lati opposti di una struttura a bott
Motivo β-α-β
a) Schema del percorso della catena principale
b) Diagrammi topologici del motivo β-α-β
-Permette il collegamento tra filamenti beta paralleli.
-Consiste di 2 fil beta paralleli, un’alfa elica e due regioni loop.
-L’alfa elica collega l’estremità carbossilica di un filamento beta
con quella aminica del filamento beta successivo
-Il loop che collega il terminale carbossilico del filamento
beta con quello aminico dell’alfa elica spesso è implicato nella
formazione del sito funzionale
Motivo β-α-β
a) Modalità di collegamento destrorsa
b) Collegamento di tipo sinistrorso
,
In linea di principio questo motivo può presentare due diverse disposizioni:
una in cui l’elica si trova al di sopra del piano della pagina (“connessione
destrorsa” perché presenta il medesimo orientamento di una alfa elica
destrorsa); ed una in cui l’elica si trova al di sotto del piano della pagina
(sinistrorsa).
In realtà la connessione è sempre destrorsa, con l’unica eccezione della
subtilisina.
I DOMINI
Strutture alfa elica superavvolta
fascio a 4 eliche
ripiegamento della globina
Strutture alfa-beta
TIM barrel
foglietto alfa- beta aperto
ferro di cavallo
Strutture beta
antiparallele Beta barrel greca
Up-and-down
Jelly roll
Strutture beta
parallele
Elica beta a 2 foglietti
Elica beta a 3 foglietti
Il dominio è
• unità compatta e semi-independente
(Richardson, 1981).
• unità stabile di una proteina che può
ripiegarsi autonomamente (Wetlaufer,
ti1973).
• modulo funzionale ed evoluzionistico
ricorrente (Bork, 1992)
icazione dei domini è essenziale:
• I domini hanno dimensioni variabili. Circa il 50% delle proteine
studiate hanno domini compresi fra 51 e 150 amminoacidi. Sono note
proteine con molti domini (13) e le interazioni interdominio sono le
stesse che stabilizzano le loro strutture interne.
• i cuori idrofobici sono fondamentali per la stabilità dei domini (nelle
sezioni proteiche i residui idrofobici sono in giallo)
lisozima da Gallus
gallus pdb: 1hel
esochinasi da Homo sapiens pdb:
Non tutti i domini sono costituiti da tratti continui di
polipeptide. In alcune proteine un dominio è interrotto da un
tratto di sequenza che si ripiega in un dominio separato,
dopo il quale continua il dominio originale.
alanina racemasi da Bacillus stearothermophilus
pdb: 1sft
Le proteine multidominio si sono probabilmente evolute dalla
fusione dei geni che un tempo codificavano per proteine separate.
tioesterasi da E. coli (pdb:
1c8u)
2 domini
fusi
tioestere deidratasi da E.
coli (pdb: 1mkb)
omodimero con ciascun
monomero simile ai domini
della tioesterasi
Il numero dei fold (motivi strutturali) proteici è vasto
ma limitato.
triptofano sintasi da
Salmonella typhimurium
α-galattosidasi da
Homo sapiens
Elica superavvolta
Struttura ad elica superavvolta.
Un’alfa elica isolata presenta una ridotta stabilità in soluzione.
Perciò, nelle proteine, le alfa eliche sono stabilizzate dal reciproco
impaccamento mediato dall’interazione delle catene idrofobe.
Il modo più semplice di realizzare tale stabilizzazione è quello di
associare 2 alfa-eliche, in modo da formare una struttura superavvolta.
Una strutura superavvolta sinistrorsa formata da 2 alfa eliche destrorse
riduce il numero di residui per giro in ognuna delle eliche costitutive da
3,6 a 3,5, in modo che le interazioni tra le catene laterali delle 2 eliche
vengono a ripetersi ogni 7 residui. Ciò si riflette nelle sequenze
aminoacidiche delle due catene, le quali risultano ripetitive,
con un periodo di ripetitività di 7 residui.
Elica superavvolta
I residui aminoacidici nell’unità ripetitiva vengono indicati
come “a-g” ed il residuo “d” è idrofobo (di solito è una leucina).
La regione idrofoba tra le due alfa eliche è completata
dai residui “a” che sono idrofobi anch’essi.
Ripetitività degli aa in un’alfa elica
superavvolta:
un residuo di leucina si ripete ogni 7
aminoacidi
Impaccamento delle
catene laterali idrofobe tra 2
alfa-eliche in una struttura
Ruolo delle interazioni elettrostatiche
a) Visione schematica dall’alto di una ripetizione di sette aa.
b) Visione schematica laterale di una struttura superavvolta
I residui “e” e “g” che orlano il
core idrofobo sono carichi e le loro
catene forniscono interazioni ioniche (ponti salini) tra le α eliche.
Fascio a 4 eliche
-Due alfa eliche impaccate insieme in una struttura
superavvolta sono elementi costitutivi di un dominio ma
non sono sufficienti a formare un dominio completo.
-Infatti, il dominio ad alfa elica più semplice consiste
in 4 alfa eliche disposte in un fascio con l’asse di ognuna
parallelo a quelli delle altre.
-Le alfa eliche sono disposte in modo tale che le eliche
consecutive nella sequenza aminoacidica vengono ad
essere adiacenti.
Dispiegamento della catena
polipeptidica
in un dominio costituito da
un fascio di 4 eliche
Fascio a 4 eliche
Proiezione del motivo su un piano
perpendicolare all’asse del fascio
Le catene laterali di ciascuna elica sono disposte in modo tale
che le catene laterali idrofobe si trovano comprese nello spazio
tra le eliche, mentre le catene laterali idrofile vengono a trovarsi alla
superficie del fascio. In questo modo, nella parte centrale del fascio
viene a formarsi un core idrofobo in cui le catene laterali si trovano
ammassate così fittamente da escludere la presenza di molecole d’acqua.
Cerchi grandi catena principale delle alfa–eliche
Cerchi piccoli catene laterali: verdi (catene laterali idrofobe ammassate);
rosse (cat lat idrofile che si trovano esposte alla superficie del fascio)
Fasci a 4 eliche come singolo dominio di proteine
monomeriche, o come motivo di dimerizzazione
Il Ripiegamento della globina
Fascio di 8 eliche
Collegate da brevi loop
Formazione di una tasca
idrofobica in cui si trova
il sito attivo
Strutture a dominio alfa-beta
(foglietto β centrale di tipo parallelo o misto circondato da α eliche)
Modalità di connessione β-α-β
Le tre strutture alfa-beta sono tutte costituite da motivi β-α-β
collegati tra loro in modo che i filamenti β vengono ad essere paralleli.
2 motivi β-α-β si possono collegare in 2 diversi modi:
a) le alfa eliche si trovano tutte sul medesimo piano del foglietto
(TIM barrel e ferro di cavallo);
b) le alfa eliche si trovano sul piano opposto (foglietti beta aperti)
TIM Barrel
Gli 8 filamenti beta racchiudono
un core idrofobo impaccato
costituito dalle catene laterali dei
residui presenti in filamenti beta
alternati.
Il core è disposto a formare tre
strati, dove ogni strato contiene
4 catene laterali provenienti dai
residui presenti in filamenti beta
alternati.
Struttura formata da 8 filamenti beta paralleli
Filamento 8 forma legami idrogeno col filamento1
Per formarla sono necessari almeno 200 aa
Presente in molti enzimi
Filamenti beta ed alfa eliche impalcatua strutturale
Regioni loop siti attivi
–
Rappresentazione vista dall’altodella strutturaa botte del
sito attivo dell’enzima RuBisCo.
Il sito di legame per il substrato (in rosso)
è generato da numerose catene laterali
cariche (in blu) presenti su differenti loop.
Nel TIM barrel il sito attivo si trova
in una tasca formata dalle regioni loop
che collegano le estremità carbossiliche
dei filamenti β con le adiacenti α-eliche.
La Piruvato chinasi
contiene diversi domini, uno dei quali con
struttura TIM barrel
• La struttura di questo enzima
illustra perfettamente in che
modo una lunga catena
polipeptidica può ripiegarsi a
formare domini di tipo
diverso dal punto di vista
strutturale.
• La funzione enzimatica
risulta sempre associata con
il dominio che presenta
struttura a botte.
Il foglietto alfa-beta aperto
Nei domini con struttura α/β aperta, il sito attivo si trova in una fessura
localizzata esternamente all’estremità carbossilica dei filamenti β.
Questa fessura è formata da 2 regioni loop adiacenti che collegano
i due filamenti con α eliche presenti su facce opposte del foglietto β.
Ciò è illustrato dalle dita curvate delle due mani, in cui la metà superiore
delle dita rappresenta le regioni loop mentre la metà inferiore
rappresenta i filamenti β. Il cilindro rappresenta un ligando posizionato
nella fessura di legame.
Esempi di tipi diversi di strutture α/β aperta
flavodossina e adenilato chinasi
esochinasi e fosfoglicerato mutasi
Ripiegamento a Ferro di cavallo
Struttura dell’inibitore della ribonucleasi, Ruolo dei residui conservati di leucina dei
costituita da motivi β-loop-α ripetuti,
motivi ricchi di leucina nella
ricorda un ferro di cavallo formato
stabilizzazione del modulo strutturale
all’interno da un foglietto β parallelo a
β-loop- α
17 filamenti e all’esterno da 16 α eliche.
Il beta Barrel
• Comprende enzimi, proteine di trasporto, anticorpi,
proteine virali di rivestimento.
• Core: costituito da filamenti β (da 4-5 a più di 10),
disposti in modo antiparallelo a formare 2 foglietti β
collegati ed impaccati l’uno con l’altro.
• Le strutture β antiparallele presentano un core di
catene laterali idrofobe all’interno di una struttura a
botte, formato dalle catene laterali dei residui
presenti nei filamenti β , mentre la superficie è
costituita da residui provenienti sia dai filamenti β
che da regioni loop
β
La maggior parte delle strutture
rientra in pochi
gruppi con topologia uguale o simile
motivi a greca
up and down
Ogni filamento β è
collegato al successivo
attraverso una breve
regione loop
Quando in una struttura a
β barrel a 8 filamenti,
il numero n è collegato
al filamento n+3
jelly roll
La catena polipeptidica
si avviluppa attorno ad
un’ideale struttura a
botte centrale come un
dolce arrotolato coperto
di gelatina
Un esempio di struttura a β-barrel
greca: la SOD
8 filamenti β antiparalleli disposti attorno alla superficie di una botte ideale.
La struttura, a " β -barrel" , evidenzia un motivo topologico a “greca”,
comune a molti sistemi biologici di grande interesse, tra cui, ad esempio,
le immunoglobuline.
RBP lega il retinolo all’interno di una
struttura a botte up and down
8 filamenti β antiparalleli ruotati e ripiegati
in modo che la struttura può anche essere vista
come 2 foglietti β (verde e blu) impaccati
l’uno contro l’altro.
Alcuni dei filamenti β ruoati (in rosso)
partecipano alla formazione
di entrambi i foglietti β.
La vitamina A si trova legata all’interno
della struttura a botte tra i 2 foglietti β,
in modo da esporre alla superficie della proteina
l’unica porzione idrofila della molecola
(coda contenente un gruppo OH)
La neuraminidasi si ripiega a
formare foglietti beta up and down
Presente nel virus dell’influenza
Catalizza idrolisi dell’acido sialico
Omotetramero costituito da
4 catene polipeptidiche identiche,
ognuna di 470 aa
Ogni monomero è ripiegato a formare
una superstruttura a botte
costituita da 24 filamenti β
disposti a formare 6 motivi strutturali
simili, ognuno dei quali contiene 4
filamenti β che rappresentano le pale
della struttura a forma di elica
Nella neuraminidasi i motivi strutturali generati col
ripiegamento formano una superstruttura a forma di
elica
Tetramero:
4 domini ognuno
con struttura ad
elica a 6 pale
Monomero:
(Dominio:elica a 6 pale)
Topologia del
monomero
Motivo:
(foglietto β:pala dell’elica)
Ognuna delle 4 subunità del tetramero è ripiegata
a formare un singolo dominio costituito da 6 motivi
strutturali simili strettamente impaccati.
Tale motivo strutturale è un foglietto β a 4 filamenti
antiparalleli up-and-down.
Questi 6 foglietti β sono disposti in modo da formare
le sei lame di un’elica a 6 pale
La regione strutturale e la regione del sito
attivo risultano ben separate.
In conseguenza della simmetria
senaria dei 6 motivi strutturali
a foglietti β, le 12 regioni loop
derivate dai foglietti si trovano
dalla stessa parte della molecola
Le regioni loop che collegano i
motivi strutturali formano
un’ampia tasca ad imbuto
contenente il sito attivo
I filamenti beta rappresentano lo scheletro
strutturale sul quale è incernierato il sito attivo
costituito da loop di connessione tra un elemento
e un altro elemento
L’emoagglutinina si ripiega a formare un
motivo strutturale a jelly roll
Formata da 3 subunità, ognuna delle quali è
ancorata alla membrana pericapsidica del
virus dell’influenza. Le teste globulari
contengono i siti per i recettori che legano
i residui di acido sialico presenti alla
superficie delle cellule eucariotiche
Singola subunità
dell’emoagglutinina
del virus
dell’influenza
Il sito di legame per il recettore è
formato dal dominio jelly roll
La testa globulare di ogni subunità
dell’emoagglutinina
ha una struttura a botte jelly roll distorta.
Il sito di legame è localizzato all’apice della
subunità, all’interno della struttura a botte jelly roll.
Domini ad eliche β parallele
• Scoperta nel 1993 in California
• Presente in molte proteasi batteriche
extracellulari e nella proteina della coda del
batteriofago P22
• La catena polipeptidica risulta ripiegata a
formare un ampio superavvolgimento ad elica
formato da filamenti β separati da regioni
loop
• I filamenti β si allineano a formare 2 o 3
foglietti β paralleli che racchiudono un core
riempito dagli atomi delle catene laterali.
Eliche beta parallele a 2 foglietti
Elica β a 2 foglietti dove
sono mostrati 3
superavvolgimenti
completi dell’elica.
- Ogni unità strutturale è costituita da 18 residui
che formano una struttura β-loop-β.
- Ogni regione loop contiene sei residui di sequenza
Gly-Gly-X-Gly-X-Asp-H-U-X dove U è un
aminoacido con catena ingombrante e idrofobica,
spesso una leucina.
-Ioni calcio sono legati ad entrambe le regioni loop,
stabilizzandole.
Eliche beta parallele a 3 foglietti
- 2 dei foglietti β sono paralleli
l’un l’altro e perpendicolari al terzo.
-Ogni giro dell’elica contiene 3 brevi
filamenti β ognuno di 3-5 residui
collegati da 3 regioni loop.
-Uno dei loop è sempre costiuito solo
da 2 residui, mentre gli altri 2 sono
più lunghi e variano in dimensioni.
-Quindi l’elica β comprende
comprende 3 foglietti
β paralleli grossolanamente disposti
come le tre facce di un prisma
Struttura della pectato liasi C
CATH
http://www.biochem.ucl.ac.uk/bsm/cath
• Banca Dati secondaria (viene fatta prima
un’analisi, una selezione ed infine viene
eseguito l’immagazzinamento dei dati)
• Classificazione delle proteine in base a
similarità di sequenza e strutturali
•La funzione non è presa in considerazione
in questa banca dati
Livelli di classificazione
• Class (α, β, mixed α-β, low 2ndry structure
content)
• Architecture: stesso arrangiamento spaziale
tra le strutture secondarie, ma connessioni
diverse
• Topology: stesso fold
• Homologous superfamily: proteine con
probabile ancestore comune
• Sequence Family: proteine con identità di
sequenza (id >= 35%) e funzionalità
CLASS (C-level)
• Assegnata in modo automatico
• Class : secondary structure composition and contacts
– Class1 : Mainly Alpha
– Class2 : Mainly Beta
– Class3 : Mixed Alpha- Beta
– Class4 : Few Secondary Structures
Class1
Class2
Class3
Class4
Architecture (A-level)
-forma generale del dominio
-descrizione dell’arrangiamento della struttura secondaria
indipendentemente dalle connessioni
- Effettuata manualmente
Topology (T-level)
Proteine che hanno la stessa topologia
hanno fold simile e core abbastanza
conservato, e quindi hanno strutture
che sono simili ma con funzioni diverse.
prende in considerazione le connessioni tra elementi di struttura secondaria,
ovvero le connessioni tra motivi strutturali
Homologous superfamily (H-level)
Raggruppa insieme proteine che si pensa abbiano un ancestore comune e che
quindi sono omologhe. In questo modo vengono definiti dei gruppi di
superfamiglie omologhe.
Metodi di classificazione
-avviene in maniera gerarchica, così che ogni proteina è riconosciuta
attraverso un numero.
-es: 1.10.490.20 vuol dire che la proteina appartiene alla Classe 1,
Architettura 10, Topologia 490 e Homologia 20.
-ad ogni numero corrisponde una ed una sola proteina
Criteri di classificazione
Step 1 : si selezionano serie di strutture su PDB
(risolte per diffrazione o NMR a risoluzione di almeno
3.0 Å)
Step 2: Comparazione di sequenze: proteine con
identità di sequenza >35% vengono messe a livello S
Step 3: dividere proteine in domini per poi analizzarle
singolarmente. L’assegnamento della classe è
automatico perché utilizza una procedura che esamina
la composizione della struttura secondaria
analizzando il valore degli angoli Φ e ψ .
Step 4: Comparazione di struttura per definire H e T
in maniera automatica utilizzando il programma SSAP
SSAP
(Sequence Structure Alignement Program)
• Programma di comparazione strutturale
• Compara distanze tra residui in modo sequenziale.
• Il parametro utilizzato per la classificazione è il
numero S che è proporzionale all’ ’inverso della
sommatoria di queste differenze. Tanto è più piccola
questa differenza tanto più saranno simili le strutture
e tanto più S sarà grande. Se S è uguale a 100 le
strutture sono completamente identiche.
• La soglia è S=70 per il livello T, e di S= 80 per il
livello H. Quindi tra 70 e 80 la proteina viene
classificata nel livello T o da 80 in su viene
classificata nel livello H.
Modalità di Ricerca
• Codice PDB
• Codice CATH
• Parole chiave
• Accesso tramite link da altri database
(es. PDB)