
Materiale online - CAPITOLO 9 1
ISBN 978-88-08-18686-7
9
REPLICAZIONE
DEL DNA
◆ 9.1 Le proteine della replicazione
◆ 9.2 L’inizio della replicazione
L’origine di replicazione di E. coli
L’inizio della replicazione a oriC
Meccanismi di controllo dell’inizio della replicazione
L’inizio della replicazione di batteri con più cromosomi
● scheda: L e Dna polimerasi
● scheda: Pol I, l’enzima “eureka” e le altre
polimerasi
● scheda: Altre proteine necessarie per la
replicazione
● scheda: Regolazione dell’inizio della replicazione
nei batteri
● scheda: Il primosoma nei batteri
◆ 9.3 Innesco della sintesi e allungamento del
DNA
● scheda: L’assemblaggio ciclico della primasi e
DNA polimerasi III sul lagging strand e
sintesi dei frammenti di Okazaki
◆ 9.4 Terminazione e risoluzione (separazione)
dei nuovi cromosomi
◆ 9.5 La replicazione degli elementi
extracromosomali e il suo controllo
La replicazione del fattore F
La replicazione di ColE1
Il controllo del numero delle copie del DNA
La replicazione dei cromosomi lineari
◆ 9.6La replicazione negli Archaea
letture consigliate
Amaldi, F., Benedetti, P., Pesole, G., Plevani, P., Biologia molecolare, 2a edizione, Casa Editrice Ambrosiana, Milano, 2014.
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 1
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 2
ISBN 978-88-08-18686-7
L
a replicazione dei cromosomi di batteri, virus e plasmidi avviene in modo analogo alla replicazione del
materiale genetico degli eucarioti. Anzi, lo studio della replicazione del DNA di E. coli e dei suoi elementi genetici
(virus e plasmidi) ha permesso di decifrare il quadro generale
di questo complesso meccanismo. L’apparato di replicazione,
detto replisoma, ha un’architettura simile nei procarioti e negli
eucarioti e la sua funzione essenziale è copiare in modo efficiente e fedele l’informazione contenuta nelle molecole di DNA.
Dal punto di vista biochimico si tratta di un processo di polimerizzazione che può essere suddiviso in tre fasi distinte:
• un evento che determina l’inizio del processo;
• una reazione iterativa che porta all’allungamento della catena del polimero;
• e una fase conclusiva che la interrompe e ne consente la terminazione.
Nonostante la relativa semplicità della reazione biochimica di
polimerizzazione (formazione di un legame fosfodiestere), la
complessità del fenomeno è facilmente intuibile. Basti pensare
ai cambiamenti dinamici che la replicazione impone alla struttura del DNA, ai problemi connessi con la velocità e soprattutto la precisione richiesta per la copiatura delle due eliche, al riconoscimento di una piccola regione, all’interno di un enorme
polimero monotono, dove avviare il processo coordinandolo
con il ciclo cellulare.
Le caratteristiche generali della replicazione nei procarioti
possono essere riassunte nei punti seguenti.
1. La sintesi del DNA di batteri e virus inizia di norma a livello di una sequenza specifica unica, chiamata origine di
replicazione (sito ori o oriC) (fig. 9.1a). Negli eucarioti,
al contrario, i cromosomi hanno molteplici origini. Il sito
ori è la struttura sulla quale si esercita l’intera regolazione
del processo di sintesi. Siti ori multipli possono essere presenti ma di norma non sono attivi contemporaneamente
e, specialmente in plasmidi e batteriofagi, possono avere
ruoli diversi.
2. Con l’inizio della replicazione i due filamenti sono separati localmente nella regione ori portando alla formazione
di una sorta di bolla a singolo filamento. Ad ogni estremità
della bolla si forma una struttura biforcata, costituendo la
cosiddetta forca replicativa, su cui possono localizzarsi le
proteine che costituiscono il replisoma (ossia la macchina replicativa), che comprende la DNA polimerasi, l’enzima che catalizza la polimerizzazione.
3. La replicazione può procedere dal sito di inizio in modo
unidirezionale o bidirezionale. Nella replicazione bidirezionale due forche replicative avanzano una in senso
orario, l’altra in direzione opposta e i due filamenti sono
copiati man mano che la replicazione procede. Nella replicazione unidirezionale il replisoma è associato a una
sola delle due biforcazioni (si ha quindi una sola forca replicativa (fig. 9.1b). I cromosomi batterici replicano normalmente in modo bidirezionale mentre situazioni diverse si osservano in fagi e plasmidi. Ad esempio, il plasmide
F replica in modo bidirezionale ma quando deve trasferirsi
a)
b)
Bidirezionale
Due
forche
replicative
Origine
Unidirezionale
Origine
Una forca
replicativa
Figura 9.1 MODELLO DI REPLICAZIONE DEL CROMSOSOMA DI E. COLI. (a) La replicazione parte da oriC e procede in modo bidirezionale (senso orario per una
forca replicativa, antiorario per l’altra) fino al raggiungimento della regione del
cromosoma detta di terminazione, dove le due forche replicative s’incontrano.
(b) Un confronto tra due tipi di replicazione nei procarioti: replicazione bidirezionale e unidirezionale (questa è caratterizzata dalla presenza di una sola forca
replicativa).
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 2
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 3
ISBN 978-88-08-18686-7
in un altro batterio tramite la coniugazione passa a una forma speciale di replicazione unidirezionale, detta “cerchio
rotante”, utilizzando un secondo sito ori.
4. La replicazione è semiconservativa, ossia le due molecole
di DNA prodotte sono costituite ciascuna da un filamento
parentale e da un filamento neo-sintetizzato (fig. 9.2).
5. La replicazione è semidiscontinua: un filamento è copiato
in modo continuo per lunghi tratti, l’altro per brevi tratti discontinui adiacenti. I frammenti del DNA complementare
neosintetizzato vengono poi saldati fra loro (fig. 9.2).
6. L’apparato di replicazione è un complesso macromolecolare, costituito da numerose subunità proteiche, in cui
vengono coordinate le molteplici attività dell’apparato
stesso.
7. Il replisoma procede in modo sequenziale (in gergo: processivo), sintetizzando lunghe catene polinucleotidiche.
Questo significa che molte proteine implicate nella replicazione formano complessi che tendono a rimanere associati al filamento di DNA per tutto il processo di replicazione. Questa proprietà contribuisce a rendere veloce la
replicazione dell’intero cromosoma.
8. Il tasso di sintesi del DNA è relativamente costante con
un ritmo di progressione dalle forche replicative che varia
tra i diversi batteri. In E. coli, ad esempio, è di circa 1000
nt s–1 (un cromosoma di circa 5000 kb è replicato bidirezionalmente in circa 40 min), in Pyrococcus abyssi 300 nt
s–1, mentre in Mycoplasma capricolum è di circa 100 nt s–1.
9. La replicazione bidirezionale termina in siti del cromosoma chiamati ter, in posizione diametralmente opposta al
sito ori, dove lega la proteina Tus (fig. 9.3).
10. La replicazione degli elementi extracromosomici è spesso limitata a una singola specie ospite o a poche a questa
correlate. Esistono tuttavia plasmidi ad “ampio spettro d’ospite”, capaci di replicare e mantenersi in specie batteriche
Frammenti di Okazaki del
filamento a replicazione discontinua
5′
3′
1° 5′ 3′
Forca
replicativa
2° 5′ 3′
Filamento
a replicazione
continua
Primer
a RNA
3°
5′
Direzione della replicazione
Filamento parentale
5′
3′
Figura 9.2 REPLICAZIONE SEMICONSERVATIVA E SEMIDISCONTINUA. La polimerizzazione dei nucleotidi nei due nuovi filamenti, utilizzando i vecchi filamenti come
stampo, procede in direzione 5′ → 3′ e richiede un solo innesco per il filamento
leading (sintetizzato in modo continuo), e più inneschi per il filamento lagging
(sintetizzato in modo discontinuo). I numeri si riferiscono all’ordine di sintesi degli
inneschi e della conseguente formazione dei frammenti di Okazaki.
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 3
rrnA
rrnC
oriC
90.5
89.8
86.5
84.5
84
5.1
13
Genoma
di E. coli
rrnD
terI
72.1
23
27
28
31
34
36
56.1
rrnG
terE
terD
terA
terH
terC
terB
49 48
terG terF
Figura 9.3 TERMINAZIONE DELLA REPLICAZIONE. La replicazione si blocca quando
il replisoma incontra il complesso Tus-ter. Questo complesso agisce bloccando l’elicasi DnaB. Le frecce indicano il senso di trascrizione degli operoni
codificanti per gli RNA ribosomali, che concorda con la direzione della forca
replicativa.
filogeneticamente distanti. In questi casi si stabiliscono
interazioni versatili tra le proteine codificate dal plasmide, l’origine di replicazione e vari fattori della replicazione
dell’ospite.
In questo capitolo la replicazione del DNA sarà affrontata
essenzialmente a due livelli: il primo analitico, che procede
mediante una dissezione dell’apparato di replicazione per
evidenziarne le singole attività enzimatiche; il secondo funzionale, che integra le attività delle varie proteine del replisoma per giungere alla descrizione del meccanismo stesso di
replicazione. Vedremo poi che la replicazione è una funzione
interconnessa ad altre, come la ricombinazione e la riparazione, non solo perché agiscono sullo stesso substrato, la molecola di DNA, ma anche perché, durante il loro svolgimento,
alcuni passi richiedono l’intervento delle altre (► capp. 10
e 11).
9.1Le proteine della replicazione
3′
5′
3′
rrnH
0/100
rrnE
rrnB
Le varie fasi della replicazione richiedono l’attività di numerose proteine specifiche che svolgono diverse operazioni complesse e tra loro integrate: riconoscere il sito di origine, convertire il DNA nella configurazione topologica adatta, separare
i filamenti della doppia elica, proteggere il DNA a singolo filamento dall’attacco delle nucleasi, catalizzare la polimerizzazione dei nucleotidi, riportare il DNA replicato nella sua corretta
configurazione topologica, decatenare le molecole di DNA al
termine del processo ecc.
Molte di queste proteine sono associate in due complessi
strutturali e funzionali: il primosoma, coinvolto nell’inizio
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
ISBN 978-88-08-18686-7
Materiale online - CAPITOLO 9 4
Movimento della
forca replicativa
Figura 9.4 IL REPLISOMA DI E. COLI. Il replisoma è l’apparato di replicazione, un
macchinario localizzato a livello delle due forche replicative. I costituenti principali sono la primasi DnaG e l’elicasi DnaB. L’oloenzima della DNA Pol III è formato da due corpi centrali (uno per ogni filamento stampo) uniti dalla subunità
τ (tab. 9.3). Il replisoma occupa una posizione fissa nella cellula e il DNA scorre
al suo interno. Secondo il modello detto “a trombone” (vedi testo) l’antiparallelismo dei due filamenti richiede che il lagging strand formi una sorta d’ansa in
modo tale da permettere la localizzazione e l’orientamento della subunità della
DNA Pol III nella stessa direzione rispetto a quella presente sul leading strand e
procedere in modo concertato in direzione 5′ → 3′. L’elicasi si lega a una singola
elica di DNA in una regione già aperta e scalza l’altra elica in modo processivo
(vedi didascalie delle figg. 9.2, 9.7 e 9.13).
Topoisomerasi
Elicasi
Primasi
Primer a RNA
Dimero di DNA
Polimerasi III
Proteine che legano
DNA a singola elica
Primer a RNA
Frammento
di Okazaki
Filamento
a replicazione
immediata
(leading)
Morsetti mobili
Polimerasi I
Filamento
a replicazione
ritardata (lagging)
Ligasi
della replicazione, e il replisoma, l’apparato che fa procedere questo processo lungo l’intera molecola stampo. Inoltre,
le proteine della replicazione intervengono anche nel “controllo di qualità” e nella “risoluzione di problemi accidentali”
che possono insorgere durante il processo (► capp. 10 e 11
e fig. 9.4).
Le principali proteine della replicazione sono brevemente presentate nella tabella 9.1.
9.2L’inizio della replicazione
Nel 1963 François Jacob, Sidney Brenner e François Cuzin
proposero il modello del replicone (replicon) come possibile sistema di regolazione della replicazione del cromosoma
batterico. Il modello supponeva che un elemento genetico,
il replicatore, fosse il bersaglio di una proteina attivatrice che
agiva positivamente in trans per scatenare l’inizio della repli-
Tabella 9.1 PRINCIPALI PROTEINE COINVOLTE NELLA REPLICAZIONE.
Proteine
N. molecole per cellula
Gene
Funzione
DnaA
DnaB
DnaC
DnaT
non noto
20
dnaA
dnaB
dnaC
dnaT
Proteina iniziatrice che separa i doppi filamenti del DNA in oriC
DNA elicasi
Chaperone di DnaB
Componente del primosoma
SSB
500
ssb
Proteine che legano DNA a singola elica (ssDNA)
Topoisomerasi
Topoisomerasi I
DNA girasi
– subunità α
– subunità β
topA
Topologia del DNA, superavvolgimento
250
gyrA
gyrB
DNA Polimerasi
DNA Pol III
DNA Pol I
20
300
Vari geni
polA
DNA polimerasi replicativa
gap filling, rimozione del primer
DNA primasi
75
dnaG
Sintesi del’RNA primer
dnaQ
Degradano il DNA a partire da un’estremità
ligA
Lega in modo covalente i frammenti di Okazaki
hupA, hupB
priA
priB
priC
tus
Histone-like si lega al DNA
Assemblaggio del primosoma, elicasi 3′ → 5′
Assemblaggio del primosoma
Assemblaggio del primosoma
Proteina di terminazione, controelicasi
Esonucleasi
DNA ligasi
Ligasi
Altre
HU
PriA
PriB
PriC
Tus
300
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 4
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 5
ISBN 978-88-08-18686-7
LE DNA POLIMERASI
all’enzima. Il nucleo enzimatico è costituito da tre subunità: α (la polimeLe DNA polimerasi (Pol) sono enzimi che catalizzano la sintesi del DNA
rasi vera e propria, codificata dal gene dnaE ), ε (un’esonucleasi 3′ → 5′
(tab. 9.1). Questi enzimi leggono il DNA stampo e utilizzano questa inforprodotta del gene dnaQ ) e θ (che stimola l’attività esonucleasica in direziomazione per sintetizzare un nuovo filamento complementare allo stampo.
ne 3′ → 5′, prodotta dal gene holE ). Gli altri tre sotto-assemblaggi sono:
È interessante notare che nessuna DNA polimerasi nota può iniziare una
catena de novo formando un legame fosfodiestere a partire da due de1. il sliding clamp o anello . Si tratta di una sorta di morsetto
sossiribonucleotidi trifosfati, mentre può aggiungere desossiribonucleosidi
scorrevole attorno al filamento di DNA. L’anello si lega al nucleo enziliberi all’estremità 3′–OH di una molecola di acido nucleico (DNA o RNA)
matico e lo mantiene strettamente associato al DNA. Questa struttura
preesistente (molecola di innesco o primer) o a un gruppo –OH di una
anulare, costituita da due omodimeri della subunità β, può, mediante
proteina. Vedremo oltre come il problema di iniziare de novo la sintesi di
un cambiamento conformazionale, aprirsi per accogliere il filamento di
una molecola di DNA è stato risolto in vari modi.
DNA appena duplicato. Le caratteristiche di questo morsetto (capacità
In un batterio possono essere prodotti diversi tipi di DNA polimerasi. In E.
di scorrere lungo il DNA e di legare il nucleo enzimatico) sono alla base
coli, ad esempio, ce ne sono cinque diverse con vari ruoli (► Pol I, l’endella processività dell’enzima (fig. 9.6)
zima Eureka e le altre polimerasi e tab. 9.2). Di queste, Pol III e Pol I
sono indispensabili per il processo di replicazione mentre le altre
Tabella 9.2 CONFRONTO TRA DIVERSE DNA POLIMERASI DI E. COLI.
(Pol II, Pol IV e Pol V) intervengono in processi di correzione di
DNA polimerasi
errori e riparazione di danni al DNA.
I
II
III
Pol III è la polimerasi replicativa. Questo enzima ha una velocia
Gene
polA
polB
polC (dnaE)
tà di polimerizzazione compresa tra 250 e 1000 nucleotidi al
secondo ed è straordinariamente processivo: infatti, durante la
Numero di subunità
1
≥4
≥ 10
replicazione Pol III può rimanere associata al DNA e alle altre
103 000 88 000b 830 000
Mr
proteine del replisoma fin dopo aver aggiunto oltre mezzo miEsonucleasi 3′ → 5′ (proofreading)
Sì
Sì
Sì
lione di nucleotidi, mentre questo valore è di 2-300 per Pol I e
Esonucleasi 5′ → 3′
Si
No
No
di 1500 per Pol II.
L’oloenzima Pol III di E. coli è un complesso asimmetrico costiVelocità di polimerizzazione (nucleotidi/sec) 16-20
40
250-1000
tuito da vari peptidi di diverso tipo che formano quattro sottoProcessività (nucleotidi aggiunti prima che
3-200
1500
≥ 500 000
assemblaggi (tab. 9.3 e fig. 9.5). Durante la replicazione sono
la polimerasi termini la polimerizzazione)
presenti due copie dell’oloenzima Pol III, una per ogni filamento
a
Per enzimi con più di una subunità, il gene elencato codifica la subunità con attività di
di DNA che funge da stampo.
polimerizzazione. Da notare che dnaE è la vecchia denominazione del gene ora definito
come polC.
Dei quattro sotto-assemblaggi, uno è il nucleo enzimatico, gli
b
Riferito alla sola subunità di polimerizzazione. La DNA polimerasi II condivide diverse
subunità con la DNA polimerasi III, comprese le subunità β, γ, δ, δ’, χ e ψ.
altri tre hanno un ruolo strutturale e conferiscono processività
Tabella 9.3 SUBUNITÀ DELLA DNA POLIMERASI III DI E. COLI.
Subunità
Numero di subunità
per oloenzima
Mr della
subunità
Gene
α
2
132 000
polC
(dnaE)
ε
2
27 000
dnaQ
(mutD)
θ
2
10 000
holE
τ
2
71 000
dnaX
γ
2
52 000
dnaX*
δ
1
35 000
holA
δʹ
1
33 000
holB
χ
1
15 000
holC
ψ
1
12 000
holD
β
4
37 000
dnaN
Funzione
Nucleo enzimatico
Attività di polimerizzazione 3′ → 5′
Esonucleasi proofreading
Dimerizzazione del nucleo enzimatico
Legame stabile allo stampo
Assemblaggio del clamp-loader intorno al DNA
Sliding clamp richiesto per la processività della sintesi
(segue)
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 5
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 6
ISBN 978-88-08-18686-7
2. il clamp loader (caricatore del morsetto), costituito dal complesso γ, composto da cinque subunità (γ, δ, δ’, χ e ψ), media
l’assemblaggio del morsetto scorrevole intorno al DNA. Questa
struttura, infatti, è responsabile dell’apertura/chiusura dell’anello β, processo che richiede ATP, e del distacco dell’enzima
dal DNA. In questo modo il complesso della DNA polimerasi responsabile della sintesi del lagging strand (vedi oltre) può, una
volta terminata la sintesi di un frammento, staccarsi dal DNA e
riposizionarsi su un nuovo primer per la sintesi del frammento
successivo.
3. la subunità τ. È una proteina (codificata dal gene dnaX) associata a ciascun complesso enzimatico di Pol III, capace di
dimerizzare. La dimerizzazione delle subunità τ permette di
mantenere legati tra loro i due nuclei enzimatici associati ai
due diversi filamenti di DNA.
Figura 9.5 RAPPRESENTAZIONE SCHEMATICA DELLA DNA POLIMERASI III.
Un nucleotide
viene aggiunto
all’estremità 3
Direzione della
polimerizzazione
5ʹ
Subunità β che
forma il morsetto
Nucleo replicativo
della DNA Pol III
Nuovo filamento
di DNA
5ʹ
3ʹ
3ʹ
Filamento
stampo di DNA
cazione. Questa idea, oltre a suggerire l’esistenza sulla molecola di DNA di un sito specifico a partire dal quale la doppia
elica viene replicata, implica che altre proteine, oltre alla DNA
polimerasi, siano necessarie per iniziare l’intero processo.
Questo modello ha poi stimolato la ricerca e l’identificazione
delle sequenze d’origine della replicazione (ori), delle proteine
iniziatrici della replicazione dei cromosomi batterici e di altri
repliconi della cellula (plasmidi e batteriofagi). L’identificazione di sequenze di DNA a replicazione autonoma (autonomously replicating sequences, ARS) in Saccharomyces cerevisiae
è stata la prima e valida estensione dello stesso modello agli
eucarioti. Negli organismi superiori, diversi siti per l’inizio
della replicazione sono stati caratterizzati biochimicamente in
vari organismi modello (in particolare S. cerevisiae, S. pombe,
Drosophila, uomo, cavia), ma la situazione è molto complessa.
In alcuni sistemi, infatti, qualsiasi sequenza di un cromosoma
può promuovere la replicazione, mentre in altri sono coinvolte
sequenze specifiche. Tuttavia, nonostante le differenze strutturali delle origini di replicazione eucariote, le proteine che
si assemblano su queste origini durante il ciclo cellulare sono
conservate nelle varie specie.
Figura 9.6 -CLAMP E DNA POL III.
Negli eucarioti la replicazione del DNA avviene esclusivamente in una fase specifica del ciclo cellulare (fase S o di sintesi),
mentre nei batteri può estendersi anche per tutto il tempo di
una divisione cellulare. Tuttavia nei procarioti l’inizio della replicazione del cromosoma è sottoposto a un rigido controllo
che fa sì che il processo inizi solo in un momento definito del
ciclo cellulare (► cap. 15).
Nonostante alcune somiglianze, il meccanismo d’inizio della replicazione varia da un replicone all’altro sia all’interno
dei procarioti sia negli eucarioti, e dipende sia dalla struttura
dell’origine di questi, sia dalla natura della proteina iniziatrice e di vari fattori che intervengono nel processo di inizio. In
questo capitolo esamineremo il caso di Escherichia coli come
esempio generale, che a grandi linee vale per molte specie batteriche.
9.2.1 L’origine di replicazione di E. coli
L’origine di replicazione del cromosoma di E. coli, detta
oriC, è costituita da una regione di 250 pb in cui sono presenti diverse sequenze di legame per la proteina iniziatrice DnaA (dette DnaA box) e sequenze ripetute ricche in
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 6
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 7
ISBN 978-88-08-18686-7
Pol I, l’enzima “eureka” e le altre polimerasi
Nel 1957 Arthur Kornberg e collaboratori scoprirono un’attività enzimatica
presente negli estratti cellulari di E. coli in grado di catalizzare l’incorporazione di desossiribonucleotidi nella molecola di DNA. La purificazione parziale dell’estratto condusse alla dimostrazione che un’attività enzimatica
poteva polimerizzare i quattro desossiribonucleotidi trifosfati (dATP, dGTP,
dCTP, dTTP) in presenza di DNA e magnesio. A questo enzima fu dato il
nome di DNA polimerasi.
La scoperta era importante non solo per il contributo che fornirà alla biologia molecolare, ma anche per il clima culturale dell’epoca. Molti scienziati
erano convinti, infatti, che la sintesi del DNA fosse così complessa da non
poter essere riprodotta in vitro. Si era, tuttavia, lontani dal capire le modalità
di funzionamento dell’enzima. L’aggiunta del DNA nella reazione, ad esempio, era dettata dall’idea che esso potesse funzionare da innesco (e non
da stampo) per essere poi allungato nello stesso modo in cui i carboidrati
allungano catene di glicogeno. Un altro motivo per aggiungere DNA nella
miscela di reazione era che il DNA potesse deviare l’attività degradativa
delle nucleasi presenti. Un anno dopo, tuttavia, il gruppo di Kornberg giunse
a comprendere che durante la reazione il DNA fungeva da stampo per la
sintesi delle nuove molecole. Il DNA sintetizzato, infatti, era caratterizzato
dall’equivalenza tra il numero delle purine e quello delle pirimidine e la sua
composizione in basi era influenzata dalla natura del DNA fornito. È importante segnalare che il classico modello di replicazione semi-conservativa
del DNA di Meselson e Stahl sarà pubblicato solo pochi mesi dopo.
Circa una decina di anni saranno necessari per ottenere l’enzima purificato e studiarne la struttura e le proprietà. Durante tutto questo periodo,
tuttavia, non mancavano i punti oscuri. John Cairns (direttore del Cold
Spring Harbor Laboratory), ad esempio, nutriva qualche perplessità sul
fatto che l’enzima purificato da Kornberg fosse implicato nella replicazione
del DNA in vivo. L’enzima, infatti, lavora in modo molto lento, con una velocità massima d’incorporazione dei dNTP di solo 20 nt s–1, e avrebbe quindi
impiegato circa 7600 min (oltre 5 giorni) per replicare l’intero cromosoma
di E. coli, che in condizioni ottimali si riproduce in 20 minuti.
Per chiarire la natura dell’attività enzimatica scoperta da Kornberg, Cairns
decise di cercare mutanti di E. coli difettivi in tale attività e di studiarne la
replicazione. Una sua collaboratrice, Paula De Lucia, identificò nel 1969
un clone (su oltre 3400 colonie i cui estratti cellulari erano stati saggiati individualmente) privo di attività polimerasica, ma che mostrava una
normale crescita. Questo primo mutante, chiamato polA1 (polA per polimerasi A, ma anche per Paula), è un mutante non senso che risulta maggiormente sensibile ai mutageni e agli effetti dei raggi UV e X rispetto al
parentale, indicando che le cellule erano difettive nella riparazione. L’analisi del comportamento del mutante PolA1 conduceva alla conclusione
che questo enzima non fosse quello coinvolto nella normale replicazione.
Gli estratti cellulari di questo mutante, inoltre, conservavano una debole
attività polimerasica a carico, come si scoprirà dopo, dell’attività congiunta
di altre polimerasi. Altri motivi che spinsero verso la ricerca di nuove polimerasi furono che la Pol I è eccessivamente abbondante (circa 4000 molecole) per un batterio con solo due forche replicative e che l’enzima non
è processivo e si dissocia dopo aver incorporato dai 2 ai 300 nucleotidi.
Ma qual è il ruolo della polimerasi I e qual è la polimerasi replicativa? La
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 7
sensibilità del mutante polA1 agli UV condurrà a scoprire che l’enzima,
oltre ad essere coinvolto nei meccanismi di escissione e riparazione di
danni al DNA, è essenziale anche nella replicazione semiconservativa. Il
suo ruolo principale è riempire i vuoti lasciati dalla rimozione degli RNA primer mediante l’aggiunta sequenziale di deossiribonucleotidi all’estremità
5′. L’enzima svolge inoltre un’importante attività di correzione degli errori
di incorporazione di basi non complementari al DNA stampo (proofreading).
Come si giustifica la vitalità dei mutanti polA1? Questa trova spiegazione
nel fatto che poche molecole funzionali di Pol I prodotte “per errore” dal
mutante polA1 sono sufficienti a garantire la funzionalità del sistema di
replicazione. Questo vale anche per altri enzimi della replicazione, come
ad esempio la ligasi.
L’enzima Pol I è costituito da una singola subunità codificata dal gene
polA. Ha attività di polimerizzazione in direzione 5′ → 3′ e di esonucleasi
in direzione sia 3′ → 5′ (detta di proofreading) sia 5′ → 3′. Hans Klenow
osservò che la proteolisi con tripsina della Pol I genera due frammenti: il più piccolo (34 kDa), corrispondente all’estremità N-terminale, che
possiede attività esonucleasica in direzione 5′ → 3′; il frammento più
grande (76 kDa, detto frammento di Klenow, molto utilizzato nei laboratori
di biologia molecolare per replicare DNA in vitro), che mantiene l’attività
polimerasica e quella di proofreading. Le altre polimerasi, Pol II, Pol III,
Pol IV e Pol V, verranno scoperte come attività presenti negli estratti del
mutante Pol I e le loro proprietà sono qui riassunte.
• Pol II, codificata dal gene polB, è una proteina di 89 kDa che si accumula durante la fase stazionaria. I mutanti di questo enzima replicano normalmente il proprio materiale genetico. Questo enzima svolge, infatti, un
ruolo nella riparazione dei danni del DNA in batteri in questo stato fisiologico. Ha un tasso di errore molto basso, ma è molto lento (40 nt s–1).
• P ol III, come abbiamo visto in questo capitolo, è l’enzima replicativo.
• P ol IV è attiva in fase stazionaria dove compete con Pol II ed è coinvolta
nella riparazione di danni del DNA. L’enzima è poco fedele e produce
errori di copiatura (replicazione error prone ► cap. 11). Si valuta che
l’attività della Pol IV sia responsabile di circa il 50% delle mutazioni
adattative durante la fase stazionaria.
Pol V è il prodotto dei geni umuC e umuD, la cui espressione è indotta
dai raggi UV. L’enzima interviene nei processi di riparazione di danni al
DNA e ha la caratteristica d’incorporare nucleotidi senza richiedere l’appaiamento specifico delle basi con il filamento stampo. Questo processo,
pertanto, permette alla replicazione di procedere nonostante la presenza
di basi danneggiate che non possono essere riconosciute dal normale
apparato replicativo. Il costo di questo sistema, che permette alla cellula
la continuazione della replicazione, è l’introduzione di errori (► cap. 11).
In conclusione, la Pol I non è l’enzima replicativo, ma merita di essere definito enzima eureka, non solo per la sua importanza nel metabolismo del
DNA, ma perché a partire da questa scoperta Kornberg e i suoi collaboratori hanno fondato e sviluppato l’enzimologia della replicazione. Ad Arthur
Kornberg, uno dei più grandi biochimici del XX secolo (come affermato dal
suo collaboratore Robert Lehman), è stato conferito il premio Nobel per la
medicina nel 1959.
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 8
ISBN 978-88-08-18686-7
ALTRE PROTEINE NECESSARIE PER LA REPLICAZIONE
Le DNA elicasi
Le DNA elicasi separano i due filamenti di DNA mediante la rottura dei
legami idrogeno tra le basi. Queste proteine si legano a una singola elica
di DNA (in una regione già “aperta”) e, procedendo in una direzione definita, scalzano l’altra elica in modo continuo (processivo) consumando una/
due molecole di ATP per ogni coppia di basi separata (fig. 9.7).
In E. coli sono note due elicasi che agiscono a livello della forca replicativa del DNA. La prima, DnaB, è indispensabile per la replicazione del
batterio. Forma un anello esamerico attorno all’elica stampo per la sintesi
discontinua e, procedendo su questa in direzione 5′ → 3′, promuove la
separazione progressiva dell’elica complementare. DnaB, inoltre, interagisce con l’enzima di innesco (priming) DnaG formando un complesso
chiamato primosoma. La seconda elicasi, Rep, non è indispensabile;
si lega al filamento di DNA opposto (lo stampo per la sintesi continua) e
promuove, procedendo in direzione 3′ → 5′, la separazione progressiva
dell’altra elica.
La DNA primasi
Come ricordato sopra, nessuna delle DNA polimerasi note è capace di
catalizzare la formazione di un legame fosfodiestere tra due desossiribonucleosidi trifosfati producendo un di-desossiribonucleotide, mentre
può formarlo con un gruppo ossidrilico fornito dall’estremità 3′–OH di un
filamento di DNA o di RNA, o (come succede per iniziare la replicazione
di alcuni genomi lineari) da un residuo aminoacidico di una proteina (fig.
9.8). In altri termini, le DNA polimerasi non sono capaci di iniziare de novo
la sintesi di una nuova elica di DNA a partire dai precursori trifosfati e
necessitano di un “innesco” (primer) per iniziare la polimerizzazione. Comunemente l’innesco viene prodotto da una RNA polimerasi che, al contrario, è capace di formare un legame fosfodiestere tra due ribonucleosidi
trifosfati. L’RNA polimerasi specializzata nel produrre i primer necessari
per innescare la sintesi del DNA è detta DNA primasi (in E. coli codificata da dnaG), che trascrive brevi sequenze ribonucleotidiche (25-40
residui) sull’elica di DNA stampo. La DNA polimerasi continuerà poi la
5ʹ
3ʹ
sintesi aggiungendo desossiribonucleotidi al 3′–OH del primer (fig. 9.2).
Per ottenere una molecola di DNA completa l’RNA primer dovrà poi essere
rimosso e sostituito mediante sintesi di un frammento di DNA. Il problema
che la sostituzione del primer pone alla replicazione delle estremità dei
genomi lineari, e come questo sia stato risolto in vari modi, è discusso
nel ► capitolo 8.
Le esonucleasi
Nella cellula di E. coli ci sono numerosi tipi di esonucleasi, che degradano il DNA a partire da un’estremità (desossiribo-esonucleasi). La DNA
Pol III usa il primer (isseco) per iniziare la sintesi del DNA nella direzione
3′ → 5′. Tra queste l’attività esonucleasica 3′ → 5′ della Pol III è particolarmente importante. Essa è a carico della proteina DnaQ, codificata dal
gene dnaQ (mutD). Questa costituisce la subunità e della polimerasi III e
svolge un ruolo importante nella correzione degli errori di incorporazione
(“correzione di bozze” o proofreading). Mutanti per questa attività mostrano una più elevata frequenza di mutazione spontanea e sono difettivi
nel sistema di riparazione del DNA, mentre l’iperespressione del gene
dnaQ abbassa di circa 100 volte la frequenza di mutazioni spontanee e di
circa 10 volte la frequenza di quelle indotte da UV.
Le DNA ligasi
Le DNA ligasi formano un legame fosfodiestere tra l’estremità 3′–OH di
un nucleotide e quella 5′-fosfato del nucleotide adiacente. Mentre le ligasi
eucariote e quelle di alcuni fagi che codificano per questo enzima (ad
esempio, T4 e T7) usano ATP come fonte di energia, le ligasi batteriche,
curiosamente, utilizzano l’energia di idrolisi del legame fosfoanidridico del
NAD generando AMP e nicotinamide, un uso insolito di questo trasportatore di elettroni.
Proteine che legano DNA a singola elica
Le proteine che legano DNA a singola elica (proteine SSB, Single-stranded DNA binding proteins), codificate in E. coli dal gene ssb, stabilizzano il
DNA
elicasi
Topoisomerasi
(anello girevole)
Proteina che lega
DNA a singola elica (SSB)
Figura 9.7 STRUTTURA DELLA FORCA REPLICATIVA. La copiatura del filamento
stampo del DNA richiede la separazione dei due filamenti complementari. Il
legame delle proteine SSB al ssDNA impedisce transitoriamente ai filamenti
complementari del DNA di appaiarsi nuovamente. Il DNA legato alle SSB
è inoltre protetto dall’attacco delle nucleasi. La DNA elicasi funziona come
un cuneo che s’inserisce a livello della forca replicativa dove rompe i legami
idrogeno tra le basi dei complementari. L’avanzamento della forcina è facilitato dall’azione della topoisomerasi che srotola progressivamente il DNA e
permette di procedere nella sintesi del DNA.
(segue)
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 8
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 9
ISBN 978-88-08-18686-7
DNA a singolo filamento prodotto dall’attività delle elicasi
impedendone la riassociazione con l’elica complementare e lo proteggono dall’attacco delle nucleasi. SSB è una
piccola proteina (18 kDa) che, in forma tetramerica, lega
selettivamente e cooperativamente il DNA a singolo filamento senza alcuna specificità di sequenza. Per questa
sua proprietà le proteine SSB intervengono in vari processi come la ricombinazione e la riparazione del DNA.
Le topoisomerasi
Un altro problema associato alla separazione dei filamenti
della molecola di DNA durante la sintesi è rappresentato dallo stato topologico del cromosoma (► cap. 8). Il
superavvolgimento negativo della molecola di DNA deve
essere convertito in una struttura maggiormente rilassata
che consenta l’avanzamento della forca replicativa. Questa attività è svolta dalla topoisomerasi I e dalle elicasi.
Successivamente il DNA neosintetizzato deve ritrovare il
suo stato di avvolgimento negativo, processo assicurato
dalla DNA girasi (► cap. 8).
Figura 9.8 INNESCO DELLA SINTESI DI UN’ELICA DI DNA DA PARTE DELLA DNA PRIMASI.
AT. In particolare vi si identificano (partendo da destra in
fig. 9.9) quattro copie della DnaA box (sequenza consenso
5′–TTATC/ACAC/AA–3′) orientate in modo inverso l’una
rispetto all’altra e designate come R1, R2, R4 (con alta affinità
di legame per la proteina DnaA) e R3 (con debole affinità).
Sono anche presenti sequenze per il legame di IHF e FIS,
due proteine istone-simili (► cap. 8). A sinistra delle DnaA
box c’è una regione ricca in AT denominata elemento DUE
(per DNA-unwinding element) e composta da tre ripetizioni in
tandem di una sequenza di 13 pb ricca in AT. L’appaiamento
meno stabile delle sequenze ricche in AT favorisce la separazione dei due filamenti del DNA e l’apertura di questa regione. Il legame della proteina DnaA a quest’ultima conduce
a un’ulteriore separazione dei due filamenti di DNA a livello
di oriC. L’origine contiene, inoltre, 11 copie della sequenza
palindromica 5′–GATC–3′ (otto di queste si trovano in posizioni molto conservate in varie specie). Questi siti sono riconosciuti dalla desossiadenosina metiltransferasi (Dam), un
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 9
enzima che metila i residui di adenina in posizione N6 (mA)
nel DNA. Immediatamente dopo la replicazione, solo le sequenze sull’elica stampo conterranno mA, mentre le A della
nuova elica saranno metilate successivamente. Lo stato transitorio di emimetilazione di questi siti al momento della replicazione della regione oriC gioca un ruolo importante nel
controllo della replicazione (► Regolazione dell’inizio della replicazione nei batteri e ► cap. 15).
La proteina DnaA
Il primo mutante letale condizionale (mutante termosensibile, ts) isolato in E. coli alla fine degli anni ’60 era difettivo nella replicazione del DNA e identificò il gene dnaA. In seguito
furono isolati numerosi altri mutanti termosensibili incapaci
di replicare il DNA, riconosciuti come tali per il fatto che,
trasferiti alla temperatura non permissiva (42-44 °C), smettevano di incorporare precursori del DNA (timidina triziata)
in macromolecole. Questi mutanti potevano essere distinti in
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 10
ISBN 978-88-08-18686-7
Siti di legame per DnaA
(DnaA box, 9 pb)
Regione ricca in AT
(ripetizioni in tandem di 13-meri)
5ʹ — G A T C T N T T N T T T T — 3ʹ
3ʹ — C TA G A N A A N A A A A — 5ʹ
Sequenza consenso del 13-meri
HU e IHF
Primasi
Proteine di DnaA +
ATP
SSB
Proteina DnaC
ADP
ATP
Primasi DnaG
ATP
Elicasi
DnaB
Complesso
DnaB-DnaC
DnaC
SSB
Figura 9.9 INIZIO DELLA REPLICAZIONE DI E. COLI A LIVELLO DI oriC. La parte superiore della figura riporta gli elementi strutturali della regione oriC, quella inferiore mostra il ruolo di questi nell’inzio della replicazione. (1) La proteina
DnaA si lega alle sequenze DnaA box in modo cooperativo. L’energia derivante dall’idrolisi dell’ATP (dalla sua attività ATPasica) è utilizzata per srotolare
la doppia elica del DNA a livello delle sequenze ricche in AT. DnaA si associa
a una delle DnaA box. Questo evento promuove il legame cooperativo delle
altre molecole di DnaA complessate all’ATP sulle altre DnaA box. L’energia
derivante dall’idrolisi dell’ATP è utilizzata dalla proteina DnaA per denaturare
le tre regioni di 13 pb ricche in AT e separare i due filamenti di DNA. (2) La
permanenza della struttura ad occhiello con i filamenti separati è facilitata dai
legami di questo con le proteine SSB e permette all’elicasi DnaB di rompere i
legami H e l’avanzamento della forca replicativa. A questo stadio si forma un
aggregato di circa 30 molecole di DnaA legato alle DnaA box ripetute di oriC
e questa regione del DNA si arrotola sulla superficie del complesso, reazione
facilitata dalle proteine HU e IHF. Il complesso DnaA-DNA, in presenza di
ATP, continua a separare progressivamente i due filamenti di DNA fino a raggiungere le tre sequenze ripetute ricche in AT. Si forma in questo modo una
regione di ssDNA, un complesso aperto di 45 pb. Dopo questi eventi, DnaA
conduce due esameri dell’elicasi DnaB associata a DnaC a legare ognuno su
ogni filamento del complesso aperto formando una struttura nucleoproteica
specializzata detta complesso di pre-innesco (prepriming). La proteina DnaC
viene quindi rilasciata dal complesso. L’attività dell’elicasi DnaB in direzione
5′ → 3′ accoppia l’idrolisi dell’ATP con lo srotolamento del DNA a livello della
forca replicativa. (3) La primasi DnaG innesca la sintesi dell’RNA primer. Dopo
un’ulteriore separazione delle eliche del DNA, i due filamenti sono ricoperti da
tetrameri delle proteine SSB, il cui ruolo è proteggere il ssDNA e contrastare la
tendenza all’appaiamento delle basi. La forca replicativa che si è così formata
permette il reclutamento della primasi DnaG e di Pol III, la sintesi del primo
innesco a RNA e l’avvio della replicazione.
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 10
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 11
ISBN 978-88-08-18686-7
REGOLAZIONE DELL’INIZIO DELLA REPLICAZIONE NEI BATTERI
1. un dominio N-terminale coinvolto nella oligomerizzazione
DnaA-DnaA e interazioni DnaA-DnaB;
2. una regione flessibile che può essere deleta senza compromettere la funzionalità della proteina, ma che potrebbe servire in particolari condizioni di crescita;
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 11
3. una regione per il legame all’ATP, implicata nell’oligomerizzazione della proteina;
4. un dominio C-terminale che si lega al DNA.
9.2.2 L’inizio della replicazione a oriC
Le molteplici funzioni di DnaA sono tutte dipendenti dalla
capacità di questa proteina di riconoscere e legare le DnaA
box non solo presenti in oriC ma anche associate ad alcuni
promotori, tra cui lo stesso promotore di dnaA. Il riconoscimento delle DnaA box in oriC da parte di questa proteina
conduce al suo assemblaggio in un complesso nucleoproteico, un pentamero di DnaA associato al DNA, chiamato complesso d’inizio. La distorsione della molecola di DNA che
si crea nel complesso d’inizio è un prerequisito per il successivo assemblaggio del primosoma, un complesso multiproteico necessario per la sintesi degli inneschi a RNA (fig. 9.9 e
► Il primosoma nei batteri). Legandosi alle DnaA box presenti nelle regioni del promotore, DnaA agisce anche come
Ceppo selvatico
30 °C
incorporata
due classi: mutanti a blocco lento (tra cui dnaAts) e mutanti a blocco rapido (fig. 9.10). Nei primi, dopo trasferimento a
temperatura non permissiva, la sintesi del DNA continuava
per la durata di un ciclo di replicazione, mentre nei secondi
si arrestava quasi istantaneamente. Questo diverso fenotipo
ha permesso di ipotizzare, in accordo con il modello del replicone, che nei mutanti a blocco lento fossero mutati geni
implicati nel controllo dell’inizio del processo di replicazione,
mentre nei mutanti a blocco rapido fossero alterate funzioni
necessarie per la polimerizzazione. Infatti, dopo la transizione dalla temperatura permissiva a quella non permissiva di un
mutante nel processo di inizio (come dnaAts), nelle cellule in
cui la replicazione è già iniziata essa continua fino a completare la duplicazione del cromosoma. Questo perché la proteina
iniziatrice non è richiesta per continuare nella sintesi di DNA,
che si arresterà solo una volta terminata la replicazione del
cromosoma, non potendo ripartire un nuovo ciclo. Al contrario, nei mutanti in geni implicati nella polimerizzazione
l’arresto della replicazione in condizioni non permissive sarà
immediato.
La proteina DnaA fa parte di una famiglia di proteine ubiquitarie con attività di ATPasi, denominate AAA+ (ATPases Associated with various cellular Activities) che hanno alcuni motivi
strutturali comuni. Molte di queste sono coinvolte nell’inizio della replicazione e in altre funzioni del metabolismo del
DNA. Proteine che contengono il motivo strutturale AAA+
spesso interagiscono per formare strutture circolari multimeriche. Per quanto riguarda DnaA, la proteina è costituita da
quattro domini funzionali:
cystis, al contrario, non ci sono regioni con parecchie DnaA box; inoltre in
questi cianobatteri la delezione del gene codificante per DnaA non è letale.
La replicazione di questi organismi non è nota, ma il fatto che il gene dnaA
non sia essenziale indica che l’inizio della sua replicazione deve procedere
mediante una via diversa.
La proteina iniziatrice DnaA è stata trovata in tutti i batteri fino ad ora studiati e il confronto di oltre 100 differenti DnaA ha rivelato simili l’organizzazione in 4 domini e l’attività. Oltre alla forte conservazione della proteina
DnaA nei procarioti, la sua controparte è stata ritrovata negli eucarioti
come subunità che riconosce il complesso d’inizio della replicazione negli
eucarioti. Quanto descritto suggerisce che l’elemento critico della regolazione della replicazione per la maggior parte delle specie batteriche è la
proteina DnaA.
3H-timidina
L’inizio della replicazione è un passaggio critico a livello del quale viene
attuato un rigido controllo che integra il ciclo di replicazione del DNA con il
ciclo cellulare (► par. 3.8). I due elementi principali del processo d’inizio
della replicazione sono la proteina DnaA e le sequenze bersaglio ori. In
questa scheda analizzeremo se il modello descritto per E. coli è estendibile ad altre specie batteriche e toccheremo alcuni problemi connessi al
controllo della replicazione in sistemi batterici diversi da quelli più studiati.
Molti batteri appartenenti a vari generi hanno origini di replicazione che
variano in dimensioni, numero delle DnaA box ed estensione delle regioni
ricche in AT. In tutti i casi analizzati (ad eccezione di Synechocystis, un
genere dei Cianobatteri) tutte le origini sono caratterizzate dal possedere
parecchie DnaA box per il legame della proteina iniziatrice e una regione
ricca in AT che facilita la formazione del complesso aperto. In Synecho-
42 °C
Blocco lento: mutanti
in geni necessari per
“iniziare” la replicazione
Blocco rapido: mutanti
in geni necessari per
“far procedere”
la replicazione
Tempo
Figura 9.10 IDENTIFICAZIONE DEI GENI NECESSARI PER LA REPLICAZIONE.
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 12
ISBN 978-88-08-18686-7
IL PRIMOSOMA NEI BATTERI
Il primosoma è il complesso proteico responsabile dello srotolamento processivo dei due filamenti del DNA e della sintesi del primo innesco durante
la replicazione. I componenti essenziali che lo costituiscono sono l’elicasi, la primasi e altre proteine ausiliarie. Alcuni costituenti del primosoma
svolgono, inoltre, un importante ruolo, come vedremo, nel fare ripartire la
replicazione quando questa è bloccata da eventi che ne ostacolano la normale progressione (danni della molecola di DNA e altri eventi). Nei batteri
i primosomi differiscono per la natura del sito del loro assemblaggio e le
proteine che lo riconoscono e per alcune proteine accessorie (► tab. 9.4).
Il termine di primosome è stato coniato da Ken-ichi Arai e A. Kornberg nel
1981, per descrivere il complesso multiproteico necessario per iniziare
la replicazione di φX174, un fago a DNA circolare a singolo filamento. Il
ssDNA circolare di φX174 (forma SS) è un substrato ideale per studiare
il processo di inizio della replicazione e identificare le proteine necessarie
per convertirlo nell’intermedio a doppio filamento (detta forma replicativa,
RF). Questo processo richiede la presenza nel genoma virale di una specifica sequenza, chiamata pas (primosome assembly site) dove si assemblano le proteine del primosoma di φX174, codificate dall’ospite batterico,
che sono: le SSB che ricoprono il DNAss, un complesso di tre proteine
(PriA, PriB, PriC, DnaT, DnaB e DnaC) richieste per assemblare queste
proteine in un complesso detto pre-primosoma che viene completato con
l’associazione della primasi DnaG che sintetizza l’RNA primer PriA e funge
da impalcatura alle altre proteine che si assemblano in modo ordinato
a livello di una piccola regione a forcina del ssDNA. Le proteine PriA,
PriB e PriC di E. coli, nonostante il loro coinvolgimento nella replicazione
di φX174, non sono necessarie per la normale replicazione del batterio anche se svolgono un ruolo importante. Mutanti del gene priA, infatti,
hanno una ridotta vitalità e mostrano un’elevata frequenza di arresto della
replicazione con forche che non giungono a termine. Inoltre le cellule di
questi batteri presentano un fenotipo filamentoso (dovuto a inibizione della
divisione cellulare), maggiore sensibilità agli UV ed espressione costitutiva
fattore trascrizionale che può condurre alla repressione di
alcuni geni, all’attivazione di altri e all’autoregolazione della
propria espressione.
9.2.3 Meccanismi di controllo dell’inizio della
replicazione
In E. coli sono noti almeno tre meccanismi che assicurano il
controllo di oriC: il sequestro transitorio di oriC, la disponibilità della proteina DnaA e l’inattivazione di DnaA. Questi
meccanismi sono generalmente presenti in molti altri batteri.
Riassumeremo pertanto la situazione in E. coli e segnaleremo
le differenze presenti in alcune altre specie batteriche.
Il sequestro dell’origine di replicazione a livello della membrana è operato dalla proteina SeqA. Questa proteina lega preferenzialmente sequenze GATC emimetilate, una condizione
presente nel DNA neosintetizzato, nella regione oriC. Subito
dopo la replicazione si genera uno stato di emimetilazione in
del “sistema SOS” (► cap. 11), difetti nel sistema di ricombinazione omologa e nella riparazione del DNA. Quanto illustrato solleva l’interrogativo
sul ruolo delle proteine Pri per E. coli. PriA, che è quella più studiata e la
più conservata evolutivamente, è una proteina multifunzionale con attività
di ATPasi, elicasi e traslocasi. PriB lega il ssDNA sul quale si assembla il
complesso PriA-PriB-DNAss. Vari studi hanno mostrato che PriA è fondamentale in E. coli per la ripresa della replicazione in cellule in cui questa
funzione è transitoriamente bloccata. In tutti gli organismi esistono sistemi
di controllo della corretta esecuzione del ciclo cellulare (detti checkpoint).
In presenza di anomalie, come ad esempio l’impedimento della progressione della forca replicativa, questi sistemi possono bloccare la progressione del ciclo inducendo processi che rimuovono l’anomalia e permettono la ripresa del ciclo. La frequenza con cui la replicazione si arresta in
condizioni naturali non è nota, ma sappiamo che molteplici fattori possono
ostacolare la progressione della forca replicativa: sequenze danneggiate,
sequenze in fase di trascrizione, regioni in corso di riparazione ecc. Nei
batteri la ripresa della replicazione alle forche replicative bloccate avviene
attraverso varie vie.
Tabella 9.4 COMPOSIZIONE DEL PRIMOSOMA IN ESCHERICHIA COLI.
Proteine
Gene
Organizzazione
Peso molecolare (kDa)
PriA
priA
Monomero
76
PriB
priB
Dimero
11,5
PriC
priC
Monomero
23
DnaT
dnaT
Trimero
22
DnaB
dnaB
Esamero
50
DnaC
dnaC
Monomero
29
DnaG
dnaG
Monomero
60
cui il filamento stampo è metilato mentre quello copiato non
lo è ancora (► cap. 15, fig. 9.11).
Un altro fattore di controllo è la disponibilità delle molecole di
DnaA a livello di oriC. Questa è regolata dal numero di copie di
datA (DnaA titration A), un locus genomico situato a circa 470
kb da oriC cui possono legarsi circa 300 molecole di DnaA in
competizione con oriC. Quando la forca replicativa attraversa
questo sito, il numero di copie della sequenza datA aumenta
sequestrando nuove molecole di DnaA che in tal modo saranno meno disponibili per oriC. Data la distanza di datA da oriC,
questa titolazione di DnaA avviene parecchi minuti dopo l’inizio della replicazione e contribuisce a dilazionare la formazione di nuovi complessi di inizio.
Infine, l’idrolisi dell’ATP legata converte la forma attiva della
proteina iniziatrice, DnaA-ATP, nella forma inattiva DnaAADP. Questo processo è stimolato dallo sliding clamp (morsetto) della Pol III.
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 12
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 13
ISBN 978-88-08-18686-7
Figura 9.11 IL CONTROLLO DELL’INIZIO DELLA REPLICAZIONE DEL DNA. La replicazione del cromosoma
è controllata durante il ciclo cellulare attraverso diverse vie di regolazione (che differiscono nelle varie
specie) e che agiscono a livello della proteina iniziatrice e sull’origine di replicazione. (a) In E. coli e B.
subtilis la proteina iniziatrice DnaA si lega ad oriC
e avvia la replicazione. Questa proteina può legare
sia ADP sia ATP ma è solo questa seconda forma
quella attiva. Vari processi nella cellula controllano
la concentrazione e il legame di DnaA-ATP ad oriC
tra questi, l’attività della proteina Hda che esercita
un controllo negativo. Per prevenire il reinizio della replicazione il complesso ADP-Hda e il morsetto
del replisoma promuovono l’idrolisi del complesso
DnaA-ATP in DnaA-ADP inattivo (la natura dell’interazione Hda-DnaA non è nota). (b) In B. subtilis, il gene
yabA codifica per un regolatore negativo dell’inizio
della replicazione. Proteine omologhe a YabA sono
state trovate in molte specie di batteri Gram-positivi. YabA agisce sulla processività del β-clamp della
DNA polimerasi e modula l’attività di DnaA (il meccanismo non è ancora chiarito). (c) In C. crescentus
(► cap. 15) il regolatore del ciclo cellulare CtrA, si
lega all’origine di replicazione Cori e inibisce l’inizio
della replicazione. La degradazione di CtrA al momento giusto del ciclo cellulare regola la replicazione.
a) Escherichia coli
SeqA
datA
oriC
PdnaA
dnaA
dnaN
DARS1 e DARS2
DiaA
DnaA-ADP
DnaA-ATP
Replisoma
Hda
Morsetto
SpoOA
b) Bacillus subtilis
parS
oriC
Soj
PdnaA
dnaA
dnaN
DnaD
ATP
Replisoma
YabA
Morsetto
CtrA
c) Caulobacter crescentus
PdnaA
Replisoma
dnaA
Cori
ATP
HdaA
Morsetto
Esaminiamo ora la partecipazione di questi meccanismi di regolazione nelle varie specie batteriche.
Il processo di sequestro di oriC è presente, oltre che in E. coli,
in altri Enterobatteri e in molti altri generi (Agrobacterium tumefaciens, Brucella abortus, Caulobacter crescentus, Rhizobium
meliloti e Rickettsia prowazekii).
La titolazione della proteina DnaA, mediante un cluster di siti
ad alta affinità per DnaA localizzati fuori della regione oriC, è
coinvolta nella regolazione dell’inizio della replicazione di altri
batteri, come S. coelicolor. È stato osservato, ad esempio, che
delezioni di questo cluster causano cicli di replicazione più frequenti.
L’inattivazione del complesso DnaA-ATP mediante idrolisi
dell’ATP è presente in tutti i batteri che hanno il gene dnaA
(Bacillus subtilis, Helicobacter pylori, Mycobacterium tuberculosis, Streptomyces coelicolor, Thermus thermophilus e Thermotoga
maritima). In genere tutti i geni dnaA sequenziati codificano
per un motivo AAA+ responsabile del legame e dell’idrolisi
dell’ATP. Questo stesso motivo è anche presente in proteine
che iniziano la replicazione dei cromosomi eucarioti e negli archei. Il legame e l’idrolisi dell’ATP agisce come un commutaMateriale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 13
tore universale che regola l’inizio della replicazione di tutti gli
organismi viventi. Tuttavia poco è noto sul meccanismo o sui
meccanismi di inattivazione dell’iniziatore.
9.2.4 L’inizio della replicazione di batteri con più
cromosomi
Il controllo della replicazione dei batteri con più cromosomi
(► cap. 15) è molto meno conosciuto rispetto agli organismi con un singolo cromosoma. Il sistema maggiormente
studiato è quello di Vibrio cholerae. I due cromosomi di questo batterio replicano in modo sincrono nonostante le loro
diverse dimensioni, le differenze nelle loro origini e nel processo d’inizio.
Il sito oriCI di V. cholerae somiglia all’oriC di E. coli, mentre oriCII ha peculiarità comuni ad alcuni plasmidi. oriCII possiede
una ripetizione interna di 12 pb e due geni che fiancheggiano
l’origine. Uno di questi codifica per la proteina RctB che lega
specificamente oriCII e ne controlla la replicazione. DnaA e
RctB sembrano controllare indipendentemente l’inizio della
replicazione rispettivamente dei due cromosomi chrI e chrII
in quanto la sovraproduzione sperimentale di DnaA o RctB
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 14
ISBN 978-88-08-18686-7
promuove esclusivamente più inizi del cromosoma I o del cromosoma II ma non di ambedue.
In che modo l’inizio della replicazione dei due cromosomi sia
coordinata non è noto. Pur avendo scarsa somiglianza di sequenza con oriCI, oriCII contiene una singola DnaA box oltre a parecchie sequenze GATC, bersagli della metiltransferasi Dam.
Anche se DnaA non inizia la replicazione del cromosoma II, la
sua presenza, seppur in concentrazione minima, è richiesta per
l’attivazione dell’inizio della replicazione di questo cromosoma.
Una simile situazione è osservata in alcuni plasmidi (come RK2)
che, pur codificando per proprie proteine iniziatrici, richiedono
DnaA.
L’abbondanza di siti per la metilasi Dam in oriCI e oriCII di V.
cholerae e la presenza dei geni seqA e dam suggerisce che questi
fattori possono mediare il coordinamento della replicazione in
modo simile ad E. coli; tuttavia fattori addizionali non ancora noti
probabilmente sono richiesti per coordinare le attività di DnaA e
RctB.
Poco è noto sui segnali che legano il ciclo intracellulare di batteri
patogeni e simbionti con quello dell’ospite. È stato recentemente
mostrato nel caso di M. tuberculosis che l’inizio della replicazione
di questo batterio è regolato da un sistema di trasduzione del segnale (costituito da due proteine MtrA-MtrB) attivato dall’interazione ospite-patogeno. In M. tuberculosis MtrA regola la replicazione inducendo l’espressione di dnaA. La proliferazione di M.
tuberculosis in vivo dipende in parte dal rapporto MtrA fosforilata
su MtrA non fosforilata.
Un caso estremo è quello di alcuni endosimbionti obbligati degli insetti (Wigglesworthia glossinidia e varie specie del genere
Blochmannia) che hanno perso il gene dnaA. Non si possono,
quindi, escludere vie alternative per l’inizio della replicazione di
questi batteri.
9.3 Innesco della sintesi e allungamento
del DNA
L’inizio della copiatura del filamento stampo e la continuazione della sintesi del DNA sono due processi complessi che
richiedono l’intervento di molte proteine. Per comprendere
la complessità del sistema dobbiamo tenere presenti alcune
caratteristiche della DNA polimerasi (direzionalità di polimerizzazione 5′ → 3′; necessità di un primer) e del DNA (doppia
elica con filamenti antiparalleli). Per iniziare la replicazione di
una molecola di DNA occorre svolgere progressivamente le
due eliche; la DNA primasi, procedendo sull’elica stampo in
direzione 3′ → 5′, polimerizza un primer a RNA con polarità
5′ → 3′. L’estremità 3′–OH del primer funge da innesco per la
DNA polimerasi, che può sintetizzare il nuovo filamento di
DNA in direzione 5′ → 3′ complementare allo stampo su cui
si muove. Virtualmente, la sintesi di questo filamento (detto
filamento guida o leading strand) può continuare indefinitamente; di fatto, la sintesi si interrompe di tanto in tanto e
riprende con un nuovo innesco. La replicazione del filamen-
to complementare, che viene spiazzato progressivamente in
direzione 5′ → 3′ dalla forca replicativa, richiede invece un
meccanismo più complesso in quanto viene copiato a tratti in modo retrogrado man mano che è reso disponibile dal
progredire della replicazione dell’altro filamento. Infatti la
DNA primasi e poi la DNA polimerasi sintetizzano il nuovo
filamento a partire da un sito prossimale alla forca replicativa
e procedono in direzione opposta a questa. Nel frattempo un
nuovo tratto di elica stampo sarà reso disponibile dal procedere della forca replicativa e la sintesi riprenderà, 1,5-2 kb più
avanti, con un nuovo primer e un nuovo tratto di DNA (fig.
9.2). Questo filamento è replicato in modo discontinuo (generando frammenti lunghi 1,5-2 kb detti frammenti di Okazaki, da Reiji e Tsuneko Okazaki che li osservarono per primi) e
leggermente ritardato (lagging strand) rispetto all’altro. È da
tener presente che in un replisoma che avanza verso una forca
replicativa ciascun filamento funge da stampo per un diverso
complesso enzimatico Pol III (fig. 9.4).
Durante la sintesi del DNA, gli RNA primer dei filamenti leading e lagging devono essere rimossi e sostituiti con frammenti
a DNA. Una ribonucleasi speciale, la RNAsi H, è responsabile
della degradazione degli RNA presenti tra i frammenti di Okazaki. La Pol I a sua volta copia il filamento stampo e aggiunge
deossiribonucleotidi all’estremità 5′ del frammento di Okazaki, colmando il tratto a singola elica. Al termine del processo la
DNA ligasi salda covalentemente il frammento neosintetizzato al frammento di Okazaki adiacente.
Il replisoma
Il replisoma completo è localizzato sulla forcina di replicazione ed è costituito dalla primasi (DnaG), dall’elicasi (DnaB) e
dall’oloenzima DNA polimerasi III (Pol III). L’oloenzima Pol
III è formato da due corpi enzimatici (uno per ogni filamento
stampo) uniti fra loro da un dimero della subunità τ (fig. 9.5).
Esamineremo qui il ruolo e la cooperazione delle attività di
questi enzimi a livello del macchinario di replicazione.
L’attività congiunta della elicasi e della primasi
La primasi DnaG è reclutata a livello della forca di replicazione
mediante un’associazione con l’elicasi DnaB. Successivamente, un dimero della subunità β di Pol III è guidato a innescare
la replicazione in modo ATP dipendente. Il processo è catalizzato dalle subunità γ in assenza del nucleo enzimatico della
polimerasi, che successivamente si unisce al complesso. Nel
replisoma così assemblato, il complesso DnaB-DnaG interagisce con la Pol III attraverso la subunità τ che assicura anche la
comunicazione tra i due nuclei dell’oloenzima Pol III. Queste
interazioni accoppiano le attività del replisoma e assicurano la
sintesi coordinata dei due filamenti stampo (figg. 9.4 e 9.9).
L’associazione diretta della primasi con l’elicasi è alla base della coregolazione delle funzioni di questi enzimi. La primasi,
infatti, incrementa sia l’attività di ATP sintetasi sia quella di
elicasi di DnaB. Nello stesso modo, DnaB può modulare tutte
le attività di DnaG, così come la lunghezza dei primer sintetiz-
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 14
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
ISBN 978-88-08-18686-7
Materiale online - CAPITOLO 9 15
L’assemblaggio ciclico della primasi e DNA polimerasi III sul lagging
strand e sintesi dei frammenti di Okazaki
Il complesso della DNA polimerasi III si forma a livello del primer in modo
ordinato e gerarchico. Terminata la sintesi del primer, i due anelli dello sliding clamp si assemblano uno sul leading strand, l’altro sul lagging strand e questo processo richiede ATP. Ad ognuno di questi anelli
si associano, successivamente, il clamp loader e il nucleo enzimatico
della DNA polimerasi III. In questo processo, mentre il complesso rimane costantemente legato al DNA sul leading strand per catalizzare una
polimerizzazione continua, sul lagging strand esso è ripetutamente rilasciato e riassociato al DNA per sintetizzare ciascun frammento di Okazaki
successivo. La sintesi di un frammento di Okazaki è iniziata, quindi, al
termine della sintesi del primer, dopodiché la primasi si dissocia per sintetizzare un nuovo primer.
La sintesi ciclica dei primer e dei frammenti di Okazaki del lagging strand
richiede alcuni eventi che vengono qui di seguito riassunti:
2. il monomero della polimerasi del lagging strand si dissocia dal frammento di Okazaki e il β-clamp si associa a un nuovo primer. In questo
modo la polimerasi può posizionarsi con un altro sliding clamp sul primer successivo. In questo sistema quindi il macchinario di replicazione
è permanentemente posizionato a livello della forcina di replicazione,
dove può espletare la sua attività in modo efficiente.
1. il clamp loader, in presenza di ATP, subisce un cambiamento conformazionale che aprendone la struttura permette il suo distacco dal DNA
ed il suo successivo assemblaggio su un nuovo primer;
Lo stesso tipo di meccanismo permette alla subunità β della polimerasi III
di associarsi con le altre DNA polimerasi di E. coli (I, II, IV e V), con la DNA
ligasi e con MutS.
zati dalla primasi e la specificità dell’inizio. DnaG è debolmente attiva in vitro e il tasso massimo di sintesi è di 3 primer per
ora. DnaB è essenziale in vitro affinché la replicazione proceda
con il tasso di sintesi osservato in vivo. L’importanza dell’interazione tra l’elicasi e la primasi è testimoniata dal fatto che in
alcuni sistemi virali, come nel batteriofago T7, i geni codificanti per l’elicasi e la primasi sono fusi dando luogo alla sintesi di
un unico polipeptide.
Poiché in una cellula di E. coli ci sono solo 50-100 molecole
di primasi ma migliaia di frammenti di Okazaki, DnaG deve
essere riciclata da ogni primer per i diversi cicli di sintesi successive.
La progressione del replisoma lungo il DNA
L’attività di polimerizzazione in direzione 5′ → 3′ della DNA
polimerasi e l’antiparallelismo dei due filamenti da copiare
implicano che i due monomeri della DNA polimerasi “procedano” in senso opposto l’uno rispetto all’altro. Una ventina
d’anni fa il modello dominante della sintesi del DNA prevedeva che la DNA polimerasi passasse velocemente e ripetutamente da un filamento all’altro. Questo modello rendeva difficile capire come la sintesi potesse essere coordinata sui due
filamenti. Bruce Alberts e collaboratori proposero nel 1983
un modello alternativo, detto modello a trombone, che prevede
che il lagging strand formi un’ansa in modo tale da permettere la colocalizzazione e l’orientamento di una subunità della
DNA polimerasi nella stessa direzione di quella presente sul
leading strand cosicché, in modo concertato, i due complessi
possano procedere in direzione 5′ → 3′ (figg. 9.4 e 9.12). Questo modello elegante ha trovato sostegno nel fatto che le due
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 15
Il completamento della replicazione richiede infine i seguenti eventi:
1. l’escissione degli RNA primer;
2. iI riempimento degli spazi lasciati dalla rimozione dell’RNA primer
mediante l’aggiunta sequenziale di desossiribonucleotidi all’estremità
5′–OH complementari allo stampo. Per questa sintesi è coinvolta la
polimerasi I;
3. il legame tra due basi adiacenti saldato dalla DNA ligasi.
subunità della polimerasi sono, come abbiamo visto, fisicamente legate e in dati citologici sulla colocalizzazione delle
due polimerasi.
Fedeltà della replicazione durante l’allungamento
La replicazione del DNA dei batteri è molto accurata. Si stima
che gli errori nell’incorporazione delle basi durante la replicazione siano prodotti con frequenze comprese tra 10–4 e 10–6,
mentre quelli dovuti a inserzioni di coppie di basi tra 10–8 e
10–6. Questo basso tasso di errore è attribuibile a:
1. selezione termodinamica delle basi ai fini di un corretto appaiamento;
2. contributo della DNA polimerasi nella selezione corretta
delle basi da aggiungere;
3. attività esonucleasica della DNA polimerasi che, in caso
di incorporazione di una base errata, degrada in direzione
3′ → 5′ il filamento neosintetizzato rimuovendo le basi appaiate scorrettamente.
9.4 Terminazione e risoluzione
(separazione) dei nuovi cromosomi
La replicazione del cromosoma di E. coli termina a livello di
una regione di terminazione di 350 kb fiancheggiata da dieci
siti di terminazione di 23 pb (siti ter) orientati diversamente su
entrambi i lati della regione di terminazione (fig. 9.3). La proteina Tus si lega ai siti ter e arresta la progressione delle forche
replicative mediante un blocco dell’attività dell’elicasi DnaB.
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 16
ISBN 978-88-08-18686-7
a)
3
3
5
5
Filamento a replicazione immediata
3
3
5
5
3
3
3
3
Filamenti parentali
Filamento a replicazione ritardata
5 3
5 3
Figura 9.12 LA REPLICAZIONE CONTINUA E QUELLA DISCONTINUA DEL DNA. (a) Le proprietà della DNA polimerasi (direzionalità della polimerizzazione 5′ → 3′ e
necessità d’un innesco) si traducono con una replicazione che procede in modo differenziale per le due
eliche del DNA (una continua, l’altra discontinua). (b)
La differenza di polarità dei due filamenti impone alla
DNA polimerasi di funzionare in opposte direzioni per
i due filamenti. Per permettere alla DNA polimerasi di
sintetizzare i due filamenti del DNA nella stessa direzione e nel senso del movimento della forcina replicativa, il filamento lagging forma un’ansa che orienta
il tratto di DNA in sintesi nella stessa direzione del
filamento leading.
5
5
Frammenti di Okazaki
Movimento della forca replicativa
b)
3
Dimero di DNA
polimerasi
5
Filamento a replicazione immediata
3
3
3
Filamento a replicazione
ritardata
5 3
5 3
5
Filamenti parentali
5
Frammenti di Okazaki
Movimento della forca replicativa
Un simile meccanismo è presente in molti altri batteri; tuttavia
né i siti ter né le proteine coinvolte nella terminazione mostrano una conservazione filogenetica. Il controllo della terminazione è comunque un processo importante. I due cromosomi
fratelli potrebbero trovarsi, infatti, al termine della replicazione intrecciati uno con l’altro oppure fusi insieme in un dimero a causa di eventi di ricombinazione e devono quindi essere
monomerizzati in vista della ripartizione dei cromosomi nelle
due cellule figlie (► cap. 15).
9.5La replicazione degli elementi
extracromosomali e il suo controllo
I plasmidi e i genomi virali hanno una replicazione autonoma,
sono cioè repliconi indipendenti rispetto al cromosoma della
cellula che li ospita. L’apparato deputato alla loro replicazione può essere costituito interamente da proteine batteriche,
da proteine codificate dall’elemento stesso, o da una combinazione di fattori di replicazione codificati sia dal genoma
del batterio ospite sia dall’elemento extracromosomale. Anche nei plasmidi la replicazione procede nelle tre fasi: inizio,
a livello di un’origine specifica, allungamento del polimero e
terminazione. Alcuni plasmidi hanno più di un’origine di replicazione, ma in genere solo una è richiesta per la replicazione vegetativa, mentre l’altra è attiva in particolari situazioni,
come ad esempio durante il trasferimento coniugativo del
DNA (► cap. 12). Per l’apertura del DNA al sito ori nella fase
d’inizio della replicazione, molti plasmidi necessitano, oltre
alle proteine richieste per l’inizio della replicazione del batterio (DnaA, HU e IHF, nel caso di plasmidi di E. coli), anche
proteine codificate dal plasmide. Il controllo del numero delle
copie del DNA plasmidico coinvolge vari tipi di meccanismi
ed è dipendente da elementi strutturali e da proteine codificate
dal plasmide.
In questo capitolo ci limiteremo a una breve sintesi sulla replicazione di alcuni plasmidi modello, rinviando al ► capitolo 17
per quella dei batteriofagi.
9.5.1 La replicazione del fattore F
La replicazione plasmidica è stata ben studiata per il fattore F, e
in particolare per alcuni suoi derivati costruiti in vitro mediante
delezioni. Questi derivati (chiamati mini F) che contengono
solo 9 kb del DNA di F conservano una replicazione autonoma
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 16
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 17
ISBN 978-88-08-18686-7
e hanno permesso di effettuare una “fine dissezione” dei geni e
dei siti necessari per la replicazione F. Altri costrutti portatori
delle funzioni per il controllo del numero delle copie, partizione e incompatibilità sono stati importanti per chiarire il meccanismo alla base di queste funzioni (fig. 9.13a).
Il replicone F possiede due origini di replicazione: oriT attiva
durante il trasferimento coniugativo (► cap. 12) e oriV (oriF)
l’origine vegetativa della replicazione plasmidica.
Il sito oriV (fig. 9.13a) consiste in due dnaA box riconosciute
dalla proteina iniziatrice del batterio DnaA, una regione ricca
in AT, una regione di 13 nucleotidi ripetuti omologa a quella
di oriC del batterio seguita da quattro sequenze DR di 19 pb
(chiamate iteroni) a cui si lega la proteina iniziatrice RepE
codificata dal plasmide. Il gene codificante per RepE (repE) è
localizzato vicino a oriV.
Il legame della proteina RepE, sotto forma di monomero, ai
quattro iteroni di oriV causa un ripiegamento di questa regione
(fig. 9.13b) che induce localmente una separazione dei due filamenti del DNA che si estende dalla sequenza ripetuta fino alla
regione ricca in AT. Alla separazione dei filamenti del DNA, e
alla stabilità di questa struttura, cooperano le proteine dell’ospite HU e DnaA. Come per il cromosoma batterico, questa
fase di apertura della doppia elica del DNA è essenziale per
dare inizio alla replicazione del plasmide F.
Un altro fattore critico per l’inizio della replicazione è la concentrazione di RepE che modula anche la frequenza degli
inizi di replicazione del fattore F. Il livello cellulare di RepE
è regolato da RepE stessa e dal livello di trascrizione del suo
gene. RepE generalmente esiste come omodimero e sotto
questa forma è inattivo per l’inizio della replicazione ma attivo per la regolazione del gene (repressione autogena). La
conversione del dimero di RepE in monomero (forma attiva
per l’inizio della replicazione) è operata da alcune proteine
chaperon.
L’allungamento della sintesi del DNA del fattore F e di molti
altri plasmidi (così pure quello virale) procede attraverso due
diverse strutture replicative che può assumere il DNA; una è
definita theta (θ) ed è quella tipica del cromosoma batterico,
l’altra è detta sigma (σ) o a rolling circle (cerchio rotante). Le
differenze tra queste risiedono nel modo in cui i genomi sono
replicati. Nel primo caso, il plasmide o il fago viene replicato
sotto forma di genoma circolare; nel secondo, l’innesco è generato dal taglio di un filamento della doppia elica e la replicazione procede mediante l’estensione continua dell’estremità
3′–OH del DNA. Molti virus e plasmidi codificano per proteine che tagliano in modo specifico il legame fosfodiesterico lungo la catena polinucleotidica, generando un’estremità 3′–OH
per l’innesco della sintesi e rimpiazzando così l’attività della
primasi. Un’elicasi srotola il filamento di DNA tagliato così da
allontanare il filamento dallo stampo, mentre avviene l’estensione dal 3′–OH libero per la copiatura del filamento circolare
intatto.
Alcuni genomi possono alternare queste due modalità di replicazione. Il fattore F e altri plasmidi affini, ad esempio, repli-
oriF
Regione
ricca in AT
a)
DnaA-box
Ripetizione
invertita
Iteroni
13mero
incC iteroni
RepE
TTGTGACAAATTGCCCTTT
Dimero RepE
incC
b)
ori represso
Figura 9.13 INIZIO DELLA REPLICAZIONE DEL FATTORE F E CONTROLLO DEL NUMERO DELLE COPIE. (a) L’origine oriF possiede due DnaA box riconosciute dalla
proteina batterica DnaA; una regione ricca in AT; una regione di 13 nucleotidi
ripetuti omologa a oriC del cromosoma batterico seguita da quattro sequenze
DR di 19 pb che legano la proteina iniziatrice RepE. (b) Il legame di RepE in
questa regione causa un ripiegamento del DNA che apre l’origine e ne separa
i due filamenti del DNA. Questa apertura consente, come nel caso del cromosoma batterico, l’inizio della replicazione. Il controllo del numero delle copie
del fattore F è assicurato da un meccanismo detto di “ammanettamento”,
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 17
che sfrutta l’affinità di legame della proteina RepE (oltre che per l’origine) per
sequenze ripetute esterne all’origine chiamate iteroni. A bassa concentrazione
di plasmide e di RepE, questa proteine lega preferenzialmente all’origine di
replicazione. Un aumento della concentrazione di plasmide e di RepA conduce a un legame di questa proteina anche a livello degli iteroni. Un ulteriore
incremento di plasmidi e di RepE provoca un “ammanettamento” del DNA
per dimerizzazione dei monomeri di RepE legati al DNA. La struttura prodotta dall’ammanettamento impedisce la replicazione e l’incremento del numero
delle copie del plasmide.
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 18
ISBN 978-88-08-18686-7
cano secondo il modello θ durante la replicazione vegetativa,
mentre durante il trasferimento coniugativo del DNA lo fanno
secondo il modello σ.
9.5.2 La replicazione di ColE1
ColE1 può essere considerato come modello per lo studio della replicazione di molti plasmidi presenti nella famiglia delle
Enterobacteriaceae.
La replicazione di ColE1 non coinvolge proteine codificate dal
plasmide. ColE1 utilizza, infatti, per la propria replicazione le
proteine dell’ospite. Una regione del plasmide di 600 nucleotidi contiene l’origine di replicazione (oriV) e da questa origine
parte la replicazione che procede in modo unidirezionale. La
replicazione di ColE1 dipende dalla sintesi di un RNA di circa
700 nucleotidi (chiamato RNA II o pre-primer) che parte da
un promotore a monte di oriV (fig. 9.14a). Quando il trascritto
giunge a livello di oriV, si forma un ibrido RNA-DNA. L’RNA
viene quindi tagliato dalla RNasi H, un’endoribonucleasi che
taglia l’RNA appaiato al DNA (H sta per hybrid). L’enzima taglia l’RNA II e lo riduce a una molecola di circa 550 nucleotidi
la cui estremità 3′–OH funge da primer per la DNA Pol I (fig.
9.14b).
L’RNA II è anche il bersaglio di un piccolo RNA complementare (RNA antisenso, detto RNA I) che permette di controllare il numero delle copie del plasmide per cellula (fig. 9.14c).
Durante la replicazione, che procede in modo unidirezionale
secondo il modello θ, l’estensione del DNA del leading strand
e la sintesi discontinua del lagging strand sono invece a carico
di Pol III, mentre il movimento della forca di replicazione è
promosso dalla girasi che partecipa all’apertura del dsDNA.
Lo studio della replicazione in vivo e in vitro di ColE1, in mutanti difettivi per Pol I o RNasi H, o ambedue le attività enzimatiche, ha evidenziato tuttavia che, oltre alla modalità descritta
sopra, ColE1 sfrutta altre due vie d’inizio della replicazione. In
assenza della RNasi H, infatti, il 3′ dell’RNA II non processato
può ancora essere utilizzato dalla DNA Pol I come primer per
la sintesi del filamento leading anche se con minor efficienza.
In assenza della RNasi H e di Pol I, il filamento non trascritto,
che si trova a singola elica a causa della trascrizione dell’RNA
II, può fungere da stampo per primasi e Pol III e promuovere
così l’inizio della sintesi del lagging strand.
9.5.3 Il controllo del numero delle copie del DNA
I plasmidi finora esaminati utilizzano per regolare direttamente o indirettamente la loro replicazione meccanismi di controllo negativo che si esercita a livello dell’inizio della replicazione.
Il risultato del controllo del tasso d’inizio della replicazione è
che ogni plasmide ha un suo numero di copie caratteristico
all’interno della cellula (1-2 copie per i plasmidi di tipo F, 10100 copie per altri). Varie strategie sono utilizzate per controllare il numero di copie di cromosomi e plasmidi; a due di
queste, chiamate iteron binding (legame a sequenze reiterate) e
inhibitor-target (inibitore e bersaglio), sono riconducibili molti
plasmidi. Si tratta nei due casi di meccanismi che permetto-
no la replicazione quando l’elemento di controllo negativo è
dilui­to in seguito all’accrescimento cellulare.
Nel meccanismo detto di “legame all’iterone”, una sequenza di
circa 20 pb ripetuta in serie nella stessa direzione (iteron, iterone) sul genoma costituisce il principale determinante in cis per
il controllo della replicazione. In plasmidi (come il fattore F)
sottoposti a questo tipo di controllo, questi iteroni sono loca-
a)
PRNA II
oriV
-445
-555
O
PRNA I
RNA I
108 nt
b)
RNA II
ori
DNA
5′
3′
RNA-pol
RNasi H
RNA II
RNasi H
DNA
5′
3′
RNA-pol
DNA-Pol I
DNA
5′
3′
RNA-pol
RNA II
c)
RNA I
DNA
5′
3′
RNA-pol
Figura 9.14 CONTROLLO DELLA REPLICAZIONE DEL PLASMIDE COLE1. (a) Organizzazione del sito di inizio della replicazione oriV. La trascrizione dal promotore
PRNA II procede oltre oriV producendo un trascritto lungo, RNA II; quella da PRNA I
genera un piccolo RNA “antisenso” complementare al 5′ di RNA II. (b) L’RNA II
rimane appaiato al DNA stampo a livello del sito oriV. l’RNasi H taglia l’RNA II
in un sito preciso generando un’estremità 3′–OH che funge dal innesco per la
DNA Poli I. (c) Se l’RNA I è appaiato al 5′ dell’RNA II, la formazione dell’ibrido
RNA-DNA a oriV è sfavorita e non si genera così l’innesco per la DNA Pol I.
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 18
RNA II
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 19
ISBN 978-88-08-18686-7
lizzati vicino a ori e al gene rep che codifica per l’iniziatore della replicazione plasmidica. Si pensa che gli iteroni controllino
la replicazione competendo per Rep con i siti di legame della
proteina presenti in ori.
Nel meccanismo inibitore-bersaglio, un RNA antisenso controlla negativamente l’inizio della replicazione. Il bersaglio di
questo RNA antisenso in alcuni casi è l’RNA messaggero che
codifica per la proteina Rep, necessaria per iniziare la replicazione plasmidica, in altri è l’RNA che serve da primer per la
DNA polimerasi, o un suo precursore. In ogni caso, l’appaiamento dell’RNA antisenso regolatore con l’RNA bersaglio
inibisce, con meccanismi diversi, l’attività del bersaglio. Nel
caso della replicazione di ColE1, ad esempio, il meccanismo
inibitore-bersaglio dipende dall’RNA antisenso RNA I che interferisce con la maturazione dell’RNA II.
L’RNA I, un piccolo RNA di circa 100 nt, viene trascritto in
senso opposto alla direzione di RNA II da un promotore localizzato tra il promotore di RNA II e l’origine di replicazione.
Questo RNA è pertanto complementare all’RNA II e forma
un ibrido RNA-RNA stabile che interferisce con l’appaiamento dell’RNA II con il DNA e quindi con il processamento
dell’RNA II da parte dell’RNasi H e la formazione del primer,
inibendo così l’inizio della replicazione di ColE1 (fig. 9.14b).
La replicazione di ColE1 è regolata anche dalla proteina plasmidica Rop, una piccola proteina di 63 aa che favorisce il
legame di RNA I con RNA II e contribuisce quindi a inibire
la replicazione. Il livello dei due inibitori (RNA I e Rop) aumenta con l’aumentare del numero di copie del plasmide, per
cui si crea un meccanismo di retroazione (feed back) negativa che controlla il numero di copie per cellula. Delezioni del
gene rop si traducono con un aumento del numero delle copie
del plasmide. È il caso, ad esempio, del plasmide pBR322 (un
derivato di un plasmide della famiglia ColE1, geneticamente
rop+, molto noto per essere stato il primo vettore utilizzato per
clonare DNA in vitro), con circa 15 copie per cellula, mentre il
numero di copie di pUC18, un suo derivato privo del gene rop,
è dell’ordine di 70 per cellula.
Oltre ai due modelli descritti (F e ColE1) sono stati riscontrati diversi altri meccanismi di replicazione e del suo controllo.
9.5.4 La replicazione dei cromosomi lineari
La presenza di cromosomi lineari nella cellula pone due tipi di
problemi: come proteggere il DNA lineare dall’attacco delle
esonucleasi e, problema ancor più complicato, come replicare
le estremità stesse che richiedono meccanismi specifici senza i
quali la replicazione condurrebbe a una progressiva riduzione
delle dimensioni della molecola di DNA. Infatti la necessità di
un innesco per la DNA polimerasi e la sintesi unidirezionale
5′ → 3′ creano problemi alla replicazione di queste molecole.
Se la sintesi di una nuova elica di DNA a partire da un’estremità avviene grazie a un primer a RNA, al termine della replicazione le due molecole risultanti presentano estremità ibride
RNA-DNA. Successivamente i primer verrebbero rimossi e
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 19
comunque non potrebbero fungere da stampo per le DNA polimerasi, per cui le estremità 5′ delle due eliche nuove risulterebbero più corte (con le estremità 3′ dei due filamenti vecchi
che rimarrebbero a singola elica). Al ciclo di replicazione successivo, i filamenti vecchi darebbero origine a molecole come
quelle ora descritte, mentre quelli nuovi (cui è stato rimosso
l’innesco a RNA al 5′) produrrebbero una molecola più corta
di quella originaria (fig. 9.15).
Diverse strategie sono state sviluppate nei procarioti (inclusi
elementi genetici accessori) per risolvere questi due problemi.
La protezione delle estremità del DNA in molti casi è assicurata convertendo la forma lineare in una circolare chiusa covalentemente oppure, per alcuni batteriofagi, inibendo esonucleasi specifiche. In altri casi cromosomi e plasmidi lineari, come
quelli di Borrelia, hanno le estremità chiuse covalentemente
(fig. 9.16), mentre nel caso di Streptomyces una proteina lega
covalentemente e protegge le estremità dei cromosomi e dei
plasmidi lineari.
La conversione del DNA lineare in una struttura chiusa è il
destino del DNA infettante di molti batteriofagi. Nel caso di
λ, ad esempio, il DNA lineare, appena entra nella cellula, ver-
Origine
Filamento
Filamento
5ʹ
3ʹ
3ʹ
5ʹ
5ʹ
3ʹ
3ʹ
5ʹ
5ʹ
3ʹ
3ʹ
5ʹ
5ʹ
3ʹ
5ʹ
Gap
3ʹ
3ʹ
Gap
3ʹ
5ʹ
5ʹ
5ʹ
5ʹ
3ʹ
3ʹ
3ʹ
5ʹ
5ʹ
3ʹ
Figura 9.15 I PROBLEMI POSTI DALLA REPLICAZIONE DEI CROMOSOMI LINEARI. La sintesi di molecole di DNA lineari genera molecole sorelle ognuna delle quali porta
al 5′ l’RNA primer. Questi primers sono rimossi dall’esonucleasi che agisce in
direzione 5′ → 3′ generando un DNA a singolo filamento che non può essere
riparato o copiato da alcuna DNA Pol nota. In assenza, quindi, di meccanismi
specifici la replicazione dei cromosomi lineari genererebbe ad ogni ciclo molecole di DNA sempre più corte.
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
Materiale online - CAPITOLO 9 20
ISBN 978-88-08-18686-7
rà chiuso covalentemente e sarà replicato secondo due tipi di
modalità, in una prima fase la replicazione theta (θ), e successivamente quella sigma (σ). In altri batteriofagi, come Mu, il
DNA lineare è chiuso mediante una proteina legata covalentemente al DNA, mentre in altri il DNA forma per ricombinazione lunghe molecole composte da genomi multipli dette
concatameri (► cap. 17).
L’innesco della replicazione dei cromosomi lineari avviene
secondo modalità che variano nei diversi organismi. Negli eucarioti una classe specifica di enzimi (telomerasi) riconosce
e replica le estremità dei cromosomi (telomeri). Le telomerasi sono enzimi ribonucleoproteici (RNP), costituiti da una
RNA telomerasi (TER), da una trascrittasi inversa (TERT) e
da parecchi altri componenti che variano nei diversi gruppi di
eucarioti. La subunità a RNA dell’enzima include una breve
regione stampo che viene copiata nella sequenza telomerica
del DNA. Rimandiamo per questo argomento a un testo di
biologia molecolare.
Nei procarioti sono note due tipologie diverse di molecole di
DNA lineare, che vengono replicate secondo modalità diverse (► cap. 8, fig. 9.16). Un primo tipo è costituito dalle molecole di DNA lineare con le estremità 3′ e 5′ dei due filamenti
complementari legate covalentemente tra loro. Hanno questa
configurazione i cromosomi dei batteri appartenenti al genere
Borrelia e i loro plasmidi lineari e il DNA intracellulare di N15,
un batteriofago di E. coli che replica come molecola lineare.
Sono noti anche virus animali (tra cui il virus vaccinico) con
DNA di questo tipo. Il meccanismo preciso di replicazione di
queste molecole non è ancora chiarito, ma è evidente che l’uso
di inneschi a RNA per replicare questa struttura non porta ad
accorciamento dei telomeri. Secondo un ipotetico modello,
la replicazione di una molecola di questo potrebbe produrre
un intermedio circolare con due copie del genoma congiunte
testa-testa e un enzima (detto protelomerasi, ossia telomerasi
procariota) separerebbe le due copie rigenerando le estremità
chiuse covalentemente.
Il secondo tipo di genomi lineari nei batteri è costituito dai
cromosomi lineari degli Streptomiceti, da molti plasmidi
linea­ri presenti in questo gruppo e da batteriofagi della famiglia di φ29, un fago di Bacillus subtilis e altri fagi di Streptococcus pneumoniae, ma anche virus animali come gli adenovirus.
Questi hanno una proteina (terminal protein, TP) legata covalentemente a ciascuna estremità 5′ del genoma. La proteina
TP funge da primer per la replicazione, fornendo come innesco
per la DNA polimerasi un residuo OH di una sua specifica serina, treonina o tirosina, rimanendo poi legata covalentemente
all’estremità 5′ della nuova elica (fig. 9.16b). Nel caso del cromosoma degli streptomiceti, la replicazione bidirezionale procede a partire da un’origine di replicazione localizzata all’interno del genoma, e le proteine terminali sembrano implicate
solo nella replicazione dei telomeri. Per genomi più piccoli,
come quello del fago di φ29 e di alcuni plasmidi, la duplicazione di ciascuna elica inizia dalla TP ad ogni estremità e attraversa l’intero genoma.
a)
L
Inizio della
replicazione
L
R
Molecole
replicate
Lʹ L
Giunzione
Rʹ R
Risoluzione
del telomero
L
R
L
R
b)
ori
TP
TP
TP
TP
TP
TP
TP
TP
TP
TP
Figura 9.16 LA REPLICAZIONE DEI PLASMIDI LINEARI DI BORRELIA E STREPTOMYCES.
(a) Modello di replicazione di molecole di DNA lineari con estremità chiuse covalentemente (telomeri a forcina). Esempio: Borrelia. La replicazione che parte dal
sito ori forma un dimero “testa-testa” che viene risolto da una “protelomerasi”
(telomerasi procariota). (b) Modello di replicazione di molecole di DNA lineari
con proteina terminale (TP). Esempio: Streptomyces. Ad ogni ciclo replicativo
due nuove TP si associano alle estremità della molecola e forniscono l’innesco
alla DNA polimerasi per aggiungere il primo desossiribonucleotide e replicare
così le regioni telomeriche. La replicazione che parte dai telomeri potrà congiungersi alla replicazione che parte da un sito ori interno.
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 20
R
ori
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07
ISBN 978-88-08-18686-7
9.6La replicazione negli Archaea
La replicazione degli Archaea è una sorta di mosaico tra il sistema eucariote e quello batterico cui si aggiungono aspetti specifici. In questi ultimi anni alcuni Archaea hanno fornito modelli
di studio attraenti per la replicazione del DNA in quanto contengono una versione semplificata, ma omologa, al core del
macchinario di replicazione eucariote. Un esempio di studio
di questo macchinario di replicazione è quello dell’ipertermofilo Sulfolobus solfataricus. Questo microrganismo è un buon
sistema modello per lo studio della replicazione degli Archaea:
può crescere in laboratorio in aerobiosi, in terreno liquido o
solido a temperature comprese tra 75-80 °C e a un pH di 2 o 3,
condizioni in cui le sue proteine sono molto stabili.
S. solfataricus, come molti archei, ha un solo cromosoma circolare che, contrariamente ai batteri, può contenere più origini di
replicazione attive (come nel caso dei cromosomi lineari degli
eucarioti con più origini).
Quasi tutti gli Archaea possiedono almeno un omologo delle
proteine iniziatrici eucariote Orc1 e Cdc6, coinvolte nella formazione del complesso pre-replicativo (pre-RC). Per quanto
riguarda il replisoma, gli aspetti più noti sono i seguenti:
Materiale collegato a Dehò, Galli - Biologia dei microrganismi - II ed.
266-268_Cap09_Deho_web.indd 21
Materiale online - CAPITOLO 9 21
1. L’elicasi in Sulfolobus, chiamata MCM (mini-chromosomes
maintenance), è omologa a quella di S. cerevisiae dove è stata
inizialmente identificata in base al comportamento di alcuni mutanti incapaci di mantenere stabilmente minicromosomi nella cellula, suggerendo un ruolo di queste proteine
nella replicazione. MCM è un’elicasi esamerica necessaria
per la progressione della forca replicativa. Negli eucarioti
l’esamero è costituito da sei subunità diverse, mentre negli
Archaea le subunità sono identiche.
2. La primasi in S. solfataricus è un complesso di due proteine
(PriS e PriL) omologhe a quelle eucariote. È presente anche
un omologo della primasi DnaG batterica, la cui funzione,
tuttavia, non è nota.
3. In S. solfataricus ci sono tre polimerasi della famiglia B (Pol
B1, B2 e B3) affini alle polimerasi eucariote, ma poco è
noto circa la loro attività in vivo. Questi enzimi purificati
hanno bassa affinità per il DNA e se ne dissociano frequentemente durante la sintesi. L’incremento della processività
dell’enzima durante la replicazione è accoppiato a uno sliding clamp specializzato che circonda il DNA senza legarlo
strettamente.
©2014 CEA - Casa Editrice Ambrosiana
01/09/14 09:07

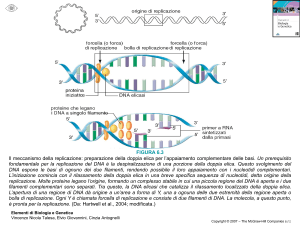


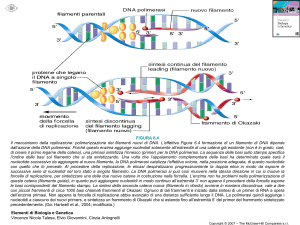




![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
