Esercitazione di Laboratorio corso Prof Tuberosa – Biotecnologie genetiche agrarie
Laurea in Biotecnologie (terzo anno).
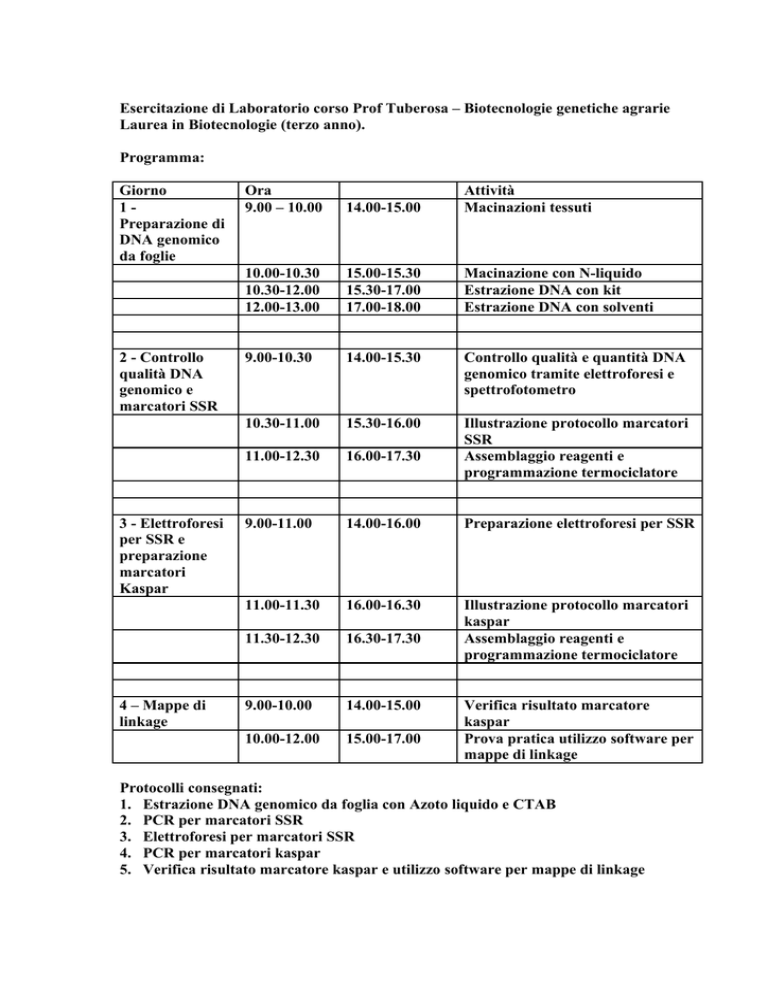
Programma:
Giorno
1Preparazione di
DNA genomico
da foglie
2 - Controllo
qualità DNA
genomico e
marcatori SSR
3 - Elettroforesi
per SSR e
preparazione
marcatori
Kaspar
4 – Mappe di
linkage
Ora
9.00 – 10.00
14.00-15.00
Attività
Macinazioni tessuti
10.00-10.30
10.30-12.00
12.00-13.00
15.00-15.30
15.30-17.00
17.00-18.00
Macinazione con N-liquido
Estrazione DNA con kit
Estrazione DNA con solventi
9.00-10.30
14.00-15.30
Controllo qualità e quantità DNA
genomico tramite elettroforesi e
spettrofotometro
10.30-11.00
15.30-16.00
11.00-12.30
16.00-17.30
Illustrazione protocollo marcatori
SSR
Assemblaggio reagenti e
programmazione termociclatore
9.00-11.00
14.00-16.00
Preparazione elettroforesi per SSR
11.00-11.30
16.00-16.30
11.30-12.30
16.30-17.30
Illustrazione protocollo marcatori
kaspar
Assemblaggio reagenti e
programmazione termociclatore
9.00-10.00
14.00-15.00
10.00-12.00
15.00-17.00
Verifica risultato marcatore
kaspar
Prova pratica utilizzo software per
mappe di linkage
Protocolli consegnati:
1. Estrazione DNA genomico da foglia con Azoto liquido e CTAB
2. PCR per marcatori SSR
3. Elettroforesi per marcatori SSR
4. PCR per marcatori kaspar
5. Verifica risultato marcatore kaspar e utilizzo software per mappe di linkage
Estrazione DNA genomico da foglia con Azoto liquido e CTAB
L’estrazione del DNA da tessuti vegetali può risultare assai diversa a seconda del materiale di partenza
utilizzato. In generale si basa sempre su una rottura meccanica della membrana cellulare che consente
l’accesso al materiale nucleare senza però degradarne il contenuto. Per questo è generalmente utilizzato
una macinatura con azoto liquido per rompere la parete cellulare e consentire l’accesso al DNA
mantenendo la maggior parte degli enzimi cellulari inattivi. Una volta che i tessuti sono
sufficientemente macinati può essere risospeso in un buffer di lisi cellulare come il CTAB.
Successivamente, la maggior parte dei protocolli di estrazione prevede un passaggio in fenolo o
fenolo:cloroformio per purificare il DNA dalla componente proteica che si solubilizza nella fase
organica mentre il DNA rimane nella fase acquosa. Il DNA viene successivamente precipitato dalla fase
acquosa mediante isopropanolo e lavato in etanolo per rimuovere sali di contaminazione. Il DNA
purificato è poi risospeso in acqua (o TE 0,1 se necessario conservarlo per periodo più a lungo). Per
verificare la qualità del materiale estratto si può utilizzare un gel di agarosio.
1. Preparare tubi falcon da 50 ml.
2. Macinare 6-9 g di tessuto fogliare fresco con azoto liquido e porlo nel tubo falcon facendo
attenzione che tutto l’azoto sia ben evaporato.
3. Aggiungere 20 ml di CTAB.
4. Mettere i campioni in stufa o bagno termostatico a 65° C per circa 90 minuti, agitare ogni 15
minuti.
5. Una volta raggiunta la temperatura ambiente aggiungere ugual volume di cloroformio e
mescolare per inversione.
6. Centrifugare a 2000 g (3400 rpm) per 10 minuti.
7. Trasferire la fase superiore in un nuovo falcon da 50 ml.
8. Ripetere i passaggi 5-6-7 per una migliore qualità del DNA.
9. Aggiungere ugual volume di isopropanolo. Mescolare per inversione alcuni minuti per far
floculare il DNA.
10. Prelevare il floculo con un’ansa sterile e porlo in un eppendorf da 2 ml e aggiungere 1,5 ml
di Etanolo 70% e lasciare a 4° per almeno 1 ora o over-night.
11. Prelevare il DNA e risospendere in TE 0,1 (1-2 ml a seconda della grandezza della
grandezza della nuvola).
12. Si conserva in freezer a -20°
13. Se al punto 10 non è visibile un floculo centrifugare a 2000 g per 15 minuti, svuotare il
liquido per inversione e aggiungere 3 ml di Etanolo 70%. Lasciare over-night. Centrifugare a
2000g per 10 minuti e rimuovere l’etanolo per inversione dei tubi. Asciugare a temperatura
ambiente per alcune ore o over-night. Risospendere il pellet in 0.5 - 1 ml di TE 0.1 e trasferirlo
in eppendorf.
C-TAB
600 ml solution
dH2O
TRIS 1M pH 7.5
NaCl 5M
EDTA 0.5M pH 8
CTAB
TE 0.1X
50 ml solution
440 ml
60 ml
84 ml
12 ml
6g
dH2O
TRIS 1M pH 8
EDTA 0.5M
49.5 ml
500 µl
10 µl
ATTENZIONE: Solventi organici come il cloroformio devono essere utilizzati sotto cappa e
maneggiati con guanti di protezione.
Estrazione DNA genomico da foglia con DNeasy Plant Mini Kit
Controllo qualità e quantità DNA genomico tramite elettroforesi e spettrofotometro
– Caricare il DNA estratto in un gel di agarosio 1%
ATTENZIONE: Il gel contiene Bromuro di Etidio, sostanza mutagena che può indurre effetti
carcinogenici. Maneggiare con guanti e occhiali di protezione sia il gel sia le vaschette di corsa
elettroforetica.
– Caricare i seguenti campioni in pozzetti separati
5 μL 100 bp ladder
λ DNA 5 ng/ μL 2 μL (10 ng DNA)
λ DNA 5 ng/ μL 10 μL (50 ng DNA)
λ DNA 10 ng/ μL 10 μL (100 ng DNA)
5 μL DNA sample + 5 μL acqua + 2 μL 6x Loading Buffer preparato su parafilm
– Correre il gel per 30 min at 100 V
– Esporre il gel alla luce UV e fotografarlo
La presenza di una banda ad alto peso molecolare indica DNA di buona qualità, la presenza di una
scia di bande a diversi pesi molecolari indica DNA degradato.
La concentrazione del DNA estratta può essere stimata anche utilizzando uno spettrofotometro che
stima la concentrazione del DNA sulla base dell’assorbanza a 260 (tipica degli acidi nucleici) e la
purezza sulla base del rapporto 260/280 (proteine) e 260/230 (zuccheri). Più comunemente nei
laboratori e presente un biofotometro cioè uno spettrofotometro semplificato in cui il parametro
lunghezze d'onda viene automaticamente impostato con la scelta del metodo. Tale metodo non
consente però di verificare la qualità del DNA estratto poiché anche DNA degradato è considerato
nella stima della concentrazione che spesso risulta quindi sovrastimata.
L’assorbanza di 1 OD è equivalente a circa 50 μg/ml
concentrazione del DNA (μg/ml) = A260 x 50 μg/ml x il fattore di diluizione
esempio di DNA genomico non degradato (prima riga) e in parte degradato (seconda riga) che
si presenta come una striscia di frammenti a diversi pesi molecolari.
esempio di DNA genomico altamente degradato in cui è comunque ancora presente una
quantità di DNA ad alto peso molecolare.
AMPLIFICAZIONE PCR per SSR
- Diluire il DNA genomico alla concentrazione di 20 ng/µl
(esempio: se la concentrazione stimata al biofotometro fosse di 300 ng/µl (Ci) e volessi preparare
30 µl di soluzione finale (Cf) si applica la formula Ci x Vi = Cf x Vf quindi 300 x Vi = 20 x 30
dove Vi = (20 x 30) /300 = 2 quindi metterò 2 µl del mio campione iniziale a 300 ng/µl in 28 µl di
acqua)
- Preparare la mix (n° campioni + 1 ogni 10 campioni):
[ ] iniziale
H2O
PCR Reaction buffer
Magnesium chloride
dNTP
Primer forward
Primer reverce
Taq polymerase
DNA
TOTALE
H2O
[ ] finale
Vx1
a volume finale
5X
1X
25 mM
2 mM
25 mM each
0.2 mM
10 µM (10 pmol/µl) 0.5 µM
10 µM (10 pmol/µl) 0.5 µM
0.3 U
5 U/µl
100 ng
20 ng/µl
20
Ci
Cf
[ ] iniziale
[ ] finale
a volume finale
PCR Reaction buffer 5 X
Magnesium chloride 25 mM
dNTP
25 mM each
Primer forward
10 µM (10
pmol/µl)
Primer reverce
10 µM (10
pmol/µl)
Taq polymerase
5 U/µl
DNA
20 ng/µl
TOTALE
Vx1
20-(4+1,6+0,16+1+1+0,06+5) 7,18
Vx5
campioni
35,9
1X
2 mM
0.2 mM
0.5 µM
(1*20)/15
(2*20)/25
(0.2*20)/25
(0,5*20)/10
4
1,6
0,16
1
20
8
0,8
5
0.5 µM
(0,5*20)/10
1
5
0.3 U
100 ng
0,3/5
100/20
0,06
5
Vf 20
0,3
25
100
Ci x Vi = Cf x Vf
Vi = Cf x Vf /Ci
- Aliquotare 5 µl di DNA gnomico diluito in ciascuna eppendorf da 0.2 ml
- Aggiungere 15 µl di master mix per eppendorf
-
V x 5 campioni
PCR: programma amplificazione:
Cicli
1
15
22
T°
95 °
95 °
63.5 ° - 0.5° ciclo
72 °
94 °
56 °
72 °
72 °
Primer
Concentrazione stock Concentrazione finale
100 M (100 pmol/µl)
10 µM (10pmol/µl)
µl
stock
10
Durata
3’
30 s
1’
1’
30 s
1’
1’
7’
+ TE 0.1X
90 µl
Preparare le soluzioni stock (100 µM ovvero 100 pmol/µl) in TE 0,1 e conservarle a –20° C
Le successive diluizioni vanno conservate a 4° C e rifatte ogni 20-30 giorni.
PCR reaction buffer
Utilizzare sempre il buffer fornito con la Taq Polimerasi che si sta utilizzando
Conservarlo a – 20° C
MgCl2
Viene, in genere venduto insieme alla TAQ e nel caso della PROMEGA alla concentrazione di 25
mM (stock solution) Si utilizza alla concentrazione finale di 0,5-4 mM
Conservare a – 20° C. e vortexare prima di usare (il magnesio tende a precipitare). I dNTPs legano
il magnesio; se si varia la concentrazione di dNTPs, la concentrazione di Mg++ va compensata.
Eccesso di Mg++: accumulo di prodotti aspecifici.
Carenza di Mg++: riduzione della resa (il Mg++ e' il cofattore della Taq Polimerasi).
DNTPs
Vengono, in genere venduti singolarmente (dATP, dCTP, dGTP, dTTP) alla concentrazione
100mM (stock solution)
Si preparara una miscela contenente una ugual dose di ciascun nucleotide.
Conservare a – 20° C
H2O
Utilizzare acqua MilliQ autoclavata e monodose
ELETTROFORESI PER MARCATORI SSR
–
–
Preparare gli appositi supporti inserendo un pettine da 15 pozzetti
Preparare un gel di agarosio 2% sciogliendo 1 g di agarosio in 50 ml di TBE 0,5x
utilizzando un forno a micronde. Lasciare raffreddare alcuni minuti ed aggiungere 1 μl di etidio
bromuro mescolando dolcemente.
ATTENZIONE: Il Bromuro di Etidio è una sostanza mutagena che può indurre effetti
carcinogenici. Maneggiare con guanti e occhiali di protezione sia il gel sia le vaschette di corsa
elettroforetica.
–
Versare il gel e lasciare raffreddare per ca. 30 minuti
– Caricare i campioni e uno standard di riferimento (5 μL) 100 bp ladder
– Correre il gel per ca. 1 h
– Esporre il gel alla luce UV per visualizzare le bande
Stima della concentrazione di agarosio: Più alta è la concentrazione di agarosio, più è piccolo il
frammento di DNA che può essere visualizzato. I polimeri del gel agiscono infatti come una maglia
e più è alta la concentrazione del gel più piccoli sono i pori della matrice e più è difficile per
frammenti lunghi di DNA muoversi attraverso di essa. C’è comunque un limite per la separazione
accurata delle molecole di DNA mediante elettroforesi come riportato nella tabella che segue. Per
stimare il peso di un frammento di DNA e stimare le condizioni di elettroforesi ottimali è comunque
sempre opportuno inserire anche un marcatore di peso molecolare (es 100 bp DNA Ladder) assieme
ai campioni. Grazie alla presenza dei gruppi fosfato (PO43-) il DNA è carico negativamente e
migrerà quindi verso il polo positivo.
Resolution of Linear DNA on Agarose Gels
Recommended % Agarose Optimum Resolution for Linear DNA
0.5
1,000–30,000bp
0.7
800–12,000bp
1.0
500–10,000bp
1.2
400–7,000bp
1.5
200–3,000bp
Minima quantità di DNA rilevabile come banda singola tramite fotografia di un gel colorato con
Etidio Bromuro: 5 ng (1-10 ng) in un pozzetto largo 4,5 mm.
Massima quantità di DNA separabile come banda singola senza effetti di "striscio" o
"trascinamento": 200 ng in un pozzetto largo 4,5 mm.
Voltaggio massimo al quale si mantiene la proporzionalità inversa tra peso molecolare e mobilità
(log10): 5 volts/cm. (lunghezza del gel).
KASP genotyping technology
Questo tipo di marcatori (KBiosciences Competitive AlleleSpecific PCR SNP genotyping system;
KASPar) è un tipo di marcatore per l’identificazione di SNP o indel in una sequenza genomica. Si
tratta quindi di una PCR allele specifica.
La chimica alla base della tecnica kaspar può essere definita FRET (Fluorescence Resonance
Energy Transfer). Generalmente in queste tecniche la sonda è costituita da due fluorofori uno detto
quencher (Q) che riduce la fluorescenza del reporter (R). Quando i due fluorofori non sono più in
contatto si ha emissione di fluorescenza alla lunghezza d’onda del reporter.
La master mix di PCR comprende la “probe” e la “mix”
Probe: Comprende
• 2 primer forward fluorescenti allele specifici (FAM e VIC) legati ad una sequenza di 18 bp (tail)
• due oligo complementari ai primer forward marcati legati ad un quencer che inizialmente
ibridizzano ai primer forward marcati annullando il segnale.
• 2 primer forward allele specifici non marcati legati ad una sequenza di 18 bp (tail)
• 1 primer reverce comune non marcato.
MIX: Nella miscela di PCR viene inoltre fornita una Taq DNA polimerasi che non è in grado di
estendere primer che al 3’ non siano perfettamente appaiati.
La mix di PCR è composta da Buffer, Mg, dNTP ed un dye standard interno (Rhodamine X, (Rox))
per normalizzare il segnale di fluorescenza dello strumento.
Maggiori informazioni possono essere trovate nel sito: http://www.kbioscience.co.uk/reagents/KASP.html
- Diluire il DNA genomico alla concentrazione di 10 ng/ul
- Preparare la mix (n° campioni + 1 ogni 10 campioni):
[ ] iniziale
H2O
Reaction mix
2X
[ ] finale
Vx1
V x 5 campioni
a volume finale
1X
containing ROX as a passive reference
Primer (Assay mix)
DNA
TOTALE
0.22
10 ng/µl
15 ng
15
- Aliquotare 1.5 µl di DNA genomico in piastre da 96 pozzetti
- Aggiungere 13.5 µl di master mix per eppendorf
- PCR: programma amplificazione:
Cicli
1
T°
94 °
94 °
63 °
34
Durata
15’
10 s
1’
Schema della piastra per lettura genotipi noti:
A
B
C
D
E
F
G
H
1
H2O
P1
P2
F1
2
3
F1
4
F1
5
6
F1
P1
P2
F1
7
8
9
10
F1
11
F1
12
H2O
P1
P2
F1
F1
H2O
F1
H2O
F1
La fluorescenza emessa dai campioni amplificati è letta ed analizzata trasferite apposito software
(7000 system SDS software). La fluorescenza di ciascun campione è rappresentata in un asse
cartesiano dove nell’asse delle ascisse vi è la fluorescenza del fluorocromo VIC e nelle ordinate
quella del fluorocromo FAM.
E’ attesa una separazione di 3 nuvole di punti corrispondenti ai due omozigoti e all’eterezigote
nonché 4 campioni con emissione ridotta che corrispondono ai controlli in cui nella reazione PCR
era stata messa acqua anziché DNA.
Per poter assegnare con maggiore precisione il corretto genotipo ai diversi campioni è quindi
importante avere numerosi controlli nella piastra di PCR.
I quattro campioni controllo in cui la reazione di PCR è stata effettuata con acqua al posto del DNA
sono rappresentati con un quadrato grigio e si collocano nella posizione attesa con fluorescenza
significativamente inferiore rispetto a tutti gli altri campioni.
Ai campioni il cui genotipo era noto a priori prima della PCR e che erano stati inseriti solo come
controlli è stato assegnato un valore corrispondente all’allele X (gaspé), all’ellele Y (B73) o
all’eterozigote. Poiché era atteso che la maggior parte dei campioni usati nella reazione di PCR
fosse omozigote per uno dei due parentali, come controlli erano stati inseriti molti eterozigoti al fine
di discriminare con precisione questa classe.
A tutti i campioni viene assegnato un genotipo sulla base della loro fluorescenza permettendo così
di distinguere le 3 classi genotipiche attese. Alcuni campioni, collocati in posizioni di difficile
interpretazione, vengono lasciati indeterminati.



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)




![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)

