Esercitazioni di Chimica Biologica
AA 2005-06
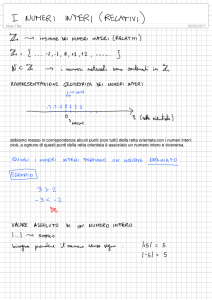
PURIFICAZIONE DI UNA PROTEINA DI FUSIONE
GST-CATECOLO 1,2 DIOSSIGENASI
Viene utilizzato il pellet cellulare congelato ottenuto dalla crescita di cellule di E. coli
BL-21 trasformate con il plasmide pGEXisoB, (contiene la sequenza che codifica ed
esprime una proteina di fusione GST-catecolo 1,2 diossigenasi).
DESCRIZIONE DETTAGLIATA
SETTIMANA 1:
•
Lisi cellulare tramite sonicazione (4 x15‘’) con sonicatore
Misonix già in dotazione (in laboratorio di Biochimica). Dato che è
disponibile un solo sonicatore verrà effettuata dagli esercitatori.
•
Chiarificazione dell’estratto in aliquote (una per ogni
gruppo) tramite centrifugazione su minifuge 12000 rpm per 10 minuti
(effettuato dagli esercitatori come sopra).
•
Ogni gruppo riceve una aliquota di ESTRATTO GREZZO
CHIARIFICATO (EG) (ossia il surnatante della centrifugazione
precedente). Gli operatori terranno da parte una aliquota di EG per il
dosaggio di proteine (settimana 2) per ciascun gruppo.
•
Montaggio colonnine: verrà illustrato in laboratorio
•
Equilibratura delle colonne di affinità GSTrap preimpaccate (Pharmacia): ogni colonna monta in testa una siringa da 10 ml in
plastica che funziona da contenitore per il liquido di lavaggio. Pipettare
nella siringa 5 ml di BINDING BUFFER. Inserire MOLTO
DELICATAMENTE il pistone della siringa ed esercitare una leggera
pressione. Il flusso non deve superare la velocità di 1 ml/minuto, ossia
una goccia in uscita dalla colonna (circa 50 µl) ogni 3/4 secondi. Il liquido
che esce dalla colonna va raccolto in un becker. ATTENZIONE: non fate
entrare aria nella colonna o si danneggia!!! Per questo è meglio lasciare un
pochino di liquido nella siringa e non svuotarla completamente. Chiudere la
colonna in uscita con il tappino nero e staccare la siringa dalla colonna
SENZA MUOVERE IL PISTONE
•
Purificazione del chiarificato su colonne di affinità
GSTrap pre-impaccate (Pharmacia): una volta equilibrata la colonna con il
BINDING BUFFER (ossia tampone di legame: favorisce l’interazione tra
la proteina di fusione che contiene la GST e il glutatione-sepharose
presente in colonna come matrice di cromatografia) è necessario caricare
il campione in colonna. Prelevare il campione (misurare il volume di 1 ml e
inserire il dato nella TABELLA B) con una siringa da 2.5 ml. Eliminare le
bolle d’aria dalla siringa e inserirla (SENZA AGO!!!) in testa alla
colonnina. Caricare in colonna ad un flusso se possibile ancora < di 1
ml/min (calcolare circa 6 secondi tra ogni goccia). Durante il caricamento
raccogliere il liquido in uscita in provetta tipo eppendorf da 1.5 ml
(prepararsi provette numerate da 1 a 15) La raccolta manuale delle
frazioni (1 ml/provetta) va effettuata durante il caricamento per
raccogliere la frazione del NON LEGATO (NL: provetta 1).
•
Finito il caricamento del campione chiudere la colonna,
staccare la siringa e sostituirla con la siringa da 5-10ml contenente
BINDING BUFFER. Effettuare il lavaggio ( almeno 8 ml) con il BINDING
BUFFER e raccogliere le frazioni in uscita ( 1 ml/provetta, provette 2-9).
•
Chiudere la colonna, staccare la siringa e sostituirla con
un’altra siringa da 10ml contenente 5 ml di ELUTION BUFFER. Questo
tampone contiene glutatione in concentrazione 10 mM e determina
l’eluizione specifica della glutatione-S-transferasi (GST) che si era
legata al glutatione-sepharose e che ora si lega al glutatione libero fatto
fluire in colonna e si ritrova quindi nel liquido in uscita. La nostra proteina
di fusione, legata alla GST dovrebbe essere eluita specificamente a
questo stadio. Raccogliere con attenzione le frazioni in uscita (1
ml/provetta, provette 10-15).
•
Mettere le provette contenenti le frazioni (1-15) in
ghiaccio. Chiudere la colonna. Sostituire la siringa in testa alla colonna
con una contenente 10 ml di BINDING BUFFER e farlo fluire a 1 ml/min
(per ristabilire le condizioni iniziali). Non è necessario conservare il
liquido in uscita. Chiudere la colonna e togliere la siringa. La colonna è
pulita e pronta per il turno successivo.
•
Le frazioni eluite contengono proteine di vario tipo, la cui
presenza e concentrazione può essere stimata leggendo la loro
assorbanza a 280 nm in cuvette di quarzo su spettrofotometro. Un
gruppo alla volta effettuerà la lettura dei valori di assorbanza delle
frazioni, da riportare nella TABELLA A.
•
Riportare i valori della TABELLA A su carta millimetrata
in un sistema cartesiano dove x= numero frazione e y=assorbanza a 280
nm. Si disegna così un cromatogramma.
•
Individuare i picchi del cromatogramma nella regione del
non legato e scegliere le frazioni di tale picco da etichettare come NLn°gruppo/n° turno/data, e quelle nella regione dell’eluito specifico, da
etichettare come CAT-n°gruppo/n°turno/data. Inserire il volume di NL e
di CAT nella tabella B
•
SAGGIO DI ATTIVITÀ ENZIMATICA della catecolo
1,2 diossigenasi tramite produzione del cis,cis muconato rilevabile a 260 nm
(FIGURA
OH
6
5
1)
1
4
Cat1,2DO
OH
OH
O2
OH
OH
2
3
OH
Catecolo
COOH
COOH
Acido cis-cis muconico
FIGURA 1
in cuvette di quarzo su spettrofotometro Beckmann DU 70 per i campioni
prelevati a varie fasi della purificazione (EG, NL e CAT).
DOSI PER SAGGIO DI ATTIVITÀ:
930 µl di tampone HEPES 50 mM pH 7.0
20 µl di catecolo 10 mM
mettere nella celletta, fare il blank (START) e poi aggiungere
50 µl di campione, mescolare velocemente e avviare il monitoraggio a 260 nm
(RUN). La lettura avviene per 10 secondi.
INTERPRETAZIONE DEL CROMATOGRAMMA E CONGELAMENTO DELLE
FRAZIONI OTTENUTE.
TABELLA A
Numero frazioni
1
2
3
4
5
6
7
8
9
10
11
12
ASSORBANZA 280 nm
Cromatogramma
3
2,9
2,8
2,7
2,6
2,5
2,4
2,3
2,2
2,1
2
1,9
Abs 280 nm
1,8
1,7
1,6
1,5
1,4
1,3
1,2
1,1
1
0,9
0,8
0,7
0,6
0,5
0,4
0,3
0,2
0,1
0
0
1
2
3
4
5
6
7
8
frazione
9
10
11
12
13
14
15
16
SETTIMANA 2:
•
Allestimento di una curva di taratura con diluizioni
crescenti di standard proteico [BSA], incubazione con reattivo di
Bradford per il dosaggio proteico e lettura delle frazioni a 595 nm.
Estrapolazione della retta e discussione sui risultati della linerizzazione
•
Diluizioni dei campioni proteici ottenuti dalla settimana 1
(aliquote di estratto grezzo, EG, eluito specifico CAT), incubazione con
reattivo di Bradford, lettura a 595 nm e applicazione della retta di
taratura PER OTTENERE LA CONCENTRAZIONE PROTEICA NELLE
VARIE FASI DELLA PURIFICAZIONE.
•
CALCOLO
DELL’ATTIVITÀ
SPECIFICA
(attività
totale/proteine totali), del FATTORE DI PURIFICAZIONE (attività
specifica finale / attività specifica iniziale) e della RESA ( (attività
totale finale / attività totale iniziale) x 100) e discussione dei risultati
TABELLA B
Volume Concentrazione Proteine Attività per
Attività Attività
Attività
Fattore
specifica
di
totale
proteine
totali
50 µl di
totale
(ml)
(mg/ml)
(mg)
campione
(∆Abs/ (∆Abs/min/
(∆Abs/min) min)
specifica
Resa %
(∆conc M purificaz
mg proteina) /min/mg
ione
proteina)
EG
1
C1,2DO
Dosaggio di proteine (metodo di Bradford)
•
Il metodo rileva la presenza delle proteine ssorb il colorante (Coomassie Blu)
si lega a residui di aminoacidi basici (es. ssorben) presenti nelle proteine.
Si utilizza uno standard a concentrazione nota (solitamente si utilizza come
proteina standard la siero albumina bovina o BSA) allestendo una retta di
taratura
•
Allestimento di una retta di taratura
operativamente si parte da una soluzione a concentrazione 100 µg/ml di BSA e
si preparano alcune diluizioni (utilizzare acqua bidistillata come diluente).
•
100
•
•
•
•
•
•
es. in questo caso ogni gruppo deve preparare (in provette tipo eppendorf sulle
quali si saranno segnati i valori delle concentrazioni di BSA!) 1 ml di ciascuna
delle seguenti concentrazioni di BSA:
o 1 µg/ml
o 2 µg/ml
o 5 µg/ml
o 7 µg/ml
o 10 µg/ml
Allestire in tabella 1 uno schema della quantità in µl di soluzione di BSA
concentrata e di diluente (acqua) per ciascuna diluizione.
Utilizzare la formula C1V1=C2V2 dove.
o C1= 100 µg/ml
o V1= x µl di soluzione di BSA concentrata da prelevare
o C2= la concentrazione voluta, es. 2 µg/ml
o V2= 1000 µl
per ciascuna diluizione: prelevare 500 µl, trasferirli in una cuvetta in plastica e
aggiungere 500 µl del reagente colorante di Bradford.
si otterrà una serie di cinque cuvette contenenti gli standard per costruire la
retta di calibrazione (ricordarsi a quali concentrazioni corrisponde ciascuna
cuvetta!!!)
si leggono allo spettrofotometro a 595 nm le intensità di colorazione ottenute
(ossia i valori di ssorbenza) dopo 15’ di incubazione con il colorante (il colore è
stabile per circa 45’)
impostando un grafico dove x=concentrazione in µg/ml e y= ssorbenza i punti
sperimentali dovrebbero disporsi a definire una retta, i cui valori di pendenza
ed intercetta (con il corrispondente coefficiente di correlazione) verranno
estrapolati tramite l’uso di una calcolatrice scientifica (metodo dei minimi
quadrati)
la retta ottenuta (retta di calibrazione) ci permette di ottenere poi i valori di
concentrazione dei nostri campioni come descritto qui di seguito.
Lettura della concentrazione dei campioni
1. diluire in acqua bidistillata i campioni secondo lo schema qui sotto, preparando
almeno 1000 µl di soluzione (NB: diluire 1:10 significa prendere 1 parte di
campione da diluire in 10 parti totali, ossia + 9 parti di diluente e non + 10 parti
di diluente! E’ un errore frequente, soprattutto all’inizio, fate attenzione!)
o frazione EG:1:10000, 1:5000 (diluizioni seriali: intermedia 1:100, 200 ul e
poi da questa preparare 1 ml diluendo 1.50 e 1:100)
o frazione CAT: per abs 280 da 0 a 0.05 non diluire e 1:4
•
per abs da 0.05 a 0.25: dil 1:4 e 1:8
• per abs da 0.25 a 0.6 dil 1:10 e 1:50
• per abs da 0.6 a 1 dil 1:50 e 1:100
• per abs oltre 1: dil 1:100 e 1:300
2. Per ciascuna diluizione prelevare 500 µl, trasferirli in cuvetta ed aggiungere
500 µl di colorante di Bradford (come per la retta di taratura!). Si otterranno
due cuvette (a diluizione diversa) per ogni campione che verranno anch’esse
lette allo spettrofotometro a 595 nm. Dopo 15’ di incubazione con il colorante
(il colore è stabile per circa 45’)
3. l’assorbanza ottenuta verrà correlata ad una concentrazione corrispondente
tramite la retta di taratura: l’assorbanza (y) corrisponderà ad un valore di x
concentrazione dedotto dall’equazione della retta. Riportare i valori di x e y
nella tabella 2 e disegnare il grafico sull’asse cartesiano fornito.
4. ottenuti i valori di concentrazione moltiplicare per il fattore di diluizione per
ottenere il valore di concentrazione nei campioni
Gruppo……………………
Data…………………………
Tabella 1
Concentrazione
1.0
2.0
5.0
7.0
10.0
Volume BSA concentrata (µl)
Volume di acqua (µl)
Tabella 2
Conc. (µg/ml) (x)
1.0
2.0
5.0
7.0
10.0
Assorbanza 595nm (y)
Tabella 2°
campione (diluizione)
1:……..
1:………
1:……….
EG (diluzione)
1:……..
1:………
1:……….
Assorbanza 595nm (y)
Riportare l’equazione della retta estrapolata dai dati della tabella 2
y= bx + a
b=……………..
a=……………..
Abs595=…………….Conc (µg/ml) +…………..
Coefficiente di correlazione (r o r2) =……………….
Assorbanza 595 nm
2
1,9
1,8
1,7
1,6
1,5
1,4
1,3
1,2
1,1
1
0,9
0,8
0,7
0,6
0,5
0,4
0,3
0,2
0,1
0
0
5
10
15
Concentrazione (µg/ml)
20
25
Dosaggio di
attività
catecolo 1,2
diossigenasica
Si misura la produzione di acido cis,cis-muconico che assorbe a 260 nm. Il saggio si
effettua in cuvette di quarzo in cui si porranno
- 960 µl di tampone HEPES 50 mM pH 7.0
- 20 µl di una soluzione 10 mM di catecolo (substrato di reazione)
- si calibra lo strumento su questo “blank” e si aggiungono poi
- 20 µl di campione da saggiare (EG, NL o CAT)
Dopo quest’ultima aggiunta la reazione deve partire velocemente e in cuvetta la
miscela di reazione deve essere omogenmeea (ben mescolata) per misurare in modo
affidabile la velocità di formazione del prodotto.
La misurazione verrà ripetuta almeno in doppio per ciascun campione
I valori di attività ottenuti devono essere utilizzati per calcolare l’attività totale,
dopo aver calcolato il valore medio delle prove effettuate su ciascun campione. Inoltre
il valore dell’attività totale si intende per il volume totale riportato in tabella, mentre
il saggio è effettuato su 20 l, quindi moltiplicate opportunamente.
Compilazione di una tabella di purificazione e discussione risultati
Dopo avere ottenuto i valori di concentrazione di proteine e di attività della catecolo
1,2 diossigenasi in ciascun campione EG, NL e CAT, completare la tabella considerando
che la resa% è definita come il rapporto tra la attività totale di ciscun passaggio
successivo e quella iniziale moltiplicata per 100, mentre il fattore di purificazione è
definito dal rapporto tra l’attività specifica del passaggio considerato e quella iniziale.
Valutate i vostri risultati considerando che l’ideale per una buona purificazione è
avere in pochi passaggi un valore il più alto possibile sia per il fattore di purificazione
che per la resa percentuale (perchè? quali sono i limiti? cosa è necessario migliorare?)
Per verificare la purezza della proteina di interesse si effettua una elettroforesi in
gel SDS-PAGE: per motivi di tempo verrà effettuata dagli esercitatori. Copia
dell’immagine del gel ottenuto verranno forniti a ciascun gruppo. I risultati dell’SDSPAGE sul campione CAT insieme ad una corsa di proteine a peso molecolare standard
dovranno essere utilizzati per calcolare Rf ed estrapolare il peso molecolare della
proteina di fusione purificata come illustrato a lezione (vanno inclusi e discussi nella
relazione).
OK, ora siete (quasi) dei PURIFICATORI DI PROTEINE! (NB: pare che la richiesta
sul mercato sia discreta...buona fortuna!)
Esercitazioni di chimica biologica
GUIDA ALLA STESURA DELLA RELAZIONE DI LABORATORIO
Indicazioni:
testo max 1500 parole (indicativamente esporre lo scopo dell’esperimento, le
metodiche e strategie adottate con la motivazione al loro utilizzo, risultati
sperimentali (positivi e negativi) e loro discussione, eventuali conclusioni e commenti)
DEVE EMERGERE IN BREVE LA VOSTRA COMPETENZA TEORICA SULLE
TECNICHE UTILIZZATE E LA COMPRENSIONE DELLA LORO APPLICAZIONE
SPERIMENTALE
inserire:
• Cromatogramma
• Tabella di purificazione (tabella B del protocollo) con tutti i dati numerici e
conversione dei valori di attività da ∆Abs/min in ∆Conc./min (ε260 per l’acido cis,
cis muconico = 16000 M-1cm-1
• Retta di taratura utilizzata per il dosaggio di proteine (con valori di a, b, r)
• Valori di concentrazione proteica ricavati con valore medio e deviazione
standard
• Immagine del gel SDS-PAGE (commento al risultato ottenuto per il proprio
campione)
• Retta di correlazione tra log. MW e Rf per il calcolo del peso molecolare della
proteina di fusione estrapolato dall’SDS-PAGE (se nel vostro campione non si
vede alcuna banda calcolate l’Rf di una banda visualizzata in qualsiasi altro
gruppo)
• Calcolo del peso molecolare della proteina di fusione purificata e dell’enzima
catecolo1,2 diossigenasi soltanto sapendo che la proteina tag GST pesa 29
kDaltons.