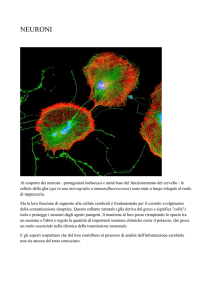
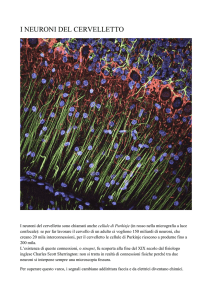
STIMA A PARTIRE DA DATI SPERIMENTALI DELLA
CONNETTIVITÀ IN RETI DI NEURONI DI TAGLIA ELEVATA
Federico Bizzarri(*), Marco Storace(*), Daniele Stellardo(*), Oscar De Feo(**)
(*) Dip. di Ingegneria Biofisica ed Elettronica, Università degli Studi di Genova
Via Opera Pia, 11A, I-16145, Genova
(**) Solianis Monitoring AG, Leutschenbachstrasse 46, CH-8050, Zurich, Switzerland
L’attività di ricerca, i cui risultati preliminari sono riportati in [1], è stata condotta
nell’ambito del Progetto di Ricerca PRIN2006 "Approssimazione di reti di sistemi
dinamici non lineari (modelli di neuroni biologicamente plausibili) e realizzazione di
circuiti a struttura parallela per la loro emulazione".
La caratterizzazione, a partire da dati sperimentali, dell’interazione tra diversi sistemi
dinamici che costituiscono una rete è un argomento di ricerca rilevante in molti settori
della moderna scienza applicata. In particolare, a partire da dati che riguardano
l’attività di una rete di neuroni, in questo lavoro ci siamo concentrati sulla stima di
come tali neuroni interagiscono tra loro tramite collegamenti sinaptici. L’idea di
partenza è che i dati sperimentali possano essere utilizzati come ingresso per una
tecnica di identificazione relativamente semplice (ispirata a quella proposta in [2]),
che sia in grado di stimare i parametri che caratterizzano una rete di neuroni di tipo
Leak Integrate and Fire (LIF) [3]. Lo scopo principale è quello di identificare la
"connettività reale", cioè il modello più semplice che prenda in considerazione ogni
interazione osservabile, diretta e non, tra i neuroni e che sia in grado di riprodurre
quasi integralmente le stesse relazioni temporali, osservate sperimentalmente, tra
agglomerati di cellule neuronali. Il metodo sviluppato si basa sulla conoscenza degli
istanti di tempo in cui si manifestano gli "spari" dei singoli neuroni, ossia si fa
riferimento a un processo puntuale. Rispetto al metodo proposto in [3], la sostanziale
differenza è l’assunzione che la costante di tempo caratteristica dei neuroni LIF, che
costituiscono la rete da identificare, sia nota e identica per tutti i neuroni.
Quest’ipotesi è piuttosto ragionevole (l’influenza di tale costante di tempo sul
comportamento globale della rete è trascurabile) e permette di identificare i parametri
della rete per mezzo di una semplice regressione lineare, facendo sì che la tecnica sia
applicabile a reti con centinaia di elementi, poiché la complessità del metodo scala
linearmente con il numero dei neuroni. Dal momento che la rete da identificare è
costituita da neuroni di tipo LIF, il metodo è piuttosto affidabile quando l’attività di
ciascun neurone che appartiene alla rete originale è piuttosto regolare (regular
spiking), mentre i risultati sono meno accurati quando gli intervalli tra spari successivi
(Inter-Spike Intervals - ISI) non sono regolari (bursting o spiking non periodico).
Per valutare le prestazioni del metodo proposto, sono stati considerati dati sintetici
ottenuti simulando sia reti di neuroni di tipo LIF sia reti di neuroni descritti dal
modello di Izhikevich [4]. In particolare, per ciascuno dei due diversi tipi di neuroni,
sono state considerate 100 reti composte da 2p elementi (p = 1,…7), connessi fra loro
con una topologia di tipo "one to all", i cui pesi sinaptici sono stati descritti da una
variabile aleatoria con distribuzione Gaussiana a media nulla e deviazione standard
pari a 10 (maggiori dettagli sulla struttura delle reti utilizzate per generare i dati
sintetici impiegati dal regressore sono disponibili in [1]). Le uscite di ciascuna rete
originale (cioè gli istanti di sparo dei suoi neuroni) sono state poi usate per la
procedura di regressione ottenendo così, per ciascuna rete identificata, un’opportuna
matrice di connettività e le correnti di polarizzazione dei neuroni LIF. Per valutare la
qualità dell’identificazione sono state prese in considerazione le statistiche del primo
ordine degli ISI dei primi due neuroni della rete originale e di quella identificata. In
Figura 1 sono riportati alcuni risultati che consentono di valutare le prestazioni del
metodo sviluppato: i riquadri di sinistra e quelli di destra si riferiscono,
rispettivamente, al primo e al secondo neurone delle diverse reti prese in
considerazione. Inoltre, i riquadri in alto si riferiscono a reti originali costituite da
neuroni di tipo LIF mentre quelli in basso a neuroni descritti dal modello di
Izhikevich. In tutti i riquadri le curve raffigurate rappresentano il valor medio degli
ISI per diversi valori di p: le curve in nero si riferiscono ai modelli originali e quelle
in grigio al modello di rete LIF identificata. Il confronto evidenzia come, in media, le
reti identificate siano in grado di riprodurre piuttosto fedelmente l’andamento medio
degli ISI delle reti originali. Al fine di poter modellare più fedelmente la varianza
degli ISI, sarà necessario aggiungere al modello opportune sorgenti di rumore che
perturbino la dinamica delle reti identificate: in caso contrario, tale dinamica non può
che essere regolare, essendo le reti costituite da neuroni di tipo LIF.
Figura 1.
Riferimenti bibliografici
[1] F. Bizzarri, M. Storace, D. Stellardo, O. De Feo, "On the problem of estimating
connectivity from spike recordings in large neuron networks", in Proc. of NOLTA
2008, Budapest, Hungary, 7-10 Settembre 2008, pp. 86-89 (invited paper,
sessione speciale "Applications of Complex Networks I: Constructing Networks
from Observed Data").
[2] R. B. Stein, "Some Models of Neuronal Variability", Biophys J., vol. 7, no. 1, pp
37-68, 1967.
[3] V.A. Makarov, F. Panetsos, O. De Feo, "A method for determining neural
connectivity and inferring the underlying network dynamics using extracellular
spike recordings," Journal of Neuroscience Methods, vol. 144, pp. 265–279, 2005.
[4] E.M. Izhikevich, "Simple Model of Spiking Neurons", IEEE Trans. Neur. Net.,
Vol. 14, No. 6, pp. 1569-1572, 2003.