Modelli di regolazione semplici
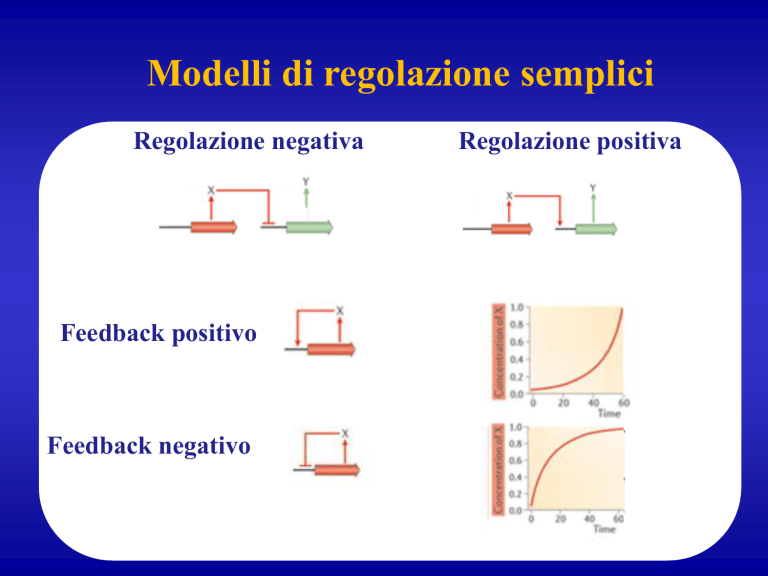
Regolazione negativa
Feedback positivo
Feedback negativo
Regolazione positiva
Circuiti genetici oscillanti
Circuiti genetici complessi
Aumento del numero
dei componenti
B
A
H
D
Interconnessione
tra circuiti
G
E
F
C
Caulobacter crescentus
C. crescentus presenta tre forme cellulari distinte
durante il suo normale ciclo cellulare:
Cellule motili e flagellate, swarmer
Cellule con stelo, stalk, sessili
Cellule predivisionali, con flagello e stalk
Il processo cellulare di differenziamento risponde alla
modulazione temporale di tre regolatori principali:
CtrA, DnaA, GcrA
Questi sono organizzati
in un circuito di
regolazione complesso
Il processo cellulare di differenziamento risponde alla
modulazione temporale di tre regolatori principali:
DnaA, GcrA, CtrA
CtrA
E’ di un regolatore trascrizionale che appartiene alla famiglia dei
“response regulator” fosforilati da uno o più “sensori” (PleD,
DivK e CckA). É il regolatore della forma flagellata ….
Questa proteina quando è presente e fosforilata è in grado di
regolare l’espressione di 95 geni determinando :
• L’inibizione della replicazione del DNA legandosi alla regione
OriC, il blocco della divisione cellulare attraverso l’inibizione
trascrizionale del gene ftsZ, l ‘induzione della biosintesi del
flagello.
•Inoltre CtrA è in grado di attivare il gene ccrM, reprimere gcrA
ed esercitare un meccanismo di feedback negativo sul suo
promotore P1 e positivo sul promotore P2 quando il DNA è
metilato.
La sua fosforilazione è determinata da numerosi
sensori. Tra questi quello più importante è CckA.
GcrA
•È il regolatore di riferimento per la cellula sessile
•È indotta trascrizionalmente da DnaA
•Induce l’espressione dei geni per la replicazione del DNA
•Induce l’espressione di ctrA sul P1 con DNA emimetilato
•Subisce la repressione trascrizionale da parte di CtrA
•Induce l’espressione degli elementi responsabili del
differenziamento.
DnaA
• È la proteina responsabile dell’inizio
della replicazione nei cromosomi
procariotici, controlla anche
l’espressione di una ventina di geni.
• È l’induttore trascrizionale di gcrA
• Il promotore del gene dnaA è
scarsamente funzionale quando il DNA è
emimetilato, mentre trascrive ad elevati
livelli quando il DNA è pienamente
metilato.
•Il gene dnaA mostra un meccanismo di
feedback negativo.
CcrM
• È Una metilasi caratteristica di Caulobacter e
corrisponde funzinalmente alla metilasi dam di E. coli.
• E’ responsabile della metilazione delle sequenze
GANTC nel DNA di Caulobacter.
• Modula l’attività del promotore del gene dnaA e quella
dei due promotori a monte del gene ctrA (P1 e P2).
• Mostra un meccanismo di regolazione di tipo “feedback
negativo”.
Cellula flagellata
Nella cellula flagellata, a causa del
predominio di CtrA abbiamo:
• Assenza di GcrA
• DNA pienamente metilato (CcrM)
• Elevati livelli di DnaA (che però non
determina la replicazione del DNA in
quanto questa è repressa da CtrA)
Ne consegue che la cellula flagellata non
è in grado di replicare ne di dividersi
Passaggio alla fase sessile
Durante questa fase i pili vengono riassorbiti dalla cellula
mentre il flagello viene rilasciato nell’ambiente. Nel medesimo
polo si ha la formazione del peduncolo.
Nel passaggio da cellula motile a quella sessile la proteina CtrA
viene totalmente degradata nel citoplasma per l'azione dalla
proteasi ClpX/P indotta dalla fosforilazione del regolatore DivK.
In mancanza di CtrA si ha:
• l’espressione di GcrA il cui gene non è più represso da CtrA
ma è indotto da DnaA (sintesi del peduncolo e eliminazione
del flagello)
• si libera OriC che ora è in grado di interagire con DnaA e
iniziare la replicazione del DNA
• si deregola l’espressione di ftsZ e si assiste alla formazione del
setto, inizia la divisione cellulare.
Cellula predivisionale
Avvenuta la replicazione del DNA, GcrA induce
l’espressione di CtrA a bassi livelli tramite attivazione
del P1 con DNA emimetilato.
I bassi livelli di CtrA vengono immediatamente
fosforilati da CckA e CtrA-P induce l’espressione di
CcrM. Questa metila il DNA favorendo il feedback
positivo di CtrA-P sul suo promotore P2, aumenta
notevolmente l’espressione di CtrA istaurando le
condizioni tipiche della cellula flagellata tra cui:
- la trascrizione dei geni per la biosintesi del flagello
- quella dei geni ftsQ ed ftsA necessari per portare a
termine la la divisione cellulare.
Divisione cellulare
Alla fine del ciclo, durante la divisione
cellulare, la proteina CtrA viene
degradata attivamente e selettivamente
solo nel compartimento della cellula
sessile che, quindi, inizia una nuova
divisione cellulare.
La proteina GcrA, che ha un’emivita molto
breve, nella cellula sessile è sostenuta da
un’attiva sintesi, mentre in quella flagellata
si degrada velocemente e non viene espressa
in quanto il gene represso da CtrA
RIASSUMENDO…..
Localizzazione delle proteine PleC/D, DivJ/K
Regolazione polare del sistema Div/Ple
Synthetic Biology: Genetic Circuit Design
Sulla base dello studio dei sistemi complessi di regolazione (come quelli
di Caulobacter) e disponendo delle tecniche e delle conoscenze
necessarie alla fine degli anni ’90, Gardner et al. (2000) e Elowitz and
Leibler (2000) realizzarono i primi semplici circuiti trascrizionali, un
interruttore e un oscillatore.
Toggles and oscillators: new genetic circuit designs
Ellen M. Judd,1,2 Michael T. Laub,2 and Harley H. McAdams2*
Oggi si è sviluppata una vera e propria disciplina che permette di
teorizzare e realizzare circuiti genetici. Questa disciplina ha
mutuato il linguaggio dei circuiti digitali.
Si fa riferimento in particolare ai «logic gates», blocchi logici che
consistono solitamente in due input e un output. I più comuni
sono:
I blocchi logici possono essere realizzati con :
DNA binding proteins (LacI, AraC, CI, TetR, ZFP e TALEsTranscriptional Activator Like Effector)
Operone Arabinosio (AraC-PBAD)
Operone Lattosio (lacI-Plac)
NOR e AND con DNA binding protein:
Invertasi come Tyr recombinasi o Ser integrasi:
Inserimento di
un terminatore
Tyr Rec
Ser Inv
NOR e AND con Ser invertase:
CRISPR
(Clustered Regularly interspaced Short Palindromic Repeats)
Presenti nel 40% dei
batteri e nel 90% degli
archea, sono loci
organizzati da sequenze
ripetute brevi e dirette
intervallate a sequenze
“spacer”. In aggiunta si
possono identificare
geni, associati a queste
sequenze, detti cas
Sono assimilabili a una
sorta di sistema
immunitario della cellula
CRISPR
con Cas9 wt
con dCas9
e un sgRNA
Uso di Cas9 modificata (dCas9) in un NOR:
o ancora basati sull’espressione di sRNA regolativi. Questi
possono agire a livello di controllo traduzionale e, con le
opportune modifiche, trascrizionale (tna).
tnaC e tnaAB
rut
Circuito NOR basato su regolazione derivata da tnaA
UP-SCALE dei circuiti regolativi
Nielsen et al 2013












