Regolazione dell’Espressione Genica
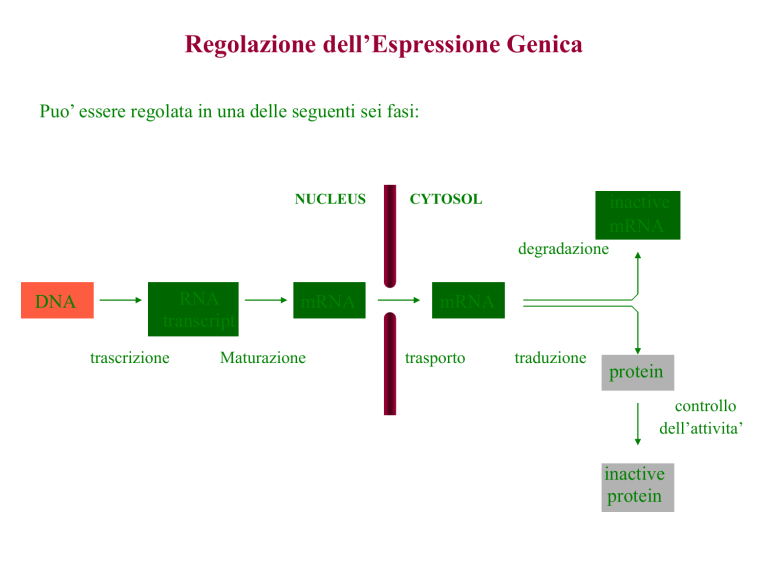
Puo’ essere regolata in una delle seguenti sei fasi:
NUCLEUS
CYTOSOL
inactive
mRNA
degradazione
DNA
RNA
transcript
trascrizione
mRNA
Maturazione
mRNA
trasporto
traduzione
protein
controllo
dell’attivita’
inactive
protein
Metodi per studiare
l’espressione dei geni
• Northern blot
• Trascrittasi Inversa -PCR (RT-PCR)
• Ibridazione In situ
• Trascrizione In vitro
• DNA Microarray
Purificazione dell’RNA
• Procedura
– Lisi delle cellule con un reagente
che dissocia le nucleoproteine e
inibisce la RNAsi
– Rimozione delle proteine
– Precipitazione specifica dell’RNA
Northern blot
• Per determinare la dimensione e la
quantita’ di specifici mRNA
• Studi sull’espressione genica
Northern blot
– Elettroforesi dell’RNA in gel contenente
formaldeide per mantenere l’RNA in
forma completamente lineare
– Trasferimento dell’RNA su una
membrana
– Ibridazione con una sonda marcata
tampone elettroforetico
es. TAE (4mM Tris-acetato, 1mM EDTA, pH 8.0)
generatore di corrente
gel di agarosio
100V
-
+
0,2A
scatola elettroforetica
Gel ElettroforesiSepara le molecole in base alla dimensione
Trasferimento su supporto solido
Trasferimento dal gel alla
membrana
Dopo il trasferimento
gel
membrana
Preparare la sonda per il Northern Blot
Ibridazione
• La sonda marcata e’ aggiunta ad una
soluzione contenente il supporto solido che
lega l’RNA da analizzare
Lavaggio
• Rimozione della sonda non legata al
supporto solido
Rivelazione degli ibridi
• Esposizione di un film se la sonda e’
marcata radioattivamente
• Se la sonda e’ marcata con un enzima si
procede alla reazione enzimatica che
produce colore direttamente sul supporto
solido
Northern blot
• La rivelazione avviene usando:
– DNA marcato con radioattivo(32P)
– DNA marcato con un enzima che
catalizza una reazione che produce
luce o colore
Northern blot
Fig. 5. Northern blot analysis of E. lagascae total RNA from leaves (L), germinating
seeds (Se), roots (Ro) and stems (St). The blot was hybridized with probes for ElLTP1
(top panel) and ElLTP2 (middle panel). The bottom panel shows ethidium bromide
staining of the gel before blotting. The numbers to the left indicate approximate
transcript sizes in kb
Svantaggi del Northern Blot
• Richiede grandi quantita’ di RNA
• E’ un processo lungo e laborioso
AMPLIFICAZIONE IN
VITRO DEL DNA
“REAZIONE A CATENA
DELLA POLIMERASI
(PCR)”
PCR: reazione polimerasica a
catena
• Inventata da Kary Mullis negli anni ‘80 (premio Nobel
1993)
• Serve per ottenere una grande quantita’ di una specifica
sequenza di DNA in vitro
• Puo’ amplificare un tratto di DNA per piu’ di 1 milione di
volte
ELEMENTI NECESSARI ALLA REAZIONE:
1- DUE OLIGONUCLEOTIDI COMPLEMENTARI A
DUE REGIONI CHE SI TROVANO SU FILAMENTI
OPPOSTI DEL DNA STAMPO AI LATI DELLA
REGIONE CHE SI VUOLE AMPLIFICARE
2- DNA STAMPO CHE CONTENGA LA REGIONE
DA AMPLIFICARE
3- POLIMERASI TERMOSTABILE (NON VIENE
DENATURATA SE PORTATA A 95° C)
4- I 4 DESOSSINUCLEOTIDI TRIFOSFATI
IL PROCESSO DI PCR PREVEDE UN CERTO NUMERO
DI CICLI. OGNI CICLO CONSISTE DI 3 PASSAGGI:
1- DENATURAZIONE: TEMP. 95°C. IL DNA STAMPO
VIENE DENATURATO, I DUE FILAMENTI SI SEPARANO
2-APPAIAMENTO: 55°C CIRCA. I PRIMERS SI APPAIANO
CON IL DNA STAMPO
3- SINTESI: TEMP.72°C E’ OTTIMALE PER IL
FUNZIONAMENTO DELLA Taq (Termus aquaticus)
POLIMERASI
PCR: Reazione a catena della polimerasi
Metodo di amplificazione del DNA usando una polimerasi
termostabile quale la Taq DNA polimerasi, uno stampo di DNA, un
eccesso di primers e dideossinucleotidi (dNTP) in un buffer.
PCR: Reazione a catena della polimerasi
PCR: Reazione a catena della polimerasi
PCR: amplificazione
n.ro cicli
n.ro sequenze bersaglio
1
0
3
2
10
256
15
8192
20
262.144
25
8.388.608
30
268.435.456
La sequenza bersaglio è la
sequenza di DNA sintetizzata
tra i due primers
Occorre ~1g di DNA per l’analisi di una sequenza o una digestione con enzimi di restrizione tali da poter
essere visibili in elettroforesi. Se vi sono ~5 pg di DNA/cellula umana (5x10-12g) allora ~1 g of DNA
potrebbe essere isolato da 200.000 cellule ma avremmo un miscuglio di tutti i geni.
In 1 g di DNA genomico, una copia singola di un gene (300 bp) equivarrebbe a ~0.1 pg di DNA.
Questo 0.1 pg di DNA potrebbe essere amplificato mediante PCR producendo 0.8 g in 25 cicli e 27 g in
30 cicli.
PCR
VANTAGGI:
• Sensibilita’
• Rapidita’
• Si presta all’analisi simultanea di molti campioni (high throughput)
• Si presta all’analisi simultanea di diverse sequenze sullo stesso
campione
• Si presta all’analisi di DNA degradato o incluso in mezzi strani, o
fissato
• SVANTAGGI:
• Sensibilita’ (rischio di contaminazioni-falsi positivi)
• Variabile efficienza di amplificazione a seconda della sequenza
• Richiede conoscenza di base delle sequenze da amplificare e messa a
punto per coppie di oligonucleotidi di innesco (primers)
• Può sintetizzare frammenti relativamente corti
• La sintesi è imprecisa e introduce errori nella sequenza (la Taq pol non
possiede attività 3’->5’ esonucleasica)
Esempi di utilizzo della PCR
• Su DNA:
– segnalare la presenza o meno di sequenze specifiche (mutazioni,
inserzioni virali, micro-organismo patogeni) -> PCR DIAGNOSTICA
– Amplificare frammenti specifici da usare in seguito come sonde
oppure da “clonare”
• Su RNA messaggero (RT-PCR):
– segnalare la presenza di specifiche molecole di
RNA (espressione genica, presenza di RNA di
micro-organismi infettivi)
– Amplificare frammenti specifici da usare in
Trascrittasi Inversa- PCR
• Rivelazione di mRNA molto rari
• Analisi di RNA con pochissime
quantita’
Procedura
• Purificazione dell’RNA
• Aggiungere i primer
• mescolare RNA con la trascrittasi inversa
per effettuare la sintesi del primo
filamento di cDNA
• Aggiungere la DNA polimerasi
termostabile per effettuare la sintesi del
secondo filamento di cDNA e per
amplificarlo con i cicli della PCR
Trascrittasi inversa - PCR (RT-PCR)
mRNA 5’
Primer 1
3’
AAAAA 3’
TTTTT 5’
reverse transcriptase
(RNA-dependent DNA polymerase)
AAAAA 3’
TTTTT 5’
mRNA 5’
cDNA 3’
Remove RNA
(RNase A)
cDNA 3’
TTTTT 5´
Add PCR primers
Primer 2 5’
3’
TTTTT
3’
5’
Primer 3
Add Taq polymerase. Run PCR
Number of molecules
Selezione del numero di cicli
necessari per trovarsi nella fase di
amplificazione esponenziale
Number of cycles
RT-PCR in tempo reale
• Utilizza particolari combinazioni di
coloranti fluorescenti la cui fluorescenza
viene smascherata quando la catena di DNA
viene sintetizzata oppure che vengono
intercalati nella catena nascente durante la
reazione di amplificazione.
• L’utilizzo di appositi apparecchi permette la
misurazione della fluorescenza accumulata
Polymerase Chain Reaction: resa
Log[DNA]
• Resa teorica: 2n
P=(2)n T Il prodotto (P) incrementa esponenzialmente
con il numero di cicli di PCR (n)
Il prodotto di PCR dipende da T,numero di
copie di template di partenza
Plateau
Lineare
Esponenziale
Prodotto
variabile
N° cicli
termici
Perché Real-Time?
Misura l'amplificazione in tempo reale
durante la fase esponenziale della PCR,
quando cioè l'efficienza di amplificazione è
influenzata minimamente dalle variabili di
reazione, permettendo di ottenere risultati
molto più accurati rispetto alla PCR
tradizionale "end point"
RT-PCR quantitativa
•Rilevamento della fluorescenza associata all’amplificazione
•Il prodotto di PCR non viene analizzato su gel di agarosio
•Analisi del prodotto di fluorescenza tramite computer
Plot lineare
Incremento di
fluorescenza
Cicli di PCR
Analisi tramite software
Chimiche fluorescenti
per PCR Real-Time
•La fluorescenza si genera durante la PCR per
effetto di diverse possibili reazioni chimiche
•Le chimiche principali sono basate sia sul
legame di coloranti fluorescenti che si intercalano
nella doppia elica di DNA, come il SYBR Green,
sia sull'ibridazione di sonde specifiche.
SYBR Green:SYBR
principio
Green I
Utilizza una molecola fluorescente non specifica che si
lega al solco minore del DNA
SYBR Green
All’inizio del processo di amplificazione, la miscela di
reazione contiene DNA denaturato, primers e la
molecola fluorescente
SYBR Green
Dopo l’annealing dei primers, si
legano poche molecole
fluorescenti alla doppia elica.
SYBR Green
Durante l’elongazione si verifica un
aumento di fluorescenza che corrisponde
all’ aumento del numero di copie
dell’amplicone
Reverse transcriptase-PCR (= RT-PCR)
Epoxide hydrolase
+ + + + - - - l
2027
1904
1584
947
831
564
l se r st l se r st pl
RT
Ibridazione dell’RNA in situ
Rivelazione di mRNA direttamente su tessuti messi su
vetrini da microscopio
Preparation of RNA probe with in vitro transcription
T7/T3 or SP6
promoter
RNA in situ hybridization
Colour substrate:
nitro blue tetrazolium (NBT)
+ 5-bromo-4-chloro-3indolyl phosphate (BCIP)
Fig. 7. RNA in situ hybridization with antisense (a-c) and sense (d) RNA probes for ElLTP1.
E. lagascae seedlings were collected 7 days after sowing. co Cotyledon, en endosperm,
hy hypocotyl, am apical meristem, ro root
• Il livello di RNA presente nella cellula non
necessariamente riflette direttamente i livelli di
trascrizione del gene
• Per poter asserire che un gene viene attivato
TRASCRIZIONALMENTE occorre poterne
visualizzare l’attivita’
Il saggio di “run-on” permette di
determinare il livello di
trascrizione di un gene
1)
2)
3)
4)
5)
Purificare i nuclei
+ NTP, 32P GTP
Trascrizione: la catena nascente
e’ radioattiva
Estrazione dell’RNA
Ibridazione a cDNA immobilizzato
su filtro
Sintesi differenziale di 12 geni codificanti mRNA
epato-specifici analizzata tramite run-on
Lodish
Figure 10-23
Northern blotting
RNA isolation
Probe labeling
AAAAAA
dCTP
dGTP
dTTP
dATP
pBS-SemaIII
Gel electophoresis
Hybridization
Blotting
Autoradiography
reverse Northern blotting
Northern
reverse Northern
specific probes
*
*
labeled target
cDNA arrays (continued)
• macro-arrays
– low-density
– filter-supported
– radioactive probes
(duplicates)
– low sensitivity
– only cDNA clones
spotted
– cheap
cDNA arrays (continued)
• micro-arrays
– high-density
– glass-supported
– fluorescent probes
(single slide)
– high sensitivity
– ability to spot
oligonucleotides
– very expensive
Tessuto della prostata,
normale
Tessuto della prostata,
tumorale
In questo esempio i cDNA
sono stati posizionati su di un
vetrino, simile ai normali
vetrini usati per l’istologia.
I microarrays
Un microarray è un supporto solido
sul quale sono stati posizionati
diverse migliaia di cDNA in spot
separati. Ciascuno spot rappresenta
un gene, in quanto contiene
numerose copie di un cDNA
corrispondente a tale gene.
Microarray technology
Probes
Microarray synthetized by photolithography or EST/oligo “linking”
http://www.ym.edu.tw/excellence/HBP/HBP_CP4/procedure.htm
Tessuto della prostata,
normale
Tessuto della prostata,
tumorale
utilizzo dei microarrays
si confrontano i profili di
espressione genica di due campioni
differenti.
E’ necessario estrarre le molecole di
mRNA dai due campioni.
Arrays
Probe DNAs are “spotted”
onto a glass slide
Each spot corresponds to a
particular gene e.g. β-actin
Each array will have
thousands of spots (typically
3000 to 30000)
reference
test
RNA is extracted from tissues
Targets
mRNA
cDNA
or cells
RNA is copied to DNA
labelled
DNA is fluorescently labelled
pooled
Samples are pooled
Hybridisation
Labelled target DNA
hybridised to array
Fluorescence of each spot
indicates how much of
particular RNA was present in
both samples
Hybridised
Array
Data Processing
Further
analysis
Scanner
Images
Normalised
Ratios
Normalisation
Spot-finding
Ratios
Cy3
Cy5
test
reference
Cy3
Cy5
Spot-finding (1)
Software identifies grid of
spots and maps individual spot
locations
Estrazione
dell’mRNA dai
2 campioni di
cellule che si
vogliono
confrontare
Conversione in
cDNA
Marcatura con
2 fluorocromi
diversi
Confronto dei profili di espressione genica in
due campioni cellulari diversi
(1)
(2)
(3)
(6)
Immagine a colori
raffigurante il
microarray
Eccitazione della
fluorescenza
tramite laser
Riconoscimento (4)
tra i cDNA
provenienti dai 2 campioni e
quelli già presenti sul microarray
(5)
STUDIO DEI PROMOTORI: siti di legame dei fattori
di trascrizione
EMSA
Trans Factor Methods: EMSA
Trans Factor Methods: EMSA
Electromobility
“Shift”
Trans Factor Methods: Consensus Sequence Capture
Il saggio EMSA identifica
interazioni tra DNA e proteine
anticorpo
Il saggio EMSA identifica
interazioni tra DNA e proteine
• Competizione con stesso sito, sito analogo, sito mutato
• Identificazione dei fattori coinvolti tramite “supershift” con anticorpi
Lodish
Figure 10-7
di localizzare il sito di interazione
tra proteina e DNA
Lodish
Figure 10-6












