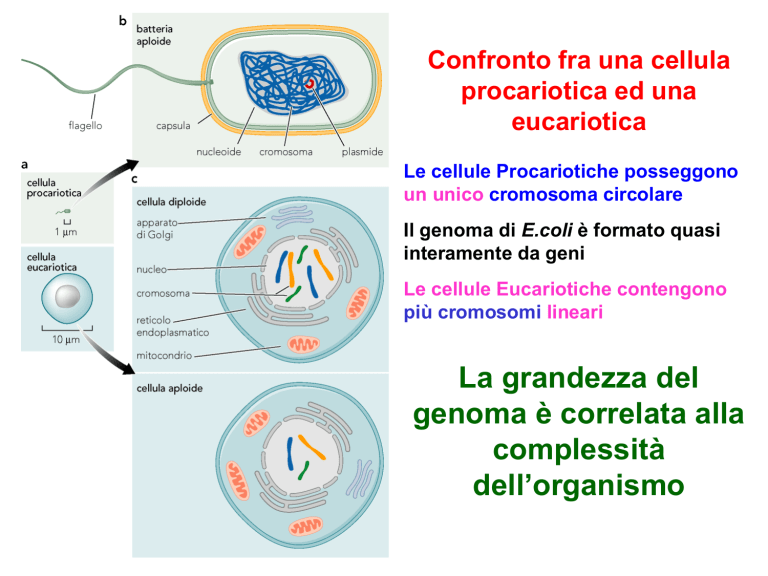
Confronto fra una cellula
procariotica ed una
eucariotica
Le cellule Procariotiche posseggono
un unico cromosoma circolare
Il genoma di E.coli è formato quasi
interamente da geni
Le cellule Eucariotiche contengono
più cromosomi lineari
La grandezza del
genoma è correlata alla
complessità
dell’organismo
EUCARIOTI
PROCARIOTI
Comparazione della densità genica in cromosomi di differenti organismi
La grandezza del genoma varia nei differenti organismi
Sono necessari un numero molto elevato di geni per dirigere la
formazione di organismi sempre più complessi
MA
Il numero dei geni codificati all’interno di un tratto di DNA della stessa
lunghezza diminuisce con l’aumentare della complessità dell’organismo
DENSITÀ GENICA = QUANTITÀ DI GENI CONTENUTI IN UNA MEGABASE
DI DNA GENOMICO
I virus utilizzano anche entrambi i filamenti per codificare geni che a volte
si sovrappongono
I GENI RAPPRESENTANO UNA PICCOLA PORZIONE DEL DNA
CROMOSOMICO EUCARIOTICO
2 FATTORI :
AUMENTO DELLE DIMENSIONI DEI GENI
Sequenze regolative
Introni
Sequenze regolative = regioni di DNA utilizzate per dirigere e regolare la
trascrizione
Introni = regioni non codificanti intersperse all’interno delle sequenze
codificanti
AUMENTO DELLA QUANTITÀ DI DNA TRA I GENI
sequenze intergeniche
Pseudogeni
Sequenze altamente ripetute e DNA microsatellitare
MECCANISMO DELLO SPLICING DELL’RNA
Solotanto il 5% del DNA di un gene rappresenta la parte codificante della proteina
I procarioti non hanno introni
Gli eucarioti più semplici sono caratterizzati da una bassa quantità di introni
Gli pseudogeni provengono dall’integrazione di prodotti
retrotrascritti da mRNA
Trascrittasi inversa
Enzima virale che copia RNA
in DNA (cDNA)
Dopo infezione virale il cDNA
può essere integrato nel
genoma della cellula ospite
Queste copie non possono
essere espresse in quanto
mancano delle sequenze
regolative
CI VOGLIONO GRANDI QUANTITÀ DI DNA PER CODIFICARE TUTTA
L’INFORMAZIONE
IL DNA DEVE ESSERE COMPATTATO
Ogni cellula dell’uomo contiene
circa 2 metri di DNA
il nucleo
misura circa 5-8
40 Km di filo in una palla da tennis
Proteine specializzate che
imballano il DNA ripiegandolo in
una serie di anse e avvolgimenti
che lo dispongono in una struttura
ordinata
I CROMOSOMI
Packaging of DNA into the Nucleus
Il DNA eucariotico è impacchettato in cromosomi
Ogni cromosoma è costituito da un’unica lunga molecola di DNA lineare
associata a proteine
Il complesso DNA- proteine prende il nome di CROMATINA (khroma =colore)
L’organizzazione dei cromosomi
In una cellula eucariotica, quasi tutto il DNA è compattato
nella cromatina, sia per consentire un ripiegamento
organizzato della lunga molecola del DNA sia per consentire
alla cellula di controllare l’espressione dei geni.
Si possono distinguere due tipi di cromatina:
Eucromatina, che consiste di DNA che viene espresso
Eterocromatina, che consiste di regioni di DNA per la maggior
parte trascrizionalmente inattive. Sembra che svolga ruoli
strutturali.
Ci sono tre livelli di organizzazione
della cromatina:
1) nucleosoma – (dà alla cromatina un
aspetto di collana di perle)
2) fibra di cromatina da 30 nm
3) cromosoma
Struttura dei cromosomi eucariotici: Nucleosomi
Un nucleosoma è l’unità di compattamento fondamentale della
cromatina ed è costituito da DNA e proteine istoniche. E’
presente nel nucleo cellulare degli eucarioti.
I nucleosomi visti al microscopio elettronico e danno alla
cromatina un aspetto di collana di perle.
COLLANA DI PERLE
Il nucleosoma è composto di un CORE di otto proteine
istoniche intorno al quale si avvolge il DNA
Il DNA che prende direttamente contatto con le proteine è definito DNA core
(perle)
Il DNA fra ciascun nucleosoma è definito DNA linker
(il filo della collana)
DNA core circa 147 bp (invariabile)
DNA linker
20-60 bp (variabile)
LE PROTEINE ISTONICHE
Le proteine della cromatina sono costituita per l’80-90 % da ISTONI.
Gli istoni nella struttura del core assumono una struttura discoidale che si
assembla soltanto in presenza di DNA
Gli istoni sono piccole
proteine basiche che
legano il DNA.
Hanno una proporzione
molto alta di a.a.carichi
positivamente (lisina e
arginina) e sono molto
conservate.
UNA STRUTTURA
CROMATINICA
CONTENENTE ISTONI, È
UNA CARATTERISTICA
UNICA DEGLI EUCARIOTI
LE PROTEINE ISTONICHE
Histone-fold
regione altamente conservata formata da
tre regioni alpha-elica separate da due
tratti a struttura non definita
Coda
Estensione all’N Terminale della proteina
regione a struttura non definita che
sporge all’esterno del nucleosoma
Le code N-terminali non sono richieste
per l’associazione con il DNA
Trattamento con tripsina idrolizza le code
lasciando inalterata la struttura del core
Sono sito di frequenti modificazioni
(fosforilazione, acetilazione e
metilazione) che possono influire sul
ruolo funzionale del nucleosoma
PROTEINE CROMOSOMICHE NON ISTONICHE.
Le proteine NON-ISTONICHE sono tutte le altre proteine associate al DNA,
Al contrario degli istoni, le proteine non-istoniche differiscono
notevolmente in numero e tipo, tra un tipo cellulare e un altro entro un
organismo, in momenti diversi nello stesso tipo cellulare e tra organismi
diversi.
Queste includono un’ampia varietà di proteine, come polimerasi e altri
enzimi nucleari, recettori degli ormoni e varie proteine regolatrici. E’
possibile osservare, su gel bidimensionali, circa 1000 differenti proteine
non istoniche in una tipica cellula eucariotica.
IL NUCLEOSOMA
I NUCLEOSOMI SONO I MATTONI DEL CROMOSOMA
UNITA’ BASE DELLA STRUTTURA CROMATINICA
Mediante assemblaggio in nucleosomi il DNA viene compattato di 6 volte
(primo stadio di compattamento)
Il nucleosoma consiste di due
giri interi di DNA (~ 80 coppie
di nucleotidi per giro) avvolti
intorno al nucleo ottamerico di
proteine, più il DNA di unione
adiacente
La microscopia elettronica dimostra
che il complesso DNA-proteine appare
sotto forma di fibra dal diametro di 10
nm.
La parte proteica del
nucleosoma è costituita da due
molecole per ognuno dei
quattro istoni, H2A, H2B, H3
and H4 (ottamero di istoni).
La fibra consiste in una stringa
ininterrotta di nucleosomi.
L’istone H1 lega il DNA linker
ASSEMBLAGGIO DEL NUCLEOSOMA
L’assemblaggio del nucleosoma consiste
nella associazione ordinata dei complessi
proteici istonici con il DNA.
Inizia con la formazione del tetramero
2xH4+2xH3
Il tetramero si lega al DNA ds
tetramero + DNA ds reclutano due dimeri di
H2A+H2B
INTERAZIONE TRA ISTONI E DNA E’
INDIPENDENTE DALLA SEQUENZA
L’interazione tra DNA e core istonico è mediata da circa 140 legami a
idrogeno (una tipica interazione DNA-proteina ne coinvolge circa 20).
Quasi tutti i legami si istaurano con gli atomi di ossigeno dei legami
fosfodiesterici vicini al solco minore,
solo 7 legami vengono formati tra le proteine e le basi attraverso il
solco maggiore (assenza di specificità).
La natura basica degli istoni maschera le cariche negative dei fosfati,
permettendone un avvicinamento fisico, determinato dalla curvatura
del DNA, altrimenti impossibile.
Tra gli istoni e lo
scheletro zuccherofosfato del DNA si
instaurano interazioni
ioniche o legami idrogeno
STRUTTURE DI ORDINE SUPERIORE
L’istone H1 interagisce con il DNA linker producendo una
maggiore adesione del DNA all’ottamero istonico
(ulteriore protezione di 20 bp all’azione della nucleasi),
ma lega anche una regione mediana dei 147 bp associati
all’ottamero istonico.
Il legame di H1 aumenta il compattamento
del DNA sul nucleosoma,
-H1
+H1
LA FIBRA DA 30 nm
L’ISTONE H1 MEDIA LA FORMAZIONE DELLA FIBRA DA 30 NM
Nella cellula vivente, la cromatina si trova in uno stato più altamente
condensato: la fibra cromatinica da 30 nm. Questa fibra mostra una
struttura spiralizzata con circa 6 nucleosomi per ogni giro. Ciò
corrisponde a un quoziente di compattamento di ~ 40. La formazione di
questo tipo di fibra richiede la presenza dell’istone H1.
2 modelli alternativi
i) modello a soleneoide
Una superelica di
6nucleosomi x giro
ii) modello a zig-zag
Nucleosomi disposti a zig
zag (DNA linker + lungo)
LE CODE ISTONICHE CONTRIBUISCONO ALLA FORMAZIONE DELLA
FIBRA DA 30 NM
Le code N-terminali degli istoni contribuiscono alla formazione della fibra
da 30 nm mediando interazioni (legami a idrogeno) tra nucleosomi
adiacenti. Istoni del core privi di code non sono in grado di formare fibre
da 30 nm.
STRUTTURE DI ORDINE SUPERIORE DELLA CROMATINA
L’impaccamento della cromatina
Nucleosomi (6 volte; 1 m
16 cm)
fibre da 30 nm (40 volte; 1 m
2.5 cm)
ancora molto lontano dall’ impaccamento necessario
(pari a 10.000-100.000 volte) per contenere il DNA nel
nucleo.
organizzazione del cromosoma eucariotico:
la compattezza del DNA del cromosoma
eucariotico è dovuta ad avvolgimenti
successivi che si sovrappongono ad
avvolgimenti già presenti
Il modello strutturale più
accreditato realizza questo
compattamento attraverso la
formazione di anse (40-90 kbp)
bloccate alla base da una
struttura proteica
Scaffold nucleare.
topoisomerasi II proteine SMC
(structural maintenance of
chromosome) sono
componenti essenziali dello
scaffold nucleare
Distesa in tutta la sua lunghezza la molecola raggiunge la dimensione di 1mm,
cioè circa mille volte la lunghezza del batterio.
Il Genoma procariotico anche se non è organizzato in
cromosomi, mostra strutture compatte che occupano circa 1/3
del volume della cellula
NUCLEOIDE
Tutti i batteri posseggono una regione,
relativamente trasparente, in cui è
addensato il materiale cromosomale,
denominato
“nucleoide”
(cromosoma
batterico o corpo cromatico).
Nucleoide
Il nucleoide è la struttura in cui è
localizzata l’informazione genetica delle
cellule procariotiche
È costituito da un’unica molecola di DNA
doppia elica con peso molecolare di circa
2109 e forma circolare, priva di
rivestimenti proteici istone like.
Nel nucleoide sono presenti molecole di
RNA, RNA polimerasi, topoisomerasi e
proteine di natura basica
La forma del nucleoide potrrebbe essere il
risultato di un’equilibrio
tra
DNA e
proteine che tendono a compattare il DNA
Il nucleoide può essere
isolato sotto forma di un
complesso di cui l’80% è
DNA
(negli eucarioti il DNA è solo
il 50%)
Che ruolo hanno le proteine associate al nucleoide ???
STRUTTURALE ????
Sono state isolate numerose proteine che legano il DNA ma nessuna
di queste ha un’evidente ruolo strutturale
INFATTI
Non mostrano forte affinità con il DNA
Non sono in quantità sufficienti x legare tutto il genoma
Mutazioni nel loro gene non provocano interruzioni nella struttura del
nucleosoma
Mutazioni nel loro gene non provocano perdita delle funzioni associate alla
sopravvivenza del genoma
Sono ridondanti
ciascuna può sostituire le altre
Sarebbe necessaria una delezione del gene di tutte per
interferire seriamente sulla struttura del nucleoide
QUINDI ????
E’ evidente il ruolo di queste proteine nella stabilizzazione
del nucleosoma ma non un ruolo strutturale
CHE RUOLO HA L’RNA ?????
TUTTE LE ANALISI SVOLTE FINORA NON HA DATO UNA RISPOSTA
L’ RNA nascente è associato al nucleoide ma non è indispensabile per
la stabilizzazione
MA
Il trattamento con RNasi provoca una destabilizzazione della struttura
dei nucleoidi
Analisi per microscopia a fluorescenza dei nucleoidi.
Le cellule sono state colorati con
DAPI che legandosi
preferenzialmente al DNA
appare in fluorescenza BLU e
con INSULINA (giallo verde)
che non penetra nello spazio
occupato dal nucleoide nel
citoplasma
Organizzazione del cromosoma batterico ad anse
Il cromosoma potrebbe
essere organizzato in
domini topologicamante
indipendenti , superavvolti
negativamente. Analizzando
il numero di tagli necessari
per rilassare
completamente il DNA è
stato possibile valutare la
quantità di topodomini
presenti in E.coli (100).
Tenendo conto che il
cromosoma di E.coli è di
4.600 kb la taglia media dei
domini è valutata di circa
50 kb
LA STRUTTURA AD ANSE DEL CROMOSOMA BATTERICO
Il DNA è probabilmente organizzato in
anse ad alta superelicità (vi sarebbero
circa 50 anse per cromosoma).
Ogni ansa sarebbe stabilizzata da una
molecola di RNA.
Un taglio nell’RNA rilassa l’ansa ma non
modifica la superelicità.
Un taglio a singola elica nel DNA rilassa
la superelica e lascia intera l’ansa.
Organizzazione del nucleoide batterico: le principali proteine associate
al nucleoide
C-domain
(DNA binding)
adapted from
Ussery et al., 2001
N-domain
(protein oligomerization
Nucleoid
proteins are
homo- or
heterodimers
La proteina MukB una proteina SMC like
La proteina MukB è una proteina SMC like ( Structural Maintanance of
chromosome) presente in molti batteri.
In assenza della proteina MukB i batteri diventano termosensibili e a
temperatura permissiva hanno una crescita ridottissima :si osserva una
decondensazione del DNA ed una perdita del nucleoide ad alta
frequenza.
La proteina MukB svolge un ruolo importante anche nella segregazione
dei cromosomi in seguito a divisione cellulare.
La perdita di MukB e della proteina del nucleoide HU è letale per la
cellula.
La proteina MukB una proteina SMC like
La proteina Muk B come molte proteine della famiglia SMC ,è
costituita da due domini globulari N- e C- terminali (teste) separati da
2 regione coiled-coil intervallate da una terza regione globulare che
costituisce una cerniera flessibile.
Le proteine MuKB sono omodimeri ed hanno nelle regioni C- e Nterminali dei domini ATPasici conservati.
La proteina MukB una proteina SMC like
Nella forma chiusa la proteina
scorre sul DNA ed è in grado
di indurne il ripiegamento.
La proteina MukB potrebbe
legare il DNA in due punti
interagendo con i domini
testa e poi grazie alla
flessibilità della regione
cerniera provocarne il
ripiegamento e la
condensazione
La proteina HU
Caratteristiche
• proteina basica
• molto abbondante 30000 copie/cellula
• la più abbondante tra le proteine del nucleoide
•nessuna sequenza consenso di legame al DNA
Eterodimero
Funzioni
• compatta il DNA in strutture nucleosoma-simili
• induce curvatura nel DNA
• riconosce il DNA curvo
HU lega il DNA e lo ripiega
Superavvolgimento mediato da HU. I dimeri di
HU si legano in vitro a distanza di circa 9 bp.
Il legame di HU ogni 9 bp prevede interazioni
con i dimeri adiacenti con formazione di una
struttura solenoidale che si avvolge in senso
sinistrorso. Ciascun avvolgimento toroidale
avverrebbe intorno a 6 dimeri di HU
La proteina IHF
Integration Host factor
Caratteristiche
• proteina basica
• 5-10 volte meno abbondante di HU
• abbondante in fase stazionaria
•debole specificità di sequenza per il legame al DNA (YAANNNNTTGATW)
Eterodimero
•Forte ruolo strutturale; curvando il DNA fino a 140°C permette a siti di
DNA distanti di trovarsi ravvicinati favorendo cosi sia processi di
trascrizione che di regolazione
La curvatura mediata da
IHF può facilitare il
legame di altre
Proteine con siti di
DNA anche molto
distanti tra loro.
Le anse di DNA risulterebbero
ancorate alla membrana durante il
processo di trascrizione/traduzione/tra-slocazione di
proteine di membrana
osservazioni immunocitochimche
mediante l'uso di anticorpi antitopoisomerasi I e antiRNApolimerasi, indicano che
entrambi gli antigeni mappano alla
superficie del nucleoide
Virus
Il virus è una piccola particella che può
infettare altri organismi.
Parassita obbligato intracellulari, cioè può
riprodursi solamente infettando una cellula
ospite poiché non possiede un macchinario
cellulare per la riproduzione.
Il termine virus in genere si riferisce a quelle
particelle che infettano eucarioti (pluricellulari
e unicellulari)
il termine batteriofago o fago è usato per
descrivere quelle particelle che infettano le
cellule procariotiche.
I genomi virali possono essere fatti sia di
DNA che di RNA. L’acido nucleico è
circondato da un rivestimento che
consiste di proteine o proteine e lipidi. Un
genoma virale codifica non solo le
proteine di rivestimento ma anche gli
enzimi necessari per la replicazione
dell’acido nucleico virale durante il ciclo
infettivo. I virus utilizzano il macchinario
metabolico della cellula ospite per
replicarsi.
CICLO LITICO
batteriofago T4
CICLO
LISOGENICO



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)







![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)