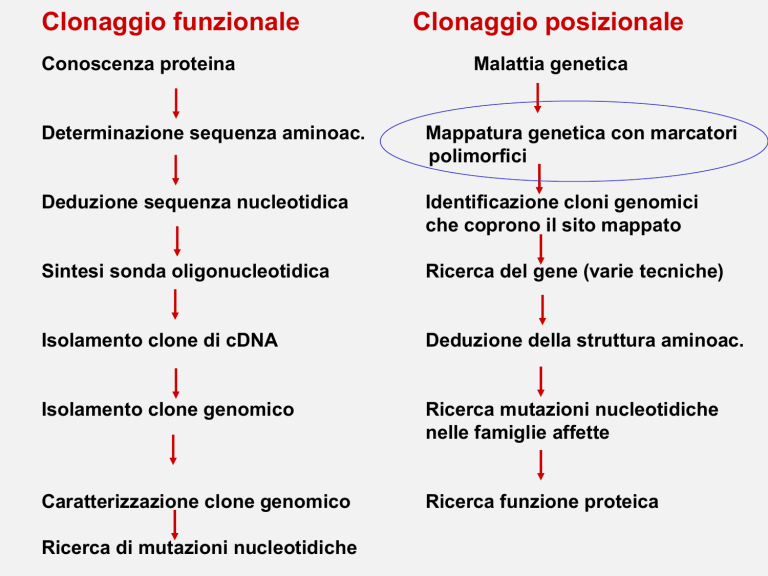
Clonaggio funzionale
Conoscenza proteina
Clonaggio posizionale
Malattia genetica
Determinazione sequenza aminoac.
Mappatura genetica con marcatori
polimorfici
Deduzione sequenza nucleotidica
Identificazione cloni genomici
che coprono il sito mappato
Sintesi sonda oligonucleotidica
Ricerca del gene (varie tecniche)
Isolamento clone di cDNA
Deduzione della struttura aminoac.
Isolamento clone genomico
Ricerca mutazioni nucleotidiche
nelle famiglie affette
Caratterizzazione clone genomico
Ricerca funzione proteica
Ricerca di mutazioni nucleotidiche
MARCATORI GENETICI
Un marcatore genetico è paragonabile ad un cartellino
colorato che serve a “marcare” gli individui in un pedigree.
Un marcatore genetico per essere tale deve essere polimorfico
ed i suoi diversi alleli possono essere utilizzati per seguire la
segregazione di alleli per un altro gene quando si effettuano
incroci.
TIPI DI MARCATORI POLIMORFICI
- Polimorfismi immunologici (es: gruppi sanguigni, rilevabili con
tecniche sierologiche)
- Polimorfismi genetici a livello proteico (rilevabili attraverso
l’elettroforesi ed altre tecniche di caratterizzazione degli isozimi)
- Polimorfismi genetici a livello del DNA
VARIABILITA’ GENETICA SEMPLICE
POLIMORFISMO: presenza di 2 o più fenotipi alternativi
(morfi) a trasmissione mendeliana
MORFI: generalmente determinati da alleli diversi ereditati
in modo mendeliano
POLIMORFISMO GENETICO
Carattere monogenico presente in una popolazione con
una frequenza 1%
f(A) = p
f(B) = q
ETEROZIGOSITA’ (H)
H = 2pq
H aumenta in relazione al numero di alleli del locus
TIPI DI MARCATORI GENETICI NELL’UOMO
Tipo di marcatore
n° loci
Caratteristiche
Gruppi sanguigni
1910-1960
20
sangue fresco, dominanza
Varianti proteiche
1960-1975
30
siero fresco, saggi specialistici,
polimorfismo limitato
RFLP del DNA
1975-
>105
2 alleli, eterozigosità massima 0,5
VNTR del DNA
(minisatelliti)
1985-
>104
Molti alleli, altamente informativi
VNTR del DNA
(microsatelliti)
1989-
>105
Molti alleli, altamente informativi
SNP del DNA
1998-
>106
Meno informativi dei microsatelliti
LIVELLO DI POLIMORFISMO
- Eterozigosità
- Contenuto d’informazione del polimorfismo (Polymorphism
Information Content = PIC)
TASSO DI MUTAZIONE
Singola Sostituzione Nucleotidica (SNS): 10-9
Variable Number of Tandem Repeat (VNTR): minisatelliti 10-2 - 10-3
microsatelliti 10-3 - 10-4
Il genoma è un serbatoio potenziale di polimorfismi così vasto da
consentire di individuare i marcatori più adatti per affrontare un
ampio spettro di problematiche della genetica delle popolazioni
umane.
MAPPAGGIO GENETICO DEI CARATTERI MENDELIANI
1
2
Analisi della segregazione degli alleli per il locus A e degli alleli del locus B
Dalle meiosi di II-1 è possibile stabilire: 5 combinazioni parentali e
2 ricombinanti
Dopo quante meiosi si può stabilire se i loci A e B sono associati o
indipendenti?
LA MAPPATURA GENETICA PRIMA DEI MARCATORI
MOLECOLARI DEL DNA
Gruppi di associazione con loci (marcatori proteici) noti:
ABO - adenilatochinasi
ABO - sindrome unghia- rotula
Duffy - cataratta congenita
Rh - ellissocitosi
Collinesterasi - transferrina
Lutheran - secretore
G6PD - emofilia
G6PD - daltonismo
A1 Y
A1 /A2
D
D/d
c
A2 = Allele enzimopenia G6PD
A2 Y A1 Y
A2 Y
d = allele daltonismo
D
D
d
MEIOSI INFORMATIVE E NON INFORMATIVE
A) NON INFORMATIVA
A1 A1
A2 A2
Gli alleli del marcatore in omozigosi
nel padre non possono essere
distinti
A1 A2
B) NON INFORMATIVA
A1 A2
A1 A2
A1 A2
La figlia può aver ereditato A1 dal
padre e A2 dalla madre o viceversa
MEIOSI INFORMATIVE E NON INFORMATIVE
C) INFORMATIVA
A1 A2
A1 A2
La figlia ha ereditato A1 dal padre
(fratria A1 A1 alternativa a A1 A2 della
famiglia B)
A1 A1
D) INFORMATIVA
La figlia ha ereditato A1 dal padre
A1 A2
A3 A4
A1 A4
UNA MEIOSI E’ INFORMATIVA, OVVERO UTILE PER GLI
STUDI DI ASSOCIAZIONE (LINKAGE), QUANDO SI PUO’
IDENTIFICARE LA FASE DEI 2 LOCI E QUINDI STABILIRE
SE IL GAMETE E’ O MENO RICOMBINANTE
PERCHE’ LA MEIOSI SIA INFORMATIVA IL GENITORE
DEVE ESSERE DOPPIO ETEROZIGOTE PER I DUE LOCI
Es: locus malattia:
Allele normale (+)/Allele mutato (-)
marcatore: A1/A2
Marcatore
Alleli (Frequenze
alleliche)
H
PIC
A
1
0
0
A
2 (0,5; 0,5)
0,50
0,375
A
2 (0,4; 0,6)
0,48
0,365
A
2 (0,3; 0,7)
0,42
0,332
A
2 (0,1; 0,9)
0,18
0,164
A
4 (0,25;0,25;0,25; 0,25)
0,75
0,703
A
10 (tutti = 0,1)
0,90
0,891
X
2 (0,5; 0,5)
0,5
0,5
X
4 (0,25;0,25;0,25; 0,25)
0,75
0,75
MAPPATURA GENETICA A DUE PUNTI
1) Raccolta famiglie con diagnosi certa di una specifica
malattia il cui locus genico è ignoto
2) Costruzione degli alberi genealogici di tutte le famiglie
3) Determinazione del genotipo di tutti gli individui per uno
o più marcatori polimorfici
4) Se la famiglia è informativa, si classificano gli individui
in ricombinanti o non ricombinanti tra il locus della
malattia ed il marcatore analizzato
………
MAPPAGGIO GENETICO DEI CARATTERI MENDELIANI
1
2
Dalle meiosi paterni: i loci A e B sono indipendenti o associati?
Non si possono classificare II-2 e tutti gli individui della III generazione
rispetto a II-2 (omozigote A1A1B1B1)
Sacchetto con monete di tre possibili tipi: TT (monete con T su
entrambe le facce), oppure TC, oppure CC
4 lanci: T, T, T, T
E’ possibile capire da che tipo di monete è fatto il sacchetto?
Ipotesi che siano CC = Scartata
Ipotesi possibili: A: che siano TT
B: che siano TC
Se non si fossero fatti lanci
Ipotesi A: Ipotesi B 1:1
Si intende per VEROSIMIGLIANZA di un’ipotesi A, rispetto ad una
serie di fatti che costituiscono il risultato R, la probabilità con cui
si sarebbe verificato R se A fosse stata vera.
Si confrontano le VEROSIMIGLIANZE di tutte le ipotesi per
accertare se una è risultata MOLTO PIU’ VEROSIMILE delle
altre
Verosimiglianza dell’Ipotesi A = 1 (che siano TT)
Verosimiglianza dell’ipotesi B = (1/2)4 = 1/16
1
Rapporto -------- = 16 :1
1/16
Quanti lanci bisogna fare prima di assumere che siano TT?
Si decide una soglia per 2 motivi:
1) ridurre al minimo i casi in cui si accetta per buona
un’ipotesi che invece è sbagliata;
2) ridurre al minimo i casi in cui si lascia senza risposta il
problema in esame
UN ESEMPIO DI CALCOLO DI LOD SCORE
Caso in cui
II 1 ha fase nota
NR
NR
NR
NR
R
NR
Probabilità di meiosi ricombinante =
Probabilità di meiosi non ricombinante = (1- )
Probabilità 5 NR e 1R = (1- )5 x 1
Probabilità totale in assenza di associazione = (0,5)6 = (1/2)6
Rapporto della verosimiglianza = (1-)5 x 1 / (1/2)6
(1-)5 x 1
Lod score Z = log10 --------------------(1/2)6
0
Z
-
0,1
0,2
0,3
0,4
0,577
0,623
0,509
0,299
0,5
0
LE CURVE DEI LOD SCORE
Z = lod score
1000
Z = log 10 -------- = 3
(p=0,05)
1
Z > 3 EVIDENZA DI LINKAGE
1
Z = log 10 -------- = - 2
100
Z < - 2 ESCLUSIONE DI LINKAGE
3 > Z > - 2 NON CONCLUSIVI PER
IL LINKAGE
SENZA RICOMBINANTI MASSIMO
DI LOD SCORE E’ PER = 0
INTERVALLO DI CONFIDENZA (95%):
LOD SCORE - 1
es. curva 2 max lod score = 3.7
per = 0.23 limiti fiduciali: 0.17 - 0.32
(1-)5 x 1
Lod score Z = log10 --------------------(1/2)6
0.7
0.6
0.5
0.4
0.3
0.2
0
Z
-
0.1
0
0
0.2
0.4
0.6
0,1
0,2
0,3
0,4
0,577
0,623
0,509
0,299
0,5
0
(1-)5 x 1
(1-) 1 x 5
Z = log10 --------------------- + --------------------(1/2)6
(1/2)6
0.35
0.3
0.25
0.2
0.15
0.1
0.05
0
0
0.1
0.2
0.3
0.4
0.5
0.6
0
Z
-
0,1
0,2
0,3
0,4
0,276
0,323
0,222
0,074
0,5
0
MAPPATURA GENETICA A DUE PUNTI
1) Raccolta famiglie con diagnosi certa di una specifica
malattia il cui locus genico è ignoto
2) Costruzione degli alberi genealogici di tutte le famiglie
3) Determinazione del genotipo di tutti gli individui per uno
o più marcatori polimorfici
4) Se la famiglia è informativa, si classificano gli individui
in ricombinanti o non ricombinanti tra il locus della
malattia ed il marcatore analizzato
………
MAPPATURA GENETICA A DUE PUNTI
......
5) Nel caso di meiosi non informative non è possibile stabilire
con certezza gli individui ricombinanti e non ricombinanti e
quindi se i due loci sono associati
6) Estensione della tipizzazione a più famiglie
Come si stabilisce se i due loci sono associati?
7) Analisi dell’associazione tra il marcatore ed il locus attraverso
il calcolo del lod score per ogni singola famiglia
8) Valutazione del lod score complessivo e utilizzazione del
criterio statistico per decidere l’associazione o l’esclusione di
associazione
STIMA DELLA DISTANZA GENETICA TRA MARCATORI
1
2
Rispetto alla combinazione parentale (II-1): 5 NR, 2 R
Frequenza di ricombinazione = 2/7 = 28,6%
Esempio di analisi di associazione tra marker M e malattia AD
Stima della frequenza di ricombinazione = 2/6 = 0,33
IL LINKAGE A DUE O PIU’ PUNTI E’ ESSENZIALE PER LA
COSTRUZIONE DELLE MAPPE MARCATORE - MARCATORE
QUESTO E’ STATO OTTENUTO CON:
- COLLEZIONE DI FAMIGLIE CON STRUTTURA IDEALE RACCOLTE A
QUESTO SCOPO DAL CEPH DI PARIGI (CENTRE POUR L’ETUDE DES
POLYMORPHISMES HUMAINES): 40 famiglie con 3 generazioni e almeno
6 figli)
- ANALISI DI MARCATORI POLIMORFICI (SOPRATTUTTO
MICROSATELLITI) LOCALIZZATI SU TUTTI I CROMOSOMI IN FAMIGLIE
CEPH ED ISTITUZIONE DI UNA BANCA DATI
- DETERMINAZIONE DELL’ORDINE DEI LOCI NELLE MAPPE
MARCATORE – MARCATORE E DEFINIZIONE DELLE DISTANZE IN
CENTIMORGAN
ESEMPIO DI FAMIGLIA CEPH
Famiglia 12
10
11
12
1
3
4
13
2
5
6
7
8
9
IL PROGETTO GENOMA UMANO
PER LA MAPPATURA DEL GENOMA UMANO SONO STATI UTILIZZATI
NUMEROSISSIMI MARCATORI POLIMORFICI (RFLP E VNTR), NON
NECESSARIAMENTE CORRELATI A GENI ESPRESSI, CON
MODALITA’ DI TRASMISSIONE MENDELIANA
Utilizzi:
1) Mappatura genetica di loci sconosciuti responsabili di malattie
genetiche;
2) Clonaggio genico;
3) Diagnosi prenatale e presintomatica.
23 GRUPPI DI ASSOCIAZIONE (22 AUTOSOMI E CROMOSOMA X)
Mappatura
genetica
Mappatura
Mappatura
fisica bassa fisica alta
risoluzione risoluzione
Mappatura
del trascritto
Sequenziamento
del gene
Associazione (linkage) tra il locus A e il locus ignoto in 3 famiglie con
la stessa patologia
A1
D
A2
A2
d
d
A3
d
A3
D
A2
A2
d
A3
d
d
A1
A2
A2
A3
A1
A3
A3
A2
D
d
d
d
D
d
D
d
A2
D
A4
d
A3
A3
D
d
A2
A3
d
d
A2
A3
d
d
A2
A2
A4
A3
A2
A3
A4
A2
D
d
d
d
D
d
d
d
STIMA DELLA RELAZIONE TRA DISTANZA
GENETICA E FISICA
Stima della distanza genetica totale nella specie umana:
somma della lunghezza totale dei cromosomi ottenuta dalla
mappatura genetica attraverso meiosi di origine paterna o
materna
= 2644 cM (maschio)
= 4481 cM (femmina)
Per 3000 Mb = 3 x 10 9 bp di genoma
Maschio
1 cM = 1.13 Mb = 1.13 x 106 bp
Femmina
1 cM = 0.67 Mb = 0.67 x 106 bp
Media : 1 cM = 0.9 Mb = 0.9 x 106 bp
Il tasso di ricombinazione non è identico lungo il cromosoma
La distribuzione dei ricombinanti lungo il cromosoma non è casuale e
dipende dal sesso












