UNIVERSITÀ
Docente: Giorgio Valentini
Istruttore: Matteo Re
C.d.l.
DEGLI
STUDI DI MILANO
Biotecnologie Industriali e Ambientali
Biologia
computazionale
A.A. 2010-2011 semestre II
p8
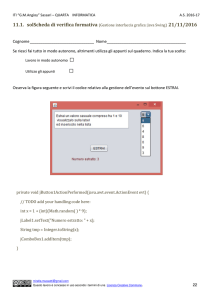
Hidden Markov Models
• Programmazione dinamica in PERL (2)
• Implementazione di un algoritmo che utilizzi tecniche di
programmazione dinamica ed un modello generativo
• Hash di hash
• Hash di array
• Biologia computazionale
• Decoding di una sequenza di osservazioni dato un HMM
• Implementazione algoritmo VITERBI
• Addestramento di un HMM a partire da una collezione di
sequenze biologiche a stati noti
Obiettivi
Linee guida
• Il livello di complessità di questa esercitazione è alto
• Cercate di risolvere il problema dopo averlo suddiviso in sottoproblemi
• Indipendentemente dal fatto che lo script Perl funzioni o meno l’esercizio
NON verrà valutato se, insieme allo script, non verrà inviato anche lo
pseudocodice.
• Modalità di svolgimento dell’esercitazione:
• Scaricare dal sito web del corso il file HMM_exampleVIT_ls.pl
• Questo script è incompleto
• La posizione delle parti da aggiungere è evidenziata da questo
commento:
########### description # FILL IN ##################
description: descrizione dell’operazione da svolgere
• Una differenza importante tra le esercitazioni precedenti e questa è che,
dopo aver reso utilizzabile lo script dell’algoritmo di Viterbi, dovrete
scrivere senza nessun template, degli script Perl che vi permettano di
stimare i parametri dell’HMM da inserire in HMM_example_VIT_ls.pl per
effettuare il decoding di una sequenza proteica. I file contenenti le
collezioni di sequenze sono reperibili sul sito del corso.
3151146666624
(osservazioni)
+
Input:
HMM
DECODING (VITERBI):
Permette di identificare:
La sequenza di stati che, con probabilità massima, ha
prodotto l’intera sequenza di osservazioni (ma non ci dice
nulla rispetto alla probabilità di essere in un certo stato ad
una determinata posizione nella sequenza).
E’ basato su:
tecniche di programmazione dinamica.
VITERBI: algoritmo
Input: x = x1……xL + HMM
Inizializzazione:
V0(0) = 1
Vk(0) = 0, per ogni k > 0
Iterazione:
Vj(i)
(0 è una prima posizione fittizia)
= ej(xi) maxk akj Vk(i – 1)
Ptrj(i) = argmaxk akj Vk(i – 1)
Terminazione:
P(x, *) = maxk Vk(L)
Traceback:
L* = argmaxk Vk(L)
i-1* = Ptri (i)
E’ di fondamentale importanza
progettare bene la parte dell’algoritmo che realizza l’iterazione !
Lo stato finale è determinato dallo stato a valore massimo
dell’ultimo step di iterazione.
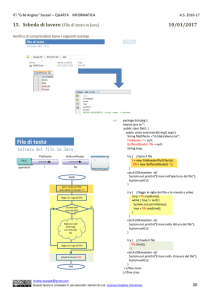
Realizzazione algoritmo VITERBI
1)
2)
3)
4)
5)
6)
7)
Acquisizione sequenza di osservazioni
Definizione parametri del modello HMM
Inizializzazione Viterbi
Iterazione Viterbi
Determinazione ultimo stato
Traceback (costruzione sequenza di stati)
Stampa output: sequenza stati
Realizzazione algoritmo VITERBI
•
Inizializzazione Viterbi
# Initialization:
my %v = ( 'F' => [0], 'L' => [-0.51] );
NB: hash di arrays .
Una variabile dichiarata in questo modo è un normale
hash ma i suoi valori sono arrays (come potete
indovinare dalle parentesi quadre) . I valori inseriti si
trovano in POSIZIONE 0 (primo elemento) degli
array. I valori possono essere aggiunti così:
$v{chiave_hash}->[indice_array] = valore ;
•
Realizzazione algoritmo VITERBI
Inizializzazione Viterbi
# Initialization:
my %v = ( 'F' => [0], 'L' => [-0.51] );
St.
F
L
V_F
V_L
p_F
p_L
3
1
5
1
1
6
6
6
Obs.
FF
0.64
LF
-1.61
FL
-2.30
LL
0.59
0
-0.51
0
-0.51
Questo passaggio corrisponde
alla prima colonna nella
Matrice di progr. dinamica.
Assumiamo uguali le probabilità
Iniziali di transire in stato F o L.
F = E_f(0) + 0 + 0 = 0
L = F_l(0) + 0 + 0 = -0.51
Realizzazione algoritmo VITERBI
•
Definizione della procedura di iterazione
Pos. i
prob. di essere
in stato k alla
posizione i
Per ogni stato k, e per una posizione
fissata i nella seq.,
Vk(i) = max{1… i-1} P[x1…xi-1, 1, …, i-1, xi, i = k]
Emissione
osservazione
in pos. i
Massimizzazione Osservazioni da
probabilità seq. posizione 1
di stati da pos. 1 a posizione i-1
a pos. i-1
ITERAZIONE
Seq. Stati da
posizione 1
a posizione i-1
Pos. i+1
Vl(i+1) = el(xi+1) maxk akl Vk(i)
emissione
transizione
Assumendo
di essere in
stato k
Viterbi stato k posizione i
Realizzazione algoritmo VITERBI
•
Emissione_F(5) = 0
Definizione della procedura di iterazione
3
F
0.64
1 transizione 5
FF
0.64
F
F
-2.12
L
L
L
max
Vl(i+1) = el(xi+1) maxk akl Vk(i)
emissione
transizione
Viterbi stato k posizione i
VF(i+1) = 0 + 0.64 + 0.64 = 1.28
Realizzazione algoritmo VITERBI
•
Emissione_F(5) = 0
Definizione della procedura di iterazione
3
F
0.64
1 transizione 5
FF
0.64
F
F
-2.12
L
L
L
HMM 1°
ordine
max
Vl(i+1) = el(xi+1) maxk akl Vk(i)
emissione
transizione
Viterbi stato k posizione i
Questo calcolo va eseguito, per ogni coppia di simboli (i, i-1), per ogni
possibile transizione.
Realizzazione algoritmo VITERBI
•
Emissione_F(5) = 0
Definizione della procedura di iterazione
3
F
0.64
1 transizione 5
FF
0.64
F
F
-2.12
L
L
L
HMM 1°
ordine
max
Vl(i+1) = el(xi+1) maxk akl Vk(i)
emissione
transizione
Viterbi stato k posizione i
Questo calcolo va eseguito, per ogni coppia di simboli (i, i-1), per ogni
possibile transizione.
VITERBI: iterazione
my $idx = 1;
foreach my $tmp_obs (@obs_list) {
my $max_state = '';
my $max_state_v = 0;
Per ogni simbolo (colonna)
Per ogni transizione
(doppio foreach)
foreach my $state2 qw(F L) {
my ($max_v, $max_prev_state, $max_path) = (-1,'X','XX');
foreach my $state1 qw(F L) {
my $tmp_trans = $state1.$state2;
####### FILL IN #### VITERBI ITERATION STEP ######
# my $tmp_v =
if(
$tmp_v > $max_v
) {
$max_v = $tmp_v;
$max_prev_state = $state1;
$max_path = $state1.$state2;
}
}
$path{$idx}->{$state2} = $max_prev_state;
$v{$state2}->[$idx] = $max_v;
}
$idx += 1;
}
Vl(i+1) = el(xi+1) maxk akl Vk(i)
VITERBI: iterazione
foreach my $state2 qw(F L) {
my ($max_v, $max_prev_state, $max_path) = (-1,'X','XX');
foreach my $state1 qw(F L) {
my $tmp_trans = $state1.$state2;
####### FILL IN #
# my $tmp_v =
if(
$tmp_v > $max_v
) {
$max_v = $tmp_v;
$max_prev_state = $state1;
$max_path = $state1.$state2;
}
symbol
(seq. cycle)
state1
}
$path{$idx}->{$state2} = $max_prev_state;
$v{$state2}->[$idx] = $max_v;
}
F
NB: Salvataggio variabili VITERBI
per lo step successivo
0.64
-2.12
L
F
idx
state2
F
? (if)
Scelta miglior percorso “locale”
VITERBI: iterazione
my $idx = 1;
foreach my $tmp_obs (@obs_list) {
my $max_state = '';
my $max_state_v = 0;
Lunghezza seq. : L
foreach my $state2 qw(F L) {
N° stati : K
my ($max_v, $max_prev_state, $max_path) = (-1,'X','XX');
foreach my $state1 qw(F L) {
N° stati : K
my $tmp_trans = $state1.$state2;
####### FILL IN #### VITERBI ITERATION STEP ######
# my $tmp_v =
if(
$tmp_v > $max_v
) {
$max_v = $tmp_v;
$max_prev_state = $state1;
$max_path = $state1.$state2;
}
}
$path{$idx}->{$state2} = $max_prev_state;
$v{$state2}->[$idx] = $max_v;
}
$idx += 1;
}
Complessità temporale = K * K * L = K2L
VITERBI: determinaz. stato finale
## Determine the last state ##
my $last_state = 'X';
if( $v{'F'}->[$idx-1] > $v{'L'}->[$idx-1] ){
$last_state = 'F';
Controllo valori ultima colonna
}else{
Valore > determina stato finale
$last_state = 'L';
}
print "Final state: $last_state (v=
$v{$last_state}->[$idx-1])\n";
Stampa stato finale “vincente” e relativo
valore VITERBI
VITERBI: Traceback
## Traceback ##
my @state_seq = ();
for(my $i=$idx-1;$i>0;$i--) {
my $prev_state = $path{$i}->{$last_state};
push(@state_seq,$prev_state);
$last_state = $prev_state;
}
St.
F
L
3
1
5
1
1
6
TRANSIZIONE:
Qui $prev_state
di L diventa F !!!
6
6
6
0.64
1.28
1.92
2.56
3.20
3.84
4.48
5.12
-2.12
-2.12
-1.96
-1.88
-1.80
-1.72
0.39
2.08
-2.81
-2.81
-1.53
-0.89
-0.25
2.00
2.64
3.28
-0.43
-0.43
-0.27
-0.19
-0.11
1.58
3.69
5.38
0
-0.51
VITERBI: Traceback
## Traceback ##
$last_state
(ultima col.)
my @state_seq = ();
for(my $i=$idx-1;$i>0;$i--) {
my $prev_state = $path{$i}->{$last_state};
push(@state_seq,$prev_state);
$last_state = $prev_state;
}
Array stati prodotto da
Traceback (ordine inverso)
necessario reverse …
Si parte da $last_state dell’ultima colonna
Ma questo viene ripetutamente sostituito
dal valore del puntatore della colonna
precedente.
VITERBI: Traceback
## Traceback ##
my @state_seq = ();
for(my $i=$idx-1;$i>0;$i--) {
my $prev_state = $path{$i}->{$last_state};
push(@state_seq,$prev_state);
$last_state = $prev_state;
}
## Print most probable path ##
print "\n[Output]\n";
print join("",@obs_list),"\n";
print join("",reverse @state_seq),"\n";
OUTPUT VITERBI: sequenza di stati che ha prodotto
l’intera serie di osservazioni con probabilità maggiore.
VITERBI: Output aggiuntivo
STAMPA MATRICE VITERBI
my $jseq = join("
\t", @obs_list);
print "\tB\t$jseq\n";
my $arrayref = $v{"F"};
my @Vf = @{ $arrayref };
my $Fvit=join("\t", @Vf);
print "F\t$Fvit\n";
$arrayref = $v{"L"};
my @Vl = @{ $arrayref };
my $Lvit=join("\t", @Vl);
print "L\t$Lvit\n\n";
Estrazione arrays
da hash di array
VITERBI: Output aggiuntivo
STAMPA MATRICE TRACEBACK (path pointers)
my $rHoH = \%path;
my $curpos=0;
for my $k1 ( sort {$a <=> $b} (keys %$rHoH) ) {
print "Step: $k1 \tEmission: $obs_list[$curpos]\n";
for my $k2 ( keys %{$rHoH->{ $k1 }} ) {
print "\tmax path to $k2 : $rHoH->{ $k1 }{ $k2 }\t";
if($k2 ne $rHoH->{ $k1 }{ $k2 }){
print "*\n";
}else{
print "\n";
}
}
$curpos++;
print "\n";
}
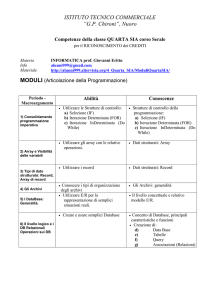
Esercizi
• Rendere lo script funzionante (3 pt) (COMMENTARE IN MANIERA
DETTAGLIATA)
• Scaricare i seguenti files dal sito del corso: soluble_sequences.txt,
transmembrane_sequences.txt e state_sequences. Essi contengono,
rispettivamente, sequenze di porzioni solubili di proteine di lievito, sequenze di
porzioni transmembrana di proteine di lievito e sequenze annotate (T=
transmembrana, S = solubile) di proteine di lievito. Scrivete 2 script Perl. Il
primo servirà a leggere I files con le sequenze amminoacidiche e a generare le
probabilità di emissione per gli stati S (solubile) e T (transmembrana). Il
secondo script leggerà il file delle annotazioni e stimerà le probabilità di
transizione tra I due stati e le probabilità iniziali per gli stati S e T. (3 pt)
• Osservate le pobabilità di emissione dei diversi aa negli stati S e T. Trovate
corrispondenza con le proprietà chimico-fisiche degli aa che li rendono più o
meno adatti a far parte di una regione transmembrana? (commentate la
risposta) (5 pt)
• Modificare lo script HMM_exampleVIT_ls.pl in modo da permettervi di predire
se gli aa di una sequenza proteica fanno parte di una regione transmembrana.
Provate poi a predire la serie di stati che, con maggior probabilità, ha emesso
questa sequenza: KKIIFFFFL. (4 pt)