ASSOCIAZIONE
P
pr+pr+vg+ vg+
F1
pr+pr vg+ vg
Gameti
F2
pr pr vg vg
pr pr vg vg
pr vg
100%
pr+vg+ 50%
pr+ pr vg+ vg
25% +25%
pr vg 50%
pr pr vg vg
25% +25%
ASSOCIAZIONE E SCAMBIO
P
pr+pr+vg+ vg+
F1
pr+pr vg+ vg
Gameti
F2
pr pr vg vg
pr pr vg vg
pr vg
100%
pr+vg+ 44%
pr+ pr vg+ vg
22% +22%
pr vg 44%
pr pr vg vg
22% +22%
pr+vg 6%
pr+ pr vg vg
3% +3%
prvg+ 6%
pr pr vg+ vg
3% +3%
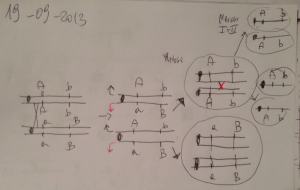
ASSOCIAZIONE e CROSSING-OVER: GAMETI
due o più geni sullo stesso cromosoma
a
b
GAMETI
a
b
ab
a
ab
senza
crossing
over
b
dopo
crossing
over
ab
a
a
b
b
ANAFASE 2
A
A
A
B
B
B
B
aB
a
a
A
A
b
B
anche
gameti
b
ricombinanti
B
ANAFASE 2
A
AB
A
a
B
AB
b
Ab
Solo
gameti
parentali
A
AB
B
Crossing-over, ricombinazione e chiasmi.
GAMETI
a
b
ab
a
a
A
A
b
b
B
B
chiasma
Crossing
over
a
a
A
A
a
B
aB
ANAFASE 1
ANAFASE 2
b
b
B
B
DIPLOTENE
A
terminalizzazione
del chiasma
METAFASE
b
Ab
A
AB
B
ASSOCIAZIONE E SCAMBIO 2
P
b+b+vg+ vg+
F1
b+b vg+ vg
b b vg vg
b b vg vg
b vg
Gameti
F2
100%
b+vg+ 40%
b+ b vg+ vg
20% +20%
b vg 40%
b b vg vg
20% +20%
b+vg 10%
b+ b vg vg
5% +5%
b vg+ 10%
b b vg+ vg
5% +5%
Distanza tra i geni e frequenza di ricombinanti
A
A
a
a
A
A
B
B
b
b
D
D
a
d
a
d
AB
Ab
aB
ab
AB
AD
Ad
aD
ad
AD
Ad
aD
ad
ab
Maggiore è la distanza tra i geni,
più alta è la probabilità che fra di essi vi sia un crossing over,
più alta è la frequenza di ricombinanti fra loro.
Aminoacidi e proteine
struttura primaria: sequenza
lineare degli aminoacidi
legame peptidico
HO
R
R’
HO
C C NH2
O
H
aminoacido 1
C C NH2
O
H
aminoacido 2
H2O
struttura secondaria:
avvolgimenti o ripregamenti
elementari della catena
polipeptidica, dovuti a legami
idrogeno tra aminoacidi vicini
nella sequenza lineare
dipeptide
a elica
foglietto b
struttura quaternaria: composizione
di più catene polipeptidiche, uguali
o diverse, a formare una proteina
multimerica
struttura terziaria: struttura tridimensionale
della catena polipeptidica dovuta alle
interazioni fra aminoacidi anche lontani nella
sequenza lineare
C
In ultima analisi le strutture
secondaria, terziaria e
quaternaria sono dovute alla
struttura primaria
omodimero
N
La natura delle mutazioni geniche: una mutazione
una sostituzione di un aminoacido in una catena
polipeptidica
Frammentando
progressivamente un polipeptide,
è possibile suddividerlo fino ai
singoli aminoacidi
Dalla posizione cambiata di singoli
frammenti o di singoli aminoacidi è
possibile identificare il frammento
che differisce fra l’allele normale e
quello mutato, fino a individuare
l’aminoacido cambiato
Alleli diversi
possono
differire per la
sostituzione di
un solo
aminoacido in
una catena
polipeptidica
(CH3)2
Se si collocano i frammenti di un polipeptide sul bordo
di un foglio di carta bibula e se si espongono 2 margini
ortogonali del foglio a 2 diversi solventi, se ne può
avere un’impronta digitale (fingerprinting) attraverso
la collocazione finale dei frammenti sul foglio
COOH
Nella catena b dell’emoglobina umana, l’aminoacido
in posizione 6 è l’acido glutammico nell’allele normale
dell’emoglobina A (HbA) ed è la valina nell’allele
mutato dell’emoglobina S (HbS)
HO
(CH2)2
HO
C C NH2
O
H
Acido Glutammico
CH
C C NH2
O
H
Valina
Un gene una catena polipeptidica
Colinearità tra gene e catena polipeptidica
Si sono mappate diverse mutazioni in
diversi siti del gene TrpA (per la sintesi del
triptofano) in Escheirichia coli
Mediante l’analisi di fingerprinting, si sono
localizzati, nella catena polipeptidica, le
posizioni degli aminoacidi caratteristici di
ciascuna mutazione mappata
N
15 22
49
175
15: Lys->STOP; 22: Phe->Leu; 49: Glu->Val,Gln,Met; 175: Tyr->Cys.
La sequenza dei siti mutati nel gene è identica alla sequenza delle posizioni degli
aminoacidi sostituiti, anche se le distanze non corrispondono esattamente
La trasformazione batterica: batteri (Streptococcus
pneumoniea) avirulenti (ceppo R) sono trasformati in
batteri virulenti (ceppo S) da batteri virulenti (ceppo S)
uccisi dal calore
Ceppo R
Ceppo S
Uccisione con
il calore
Ceppo
S
Ceppo
S ucciso
dal calore
proteine
lipidi
polisaccaridi
RNA
Ceppo R
DNA
Ceppo R
trasformazione
Ceppo S
Nessuna
trasformazione
trasformazione
Le basi azotate
H
H
N
O
H
C
C
C
N
C
N
C
H
C
O
H
Pirimidine
N
C
C
N
H
N
H
C
C
C
C
N
H
H
Timina
H
Adenina
H
O
C
N
C
C
C
N
C
N
H
C
N
C
N
O
N
Guanina
H
C
Purine
C
C
N
H
H
N
H
Citosina
C
H
La struttura del DNA
O-
Legami idrogeno
O-
C
P
G
Legami idrofobici
O
O
T
H2C5’
BASE …..
O
P
3’
C1’
C4’
H H
H H
C3’
A
5’
C2’
HO
H
1’
P
5’
3’
P
3’
5’
P
3’
1’
1’
5’
1’
Nucleotide
Il DNA è costituito da due catene polinucleotidiche
antiparallele avvolte tra loro a doppia elica
Replicazione semiconservativa del
DNA nei cromosomi degli eucarioti
BrUdR
G1
S
G2
Mitosi (M1)
2n
BrUdR
G1
S
G2
Mitosi (M2)
Struttura dell’RNA
O-
RNA compl. DNA
OP
desossiribosio
ribosio
O
O
H2C5’
BASE …..
O
A
T
G
C
C
G
U
A
C1’
C4’
HH
HH
C2’
C3’
P
5’
OH
H
HO
H
H
H H
C
5’
H
N
N
C
C
O
Timina
Uracile
1’
5’
C
C
3’
P
3’
P
O
C
1’
H
5’
3’
P
3’
1’
1’
La Trascrizione
-35
11-15
nucl
TGTTGACA
-10
5-8
nucl
TATAAT
Sito di inizio
terminazione
promotore
C-G
G-C
C-G
G-C
C-G
G-C
RNA trascritto
Sito di attacco
dell’aminoacido
RNA trascritto
Filamento di DNA da
non trascrivere
Filamento di DNA
da trascrivere
Promotore
Sito di inizio
Terminazione
RNA polimerasi
anticodone
Tipo
abbondanza
coeff. sed.
rRNA
80%
23S, 16S,
5S
tRNA
15%
4S
mRNA
5%
vario
I codoni
UUU
UUC
UUA
UUG
Phe
Phe
Leu
Leu
UCU
UCC
UCA
UCG
Ser
Ser
Ser
Ser
UAU
UAC
UAA
UAG
Tyr
Tyr
STOP
STOP
UGU
UGC
UGA
UGG
Cys
Cys
STOP
Trp
CUU
CUC
CUA
CUG
Leu
Leu
Leu
Leu
CCU
CCC
CCA
CCG
Pro
Pro
Pro
Pro
CAU
CAC
CAA
CAG
His
His
Gln
Gln
CGU
CGC
CGA
CGG
Arg
Arg
Arg
Arg
AUU
AUC
AUA
AUG
Ile
Ile
Ile
Met
ACU
ACC
ACA
ACG
Thr
Thr
Thr
Thr
AAU
AAC
AAA
AAG
Asn
Asn
Lys
Lys
AGU
AGC
AGA
AGG
Ser
Ser
Arg
Arg
GUU
GUC
GUA
GUG
Val
Val
Val
Val
GCU
GCC
GCA
GCG
Ala
Ala
Ala
Ala
GAU
GAC
GAA
GAG
Asp
Asp
Glu
Glu
GGU
GGC
GGA
GGG
Gly
Gly
Gly
Gly
I codoni sono le
triplette di basi di
mRNA che
codificano per
specifici
aminoacidi; sono
complementari e
antiparallele sia
alle
corrispondenti
triplette sul DNA
del gene
trascritto, sia a
quelle degli
anticodoni,
presenti sul tRNA
corrispondente;
la prima base è
all’estremità 5’,
l’ultima
all’estremità 3’
Il codice genetico non è ambiguo: ogni codone codifica per un solo aminoacido
Il codice genetico è degenerato: molti aminoacidi sono codificati da più di un codone
Il codice genetico è universale: quasi tutti i viventi condividono lo stesso codice genetico
Sintesi proteica e traduzione
5S
23 S
50 S
30 S
Sito P
Sito A
16 S
Negli eucarioti l’RNA
prima di entrare nel
citoplasma subisce
una maturazione: al 5’
viene aggiunta una 7metilguanosina, al 3’
una catena di poli-A
aa1
aa2
aa1
aa3
aa1
aa2
AAAA
Il sito di inizio della traduzione
nell’mRNA è una tripletta AUG,
GUG preceduta di poco da
sequenze complementari all’rRNA
16 S; tale tripletta codifica per Nformil-metionina (in E. coli)
il tRNA con l’anticodone complementare
al codone esposto nel sito A vi si lega,
essendo già associato al proprio
aminoacido (aminoacil-tRNA)
Sul sito P è presente un tRNA entrato in
precedente, cui è legato il nascente
polipeptide (peptidil-tRNA);il
polipeptide si stacca dal tRNA sul sito P
e si lega all’aminoacido legato al tRNA
sul sito A
Il tRNA libero sul sito P si allontana, il
codone e il peptidil-tRNA sul sito A si
spostano sul sito P
Non tutto l’mRNA viene tradotto: una
parte, corrispondente agli introni dei geni,
viene staccato e rimosso; l’altra parte,
corrispondente agli esoni dei geni, viene
saldata e tradotta
Quando sul sito A giunge un codone di
terminazione, al posto di un tRNA vi si lega
un Fattore di Rilascio che impedisce
l’allungamento del poipeptide e conclude il
processo
Le fonti della variabilità genetica
Perché sia possibile l’evoluzione, la selezione deve operare su una preesistente
variabilità genetica; ma per effetto della selezione la variabilità genetica viene
ridotta nelle generazioni successive, poiché si trasmettono alla progenie solo gli alleli
e i genotipi più adatti all’ambiente.
Ma per adattarsi a un ambiente mutevole, le specie debbono mantenere un livello adeguato
di variabilità genetica per rispondere tempestivamente alla mutabile pressione selettiva.
Fonti primarie
Mutazioni geniche
Poliploidia, duplicazioni
Nuovi alleli
Geni duplicati
Nuovi geni
Amplificazione
(esponenziale)
Riproduzione sessuale
Ricombinazione
Nuove combinazioni di alleli
Localmente
Migrazioni
Nuovi alleli (localmente)
La genetica delle popolazioni
La genetica di popolazione si occupa della frequenza degli alleli nelle
popolazioni e del loro andamento nel tempo, quindi studia la variabilità
genetica e i fattori che ne influenzano nel tempo i cambiamenti, mirando alla
comprensione dei meccanismi genetici alla base dell’evoluzione.
La genetica formale studia i risultati di
singoli incroci fra 2 individui, che, per i geni
studiati, possono avere al massimo 2 alleli
diversi (se sono eterozigoti); nell’incrocio
tra 2 eterozigoti, ciascuno produce metà
(0,5) gameti con il 1° allele, metà con il 2°;
nella progenie ci si aspetta che un quarto
(0,25) sia omozigote per il 1° allele, un
quarto sia omozigote per il secondo e metà
eterozigote.
A1 0,5
A2 0,5
A1 0,5
A1A1 0,25
A1A2 0,25
A2 0,5
A1A2 0,25
A2A2 0,25
Una popolazione si dice polmorfa per un
gene, se per esso presenta più di un allele; si
dice monomorfa se presenta un solo allele
La genetica di popolazione studia i risultati
di tutti i possibili incroci fra tutti gli
individui di sesso opposto della
popolazione, immaginando di mettere
insieme tutti i gameti dello stesso sesso e di
accoppiare casualmente a 2 a 2 i gameti di
sesso opposto; per i geni studiati il numero
degli alleli diversi può essere qualsiasi,
come può esserlo la loro frequenza.
A1
0,2
A2
0,3
A3
0,5
A1 0,2
A1A1
0,04
A1A2
0,06
A1A3
0,1
A2 0,3
A1A2
0,06
A2A2
0,09
A2A3
0,15
A3 0,5
A1A3
0,1
A2A3
0,15
A3A3
0,25
Le leggi di Hardy-Weinberg
1° legge di Hardy-Weinberg: le frequenze degli alleli in una popolazione non
cambiano passando da una generazione all’altra se:
1) Non c’è selezione
2) Non c’è mutazione
3) Non c’è migrazione
4) La popolazione è infinitamente grande
Se pn è la frequenza relativa dell’allele A1 alla generazione n, quando le 4 condizioni sono
rispettate, la popolazione è all’equilibrio (e non c’è evoluzione!) e:
pn+1 = pn; pn+1-pn= p=0
A1 p
A2 q
A3 r
A1
p
A2
q
A3
r
A1A1
p2
A1A2
pq
A1A3
pr
A1A2
pq
A2A2
q2
A2A3
qr
A1A3
pr
A2A3
qr
A3A3
r2
2° legge di Hardy-Weinberg: le frequenze dei
genotipi diploidi in una popolazione sono
uguali al prodotto delle frequenze degli alleli
(se queste ultime sono i coefficienti di un
polinomio, le prime sono i coefficienti del
quadrato del polinomio ) se:
1) C’è panmissia, cioè se ogni incontro tra i
gameti di sesso opposto ha la stessa probabilità
Se p e q sono le frequenze relative degli
alleli A1 e A2 in una data generazione, le
frequenze relative dei genotipi A1A1,
A1A2 e A2A2 della stessa generazione
sono, rispettivamente: p2, 2pq e q2