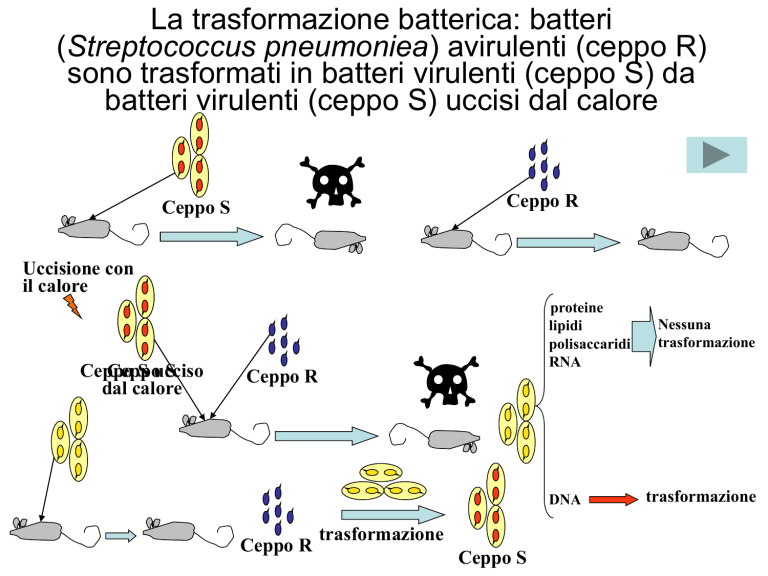
La trasformazione batterica: batteri
(Streptococcus pneumoniea) avirulenti (ceppo R)
sono trasformati in batteri virulenti (ceppo S) da
batteri virulenti (ceppo S) uccisi dal calore
Ceppo R
Ceppo S
Uccisione con
il calore
Ceppo
S
Ceppo
S ucciso
dal calore
proteine
lipidi
polisaccaridi
RNA
Ceppo R
DNA
Ceppo R
trasformazione
Ceppo S
Nessuna
trasformazione
trasformazione
L’infezione di cellule del batterio Escheirichia coli
da parte del fago T2 avviene mediante il DNA del
virus, non mediante le sue proteine
32P
Solo nel DNA
La radioattività
(e il DNA) entra
35S
Solo nelle proteine
La radioattività
(con le proteine)
non entra
Le basi azotate
H
H
N
O
H
C
C
C
N
C
C
H
Pirimidine
N
N
C
H
O
Timina
C
N
H
N
H
C
C
C
C
N
H
H
C
H
Adenina
H
O
C
N
C
C
C
N
C
N
N
Guanina
H
H
C
N
C
C
Purine
C
C
N
H
H
N
H
N
O
Citosina
C
H
La struttura del DNA
O-
Legami idrogeno
O-
C
P
G
Legami idrofobici
O
O
T
H2C5’
BASE …..
O
P
3’
C1’
C4’
H H
H H
C3’
A
5’
C2’
HO
H
1’
P
5’
3’
P
3’
5’
P
3’
1’
1’
5’
1’
Nucleotide
Il DNA è costituito da due catene polinucleotidiche
antiparallele avvolte tra loro a doppia elica
Il modello della doppia elica del DNA
Conoscenze preesistenti: il DNA è un
polimero costituito da un numero molto
elevato di subunità: i nucleotidi (fosfato
+ desossiribosio + 1 base azotata)
Dati cristallografici: la figura ottenuta
dalla diffrazione dei raggi x suggeriva
che il DNA è una lunga molecola lineare,
costituita a due filamenti paralleli
avvolti a doppia elica
Dati chimici:
1) Le molecole puriniche sono nello stesso
numero delle molecole pirimidiniche
(A+G=T+C);
2) Ciascuna delle molecole puriniche ha
una e una sola molecola pirimidinica in
uguale numero e precisamente: A=T, G=C
Ammettendo che le basi azotate si trovino all’interno della doppia elica, solo
l’affacciamento di una purina con una pirimidina garantiscono il diametro
realmente riscontrato della doppia elca
Originalità del modello di Crick e
Watson: la complementarità di coppie di
basi (una purina e una pirimidina) che
con i loro legami idrogeno stabilizzano la
struttura a doppia elica e che, mediante
la separazione dei due filamenti
polinucleotidici e l’incoprorazione di
nucleotidi complementari sullo stampo
dei filamenti preesistenti, garantiscono la
precisione della replicazione del DNA e la
correttezza della trasmissione ereditaria
T
AT
A
G
CG
C
Replicazione semiconservativa:
l’esperimento di Meselson e Sthal
Prima
generazione
controlli
14N
15N14N
15N
Bande di DNA
“pesante e “leggera”
Centrifugazione
DNA
in
Quando le celluledifatte
crescere
gradiente
di cloruro
di cesio.
in 15N vengono
trasferite
in un
Seconda
14
la primaper
generazione
Le coltureN,cresciute
molte
generazione terreno
produce
unainbanda
di DNA
generazioni
un terreno
14N forniscono
intermedia
eN
la eseconda
producele
contenente 15
due
bande,
intermedia
una
posizioni
di una
controllo
per le ebande
leggera.
di DNA “pesante “ e “leggera”.
Incubazione di cellule
pesanti in 14N
Solo il modello semiconservativo
di replicazione di DNA spiega
questi risultati
conservativa
Replicazione semiconservativa
Due bande
15N15N e 14N14N
Due bande
15N15N e 14N14N
dispersiva
Una banda
15N14N
Due bande
15N14N e 14N14N
Una banda
15N14N
Una banda intermedia
tra 15N14N e 14N14N
Replicazione semiconservativa del
DNA nei cromosomi degli eucarioti
BrUdR
Blocco della M1
con colchicina
G1
S
G2
Mitosi (M1)
2n
BrUdR
G1
S
G2
Mitosi (M2)
4n
La forca replicativa
DNA ligasi
girasi
elicasi
5’
3’
3’
5’
Correzione
di bozze
SSB
primasi
lagging strand
RNA primer
DNA polimerasi I
DNA polimerasi III
leading strand
frammento di Okazaki
L’incorporazione di nucleotidi “sbagliati” causa una
distorsione della doppia elica; la DNA polimerasi rimuove
il nucleotide sbagliato e inserisce quello corretto
I cromosomi degli eucarioti e il DNA
1) Misure viscosimetriche e microspettrofotometriche in neuroblasti di Drosophila:
conoscendo la quantità totale di DNA per nucleo diploide (microspettrofotometria)
e la lunghezza media delle molecole di DNA estratte (viscosimetria) si è verificato
che ogni cromosoma è costituito da un’unica molecola di DNA
Quantità di DNA
corrispondente a
circa 108 coppie
di nucletidi
Lunghezza media delle
molecole: circa 1,25x107
coppie di nucleotidi
2) Misure metriche e microspettrofotometriche in cromosomi a spazzola di oociti di
Urodeli: conoscendo la quantità totale di DNA per nucleo diploide
(microspettrofotometria) e la lunghezza media delle fibre di cromatina estese con
microaghi a partire dai cromomeri, verificando che tali fibre sono frammentate
solo con l’uso di endonucleasi, e che, quindi, la loro continuità è dovuta al DNA, si è
verificato che ogni cromosoma è costituito da un’unica molecola di DNA
cromomero
Quantità di DNA
corrispondente a circa 109
coppie di nucletidi
endonucleasi
Lunghezza corrispondente a
circa 109 coppie di nucletidi
Il genoma aploide umano consta di circa 3 miliardi di nucleotidi, organizzati in 23 molecole a
doppia lica di DNA, corrispondenti ai 23 cromosomi dell’assetto aploide umano
La struttura dei cromosomi
degli eucarioti
Solenoide
Impalcatura
nucleare
Condensazione
cromosomica
(mitosi, meiosi)
DNA
Cromatina
Ottamero
di istoni
Nucleosoma
Impalcatura
cromosomica
La replicazione negli
eucarioti e il telomero
Origini di replicazione: singola
nei procarioti, multiple negli
eucarioti
Negli eucarioti, quando si avvicina la forca replicativa, gli
istoni vengono rimossi per consentire l’azione del complesso
di replicazione; a replicazione avvenuta avviene di nuovo
l’aggregazione degli istoni a formare i nucleosomi
All’estremità 3’ del lagging strand, dopo la rimozione del
primer, la DNA polimerasi non può operare
…TTAGGG TTAGGG
3’
TTAGGG…
…CCCAAT
Interviene un enzima ad RNA, la telomerasi che allunga il
lagging strand all’estremità 3’ aggiungendo copie della
sequenza telomerica ripetuta
GGG…
L’allungamento del filamento all’estremità
3’ offre spazio ulteriore all’RNA primer
che consente alla DNA polimerasi di
operare, evitando l’accorciamento del
DNA
G-G
G-G
Il filamento allungato si ripiega più volte a formare
un’elica quadrupla, stabilizzata da un legame particolare
tra 4 guanine affacciate
DNA ripetitivo negli eucarioti
DNA altamente ripetitivo
(“satellite”): 105 – 106 copie di
sequenze fra loro adiacenti di
10-100 nucleotidi
DNA mediamente ripetitivo: 103 –
105 copie di sequenze intersperse
di 200-500 nucleotidi
Geni a copia multipla adiacenti
mediamente ripetitivi: 102 – 103
copie degli stessi geni
Minisatelliti:
–
copie di
sequenze fra loro adiacenti di
10-100 nucleotidi
103
104
Microsatelliti: 10 – 102 copie di
di-, trinucleotidi fra loro
adiacenti
Sequenza
base
Sequenza
base
105 – 106
copie
Localizzazione: p. es.
103– 105
eterocromatina
copie
centromerica
Localizzazione: lungo
2 – 103
Uno o tutti i10
cromosomi
copie
pochi geni
SequenzaLocalizzazione:
103 – 10p.4 es. geni per
RNA ribosomale,
braccio
base
copie
corto dei cromosomi
umani
Localizzazione:
siti specifici
13, 14, 15,
21, 22
di cromosomi
specifici
CAG…………….CAG
Localizzazione: siti specifici
di cromosomi specifici, anche
entro geni
Microsatellite
(CAG)n
Gene della corea di
Huntigton
Cromosomi di cloroplasti e mitocondri
I cromosomi di
cloroplasti e
mitocondri
sono circolari,
come quelli dei
procarioti
hanno un’unica
origine di
replicazione
non effettuano
alcun crossing over
sono presenti in
più copie entro lo
stesso organello
cloroplasto
I cromosomi dei
cloroplasti
I cromosomi dei
mitocondri
non sono autosufficienti: sono necessarie
anche istruzioni dei geni nucleari per il
funzionamento dell’ organello
hanno 100.000 –
300.000 basi
hanno 16.000 –
300.000 basi
mitocondrio
geni singoli
geni duplicati
hanno alcune
duplicazioni e intervalli
non codificanti
hanno geni in un’unica
copia, senza intervalli
non codificanti
È stata formulata l’ipotesi che questi organelli siano il risultato dell’evoluzione
di procarioti simbionti all’interno delle primitive cellule eucariotiche
Meccanismi molecolari del crossing over
Le 2 doppie eliche di DNA appartengono a 2
cromatidi non fratelli di 2 cromosomi omologhi
A
b
b
A
DNA eteroduplex
B
1) Si producono 2 rotture a singolo filamento
a
b
b
A
a
B
2) Le 2 rotture a singolo filamento vengono
saldate mediante uno scambio a singolo
filamento
Se prima delle
a
rotture
del punto 3
avviene uno
scorrimento dei
filamenti del DNA,
si possono formare
dopie eliche
“ibride”
(eteroduplex)
Ruotando la figura
precedente, si
osserva che si è
compiuto il crossing
over
Ruotando la figura precedente in modo da evitare
accavallamenti, si ottiene questa configurazione di DNA
cruciforme, effettivamente osservato in meiosi di lievito
3) Si possono produrre 2 rotture a singolo filamento sui due
filamenti di DNA non coinvolti negli scambi precedenti
B
b
A
a
4) Si saldano le 2 rotture
con uno scambio a singolo
filamento
A
b
B
a
B
Aminoacidi e proteine
struttura primaria: sequenza
lineare degli aminoacidi
legame peptidico
HO
R
R’
HO
C C NH2
O
H
aminoacido 1
C C NH2
O
H
aminoacido 2
H2O
struttura secondaria:
avvolgimenti o ripregamenti
elementari della catena
polipeptidica, dovuti a legami
idrogeno tra aminoacidi vicini
nella sequenza lineare
dipeptide
a elica
foglietto b
struttura quaternaria: composizione
di più catene polipeptidiche, uguali
o diverse, a formare una proteina
multimerica
struttura terziaria: struttura tridimensionale
della catena polipeptidica dovuta alle
interazioni fra aminoacidi anche lontani nella
sequenza lineare
C
In ultima analisi le strutture
secondaria, terziaria e
quaternaria sono dovute alla
struttura primaria
omodimero
N
Un gene una catena polipeptidica
Frammentando progressivamente un polipeptide, è
possibile averne un’impronta digitale (fingerprinting)
attraverso lo spostamento dei frammenti in 2 diversi
solventi: così è possibile identificare il frammento che
differisce fra l’allele normale e quello mutato, fino a
individuare l’aminoacido cambiato
Alleli diversi
possono
differire per la
sostituzione di
un solo
aminoacido in
una catena
polipeptidica
Nella catena b dell’emoglobina umana, l’aminoacido
in posizione 6 è l’acido glutammico nell’allele normale
dell’emoglobina A (HbA) ed è la valina nell’allele
mutato dell’emoglobina S (HbS)
Colinearità tra gene e catena polipeptidica
Mediante l’analisi di cotrasduzione con il
fago P1, si sono mappate diverse mutazioni
in diversi siti del gene TrpA (per la sintesi
del triptofano) in Escheirichia coli
Mediante l’analisi di fingerprinting si sono
localizzati, nella catena polipeptidica, le
posizioni degli aminoacidi caratteristici di
ciascuna mutazione mappata
N
15 22
49
175
15: Lys->STOP; 22: Phe->Leu; 49: Glu->Val,Gln,Met; 175: Tyr->Cys.
La sequenza dei siti mutati nel gene è identica alla sequenza delle posizioni degli
aminoacidi sostituiti, anche se le distanze non corrispondono esattamente
Struttura dell’RNA
O-
RNA compl. DNA
OP
desossiribosio
ribosio
O
O
H2C5’
BASE …..
O
HH
HH
C2’
C3’
G
C
C
G
U
A
P
OH
H
HO
5’
H
H
H H
C
5’
O
C
1423
1’
3’
P
1’
3’
P
C
N
N
H
C
O
Timina
Uracile
5’
3’
P
3’
1’
1’
231
21
1
RNA
trascritti DNA
5’
C
C
T
C1’
C4’
H
A
Ibrido
RNA-DNA
Ibrido
RNA-DNA
Filamenti di RNA
non ibridato
La Trascrizione
-35
11-15
nucl
TGTTGACA
-10
5-8
nucl
TATAAT
promotore
Sito di inizio
terminazione
C-G
G-C
C-G
G-C
C-G
G-C
RNA
Sito di attacco
dell’aminoacido
Tipo
anticodone
abbondanza
coeff. sed.
rRNA
80%
23S, 18S,
5S
tRNA
15%
4S
mRNA
5%
vario
La trascrizione e il processamento
dell’RNA negli eucarioti
promotore
GGCCAATCT
25-30 nucl
TATAAAA
RNA polimerasi II
Sito di inizio
Fattori di
trascrizione
Regione centrale del promotore
Intensificari
r.c.p
“snurp”
AAAA
Nucleo
Negli eucarioti l’RNA prima di
entrare nel citoplasma subisce
una maturazione: al 5’ viene
aggiunta una 7-metilguanosina,
al 3’ una catena di poli-A
Non tutto l’mRNA viene tradotto: una
parte, corrispondente agli introni dei geni,
viene staccato e rimosso; l’altra parte,
corrispondente agli esoni dei geni, viene
saldata e tradotta
Le mutazioni
Le mutazioni sono cambiamenti accidentali,
casuali ed ereditabili del materiale genetico
Le mutazioni geniche consistono nel
cambiamento di un gene da una forma allelica
all’altra.
Le mutazioni cromosomiche strutturali consistono in
riordinamenti dei cromosomi che portano alla perdita,
all’acquisto o allo spostamento di geni.
Le mutazioni cromosomiche numeriche consistono in
cambiamenti del numero dei cromosomi che portano alla
perdita oall’acquisto di geni.
A B
C
c D
A B D
C D
A B
A B
C D
C D
Natura molecolare delle
mutazioni geniche
Delezione di base
Sostituzione di base
TAG AGTAG
TAGTAG
TAG
TAG
TAG
TAG
transizione
TAG TAG TAG TAG TAG
TAG T
TAG
TAG
TAG
TAG
TAG
TAG
TAG
TAG
G
A
Inserzione di base
T
C
CODICE GENETICO
trasversione
purina
pirimidina
Per codificare 20 aminoacidi, non bastano 4
nucleotidi e nemmeno 16 coppie di nucleotidi;
sono necessarie combinazioni di almeno 3
nucleotidi (triplette) che sono 64
1
2
3
tripletta
Lettura per giustapposizione
TAG TAG TAG TAG TAG
1 23
tripletta
Lettura per sovrapposizione
Natura del codice genetico
1
2
3
tripletta
GTAG TAG TAG TAG
TA C
1 23
tripletta
proflavina
rII+
Se la lettura del codice fosse stata per sovrapposizione,
una singola sostituzione di base avrebbe comportato la
modificazione di tanti aminoacidi quante sono le basi
che fanno parte dell’unità codificante (nell’esempio
presente, supponendo un codice a triplette, gli
aminoacidi modificati da una singola sostituzione di
base avrebbero dovuto essere 3); invece si osserva la
modificazione di un solo aminoacido; pertanto la
lettura del codice è per giustapposizione.
proflavina
rII
rII+
rII+
soppressore
FCO
proflavina
rII+
rII
proflavina
rII
rII+
proflavina
rII
rII+
3 mutazioni successive dello stesso segno, separabili per
crossing over (3 inserzioni o 3 delezioni) ripristinano la
corretta cornice di lettura; le sfasature dovute alla
prima mutazione si mantengono a valle di essa fino a
che non sono neutralizzate dalla seconda e dalla terza;
quindi il codice è a triplette
TAG TG TA AG TAG
TAG TAG TAG TAG TAG
rII
+
-
2 mutazioni successive di
segno opposto (inserzione +
delezione), separabili per
crossing over, ripristinano la
corretta cornice di lettura;
quindi il codice è letto senza
interruzioni e le sfasature
dovute alla prima mutazione si
mantengono a valle di essa
fino a che non sono
neutralizzate dalla seconda
TAG TTAG TAG AG TAG
Decifrazione del codice genetico
UUU UUU UUU
Phe Phe Phe
Costruendo un mRNA in vitro costituito di un unico nucleotide
(X), ci sarà una sola tripletta (XXX); effettuando la sintesi
proteica in vitro, si formerà un polipeptide costituito da un unico
aminoacido (Z-Z-Z-…), codificato dalla tripletta XXX
Costruendo un mRNA in vitro costituito di una miscela di nucleotidi (X e Y) in
proporzioni note (p e q), ci sarà una miscela di triplette in proporzioni prevedibili (p3
XXX; p2q XXY, XYX, YXX; pq2 XYY, YXY, YYX; q3 YYY); effettuando la sintesi
proteica in vitro, si formerà un polipeptide costituito da una miscela di aminoacidi nelle
stesse proporzioni delle triplette che li codificano
U:G = 3:1
UUU
27/64
UUG, UGU, GUU
9/64
Phe
27/64
Val
12/64
Leu, Cys
9/64
UGG, GUG, GGU
3/64
Gly
4/64
GGG
1/64
Trp
3/64
Usando mini mRNA (1 sola tripletta), capaci di determinare il legame tra il tRNA
complementare e il suo aminoacido consentendone l’identificazione, e mRNA
costituiti da copolimeri ripetitivi della stessa tripletta (XYW)n, si è completamente
decifrato il codice genetico
I codoni
UUU
UUC
UUA
UUG
Phe
Phe
Leu
Leu
UCU
UCC
UCA
UCG
Ser
Ser
Ser
Ser
UAU
UAC
UAA
UAG
Tyr
Tyr
STOP
STOP
UGU
UGC
UGA
UGG
Cys
Cys
STOP
Trp
CUU
CUC
CUA
CUG
Leu
Leu
Leu
Leu
CCU
CCC
CCA
CCG
Pro
Pro
Pro
Pro
CAU
CAC
CAA
CAG
His
His
Gln
Gln
CGU
CGC
CGA
CGG
Arg
Arg
Arg
Arg
AUU
AUC
AUA
AUG
Ile
Ile
Ile
Met
ACU
ACC
ACA
ACG
Thr
Thr
Thr
Thr
AAU
AAC
AAA
AAG
Asn
Asn
Lys
Lys
AGU
AGC
AGA
AGG
Ser
Ser
Arg
Arg
GUU
GUC
GUA
GUG
Val
Val
Val
Val
GCU
GCC
GCA
GCG
Ala
Ala
Ala
Ala
GAU
GAC
GAA
GAG
Asp
Asp
Glu
Glu
GGU
GGC
GGA
GGG
Gly
Gly
Gly
Gly
I codoni sono le
triplette di basi di
mRNA che
codificano per
specifici
aminoacidi; sono
complementari e
antiparallele sia
alle
corrispondenti
triplette sul DNA
del gene
trascritto, sia a
quelle degli
anticodoni,
presenti sul tRNA
corrispondente;
la prima base è
all’estremità 5’,
l’ultima
all’estremità 3’
Il codice genetico non è ambiguo: ogni codone codifica per un solo aminoacido
Il codice genetico è degenerato: molti aminoacidi sono codificati da più di un codone
Il codice genetico è universale: quasi tutti i viventi condividono lo stesso codice genetico
Anticodoni e tRNA
Ogni specie di tRNA ha un anticodone specifico e lega un solo aminoacido
DEGENERAZIONE DEL CODICE
CODONI NON SENSO
1) Alcuni aminoacidi si legano a più di un tRNA
UAA, UAG, UGA
2) Alcune basi all’estremità 5’ dell’anticodone
1) Usati come mini
(corrispondenti all’ultima base, in 3’, del codone)
mRNA non legano alcun
presentano un appaiamento non stringente (vacillamento)
aminoacil-tRNA
che riconosce più di una base in 3’ del codone; quindi
alcuni tRNA riconoscono più di un codone
2) Prodotti in seguito a una singola
al 5’ dell’anticodone
al 3’ del codone
sostituzione di base a partire da
triplette molto simili, producono, in
G
U, C
corrispondenza della loro posizione,
un’interruzione della catena
polipeptidica (mutazioni non senso)
C
G
3) Le mutazioni non senso possono
A
U
essere neutralizzate da mutazioni
soppressori esterne al gene: si tratta di
mutazioni nell’anticodone di un tRNA
U
A, G
(p.es. GUA -> UUA, anticodone di UAA)
I
U, C, A
I codoni non senso sono detti di
terminazione perché interrompono la
sintesi di una catena polipeptidica
Sintesi proteica e traduzione
5S
23 S (28S)
50 S
30 S
Sito P
Sito A
16 S (18S)
aa1
aa2
aa1
aa3
aa1
aa2
Il sito di inizio della traduzione
nell’mRNA è una tripletta AUG,
GUG preceduta di poco da
sequenze complementari all’rRNA
16 S; tale tripletta codifica per Nformil-metionina (in E. coli)
il tRNA con l’anticodone complementare
al codone esposto nel sito A vi si lega,
essendo già associato al proprio
aminoacido (aminoacil-tRNA)
Sul sito P è presente un tRNA entrato in
precedente, cui è legato il nascente
polipeptide (peptidil-tRNA);il
polipeptide si stacca dal tRNA sul sito P
e si lega all’aminoacido legato al tRNA
sul sito A
Il tRNA libero
sul sito P si
allontana, il
codone e il
peptidil-tRNA
sul sito A si
spostano sul sito
P
Quando sul sito A giunge un codone di
terminazione, al posto di un tRNA vi si lega
un Fattore di Rilascio che impedisce
l’allungamento del poipeptide e conclude il
processo


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)







![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)