PCR
Polymerase Chain Reaction
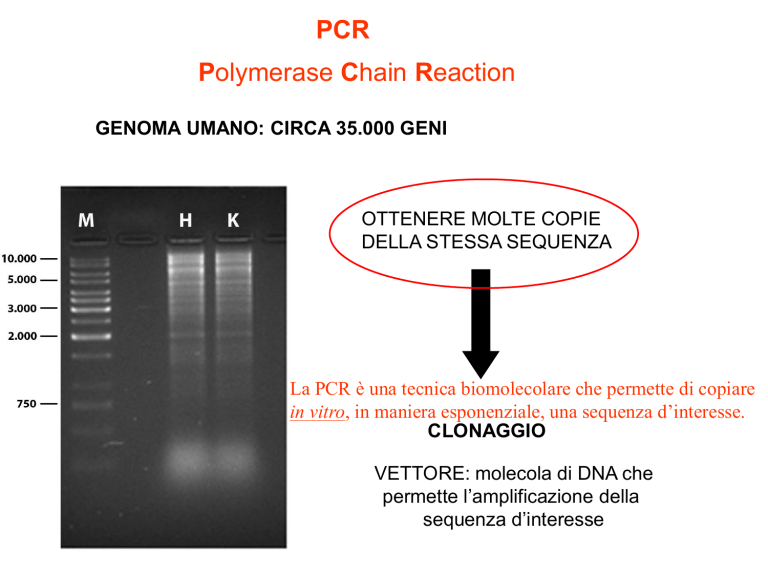
GENOMA UMANO: CIRCA 35.000 GENI
OTTENERE MOLTE COPIE
DELLA STESSA SEQUENZA
La PCR è una tecnica biomolecolare che permette di copiare
in vitro, in maniera esponenziale, una sequenza d’interesse.
CLONAGGIO
VETTORE: molecola di DNA che
permette l’amplificazione della
sequenza d’interesse
PCR
Miscela composta da:
- Stampo
- DNA pol
- dNTP
- Tampone
- MgCl2
Replicazione del DNA
5’-AAGTCGCCGTAAATGCGTACTGTTGCATGCGTAAATGCGCCCTGAAATCGATT-3’
3’-TTCAGCGGCATTTACGCATGACAACGTACGCATTTACGCGGGACTTTAGCTAA-5’
Filamento stampo
DNA pol
5’-AAGTCGCCGTAAATGCGTACTGTTGCATGCGTAAATGCGCCCTGAAATCGATT-3’
3’-TTCAGCGGCATTTAC GCATGACAACGTACGCATTTACGCGGGACTT TAGCTAA-5’
Innesco (primer)
Innesco (primer)
5’-AAGTCGCCGTA AATGCGTACTGTTGCA TGCGTAAATGCGCCC TGAAATCGATT-3’
3’-TTCAGCGGCATTTACGCATGACAACGTACGCATTTACGCGGGACTTTAGCTAA-5’
DNA pol
Filamento stampo
POLIMERASI
DNA polimerasi usate nella PCR: DNA polimerasi DNA-dipendente
Nelle prime versioni della tecnica si utilizzava la polimerasi termolabile DNA pol I di E. Coli.
oggi si utilizzano polimerasi termostabili
Taq polimerasi: estratta del batterio termofilo Thermus aquaticus
non ha proofreading (3’-5’ esonucleasi)
(correzione di bozza)
bassa fedeltà
Pfu polimerasi: estratta dal batterio ipertermofilo Pyrococcus furiosus
presenta proofreading (3’-5’ esonucleasi)
(correzione di bozza)
alta fedeltà
INNESCHI
Inneschi (primers): Le DNA polimerasi hanno bisogno di una doppia elica di DNA,
l’appaiamento dell’innesco con lo stampo fornisce la doppia elica e
l’estremità 3’ OH a cui aggiungere i nucleotidi.
Progettazione degli inneschi (primers): definizione della loro sequenza.
È tra i fattori che maggiormente influenzano l’efficienza della reazione.
5’-AAGTCGCCGTAAATGCGTACTGTTGCATGCGTAAATGCGCCCTGAAATCGATT-3’
3’-TTCAGCGGCATTTACGCATGACAACGTACGCATTTACGCGGGACTTTAGCTAA-5’
5’-AAGTCGCCGTAAATGCGTACTGTTGCATGCGTAAATGCGCCCTGAAATCGATT-3’
3’-AACGT-5’
3’-AGCTA-5’
5’-TCGCC-3’
5’-GTAAA-3’
3’-TTCAGCGGCATTTACGCATGACAACGTACGCATTTACGCGGGACTTTAGCTAA-5’
Caratteristiche degli inneschi
Specificità: si ottiene quando una coppia di inneschi (primers) si lega in modo
stabile esclusivamente alle sequenze complementari
Innesco1: TC
Innesco2: AC
5’-AAGTCGCCGTAAATGCGTACTATTGCATGCCTCAATGCGCCCTTAAATGGGTT-3’
CA
CA
TC
TC
3’-TTCAGCGGCATTTACGCATGATAACGTACGGAGTTACGCGGGAATTTACCCAA-5’
PRODOTTO SPECIFICO
PRODOTTO ASPECIFICO
La dimensione media degli inneschi si aggira intorno ai 20 nucleotidi
Composizione delle basi
Contenuto in G + C compreso tra 40 e 60%
Lunghezza
Tra i 18 e 25 nucleotidi
Sequenza
No sequenze complementari nello stesso primer
superiori a 3 basi
Complementarietà tra i due
inneschi
Evitare la formazione di dimeri tra i due primers
dovuti a sequenze complementari
Estremità degli oligonucleotidi
Se possibile l’estremità 3’ deve essere una C o una G
Temperatura di fusione (melting temperature)
Tm (°C): 2(A + T) + 4(G + C)
Valida per primers di 15-20 basi in soluzioni
ad alta forza ionica (1 M NaCl)
Tm (°C): 81,5 °C + 16,6 (log10[K+]) + 0,41 (%[G+C]) – 675/n
n: numero delle basi del nucleotide
Valida per primers di 14-70 in soluzioni a concentrazioni cationiche
di 0,4 M o meno
STAMPO
DNA genomico
Es: studio di un gene o del promotore
DNA genomico virale
(diagnosi molecolare)
Es: infezione da HPV (Human Papilloma Virus)
cDNA
DNA complementare
cDNA
RNA
retrotrascrizione
Es: profilo di espressione genica
Gli altri elementi essenziali
Tampone: il più usato è il Tris-HCl che a temperatura ambiente stabilizza il pH della
miscela di reazione intorno a 8,3-8,8.
Deossiribonucleosidi trifosfato (dNTP): sono i mattoni di cui è costituito il DNA
Cationi bivalenti: richiesti dall’enzima per svolgere la propria azione (Mg2+)
FASI DELL’AMPLIFICAZIONE
VENGONO RIPETUTE CICLICAMENTE
APPAIAMENTO
DENATURAZIONE
ALLUNGAMENTO
(ANNEALING)
5’
3’
APPAIAMENTO
DENATURAZIONE
ALLUNGAMENTO
(ANNEALING)
3’
5’
3’
5’
5’
3’
APPAIAMENTO
ALLUNGAMENTO
DENATURAZIONE
(ANNEALING)
5’
3’
3’
5’
3 FASI
DENATURAZIONE
ALLUNGAMENTO
APPAIMENTO
~ 30 CICLI
DENATURAZIONE
APPAIAMENTO (ANNEALING)
POLIMERIZZAZIONE
94 °C
~ 55 °C
72 °C
3’
5’
3’
5’
5’
3’
3’
5’
3’
5’
5’
3’
5’
3’
3’
5’
5’
3’
5’
3’
4 COPIE
2 COPIE
3’
5’
3’
5’
5’
3’
5’
3’
3’
5’
STAMPO
3’
5’
8 COPIE
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
5’
3’
5’
3’
3’
5’
3’
5’
5’
3’
RT-PCR
Reverse Transcription Polymerase Chain Reaction
Si parte dall’RNA (totale o mRNA polyA+)
RT
retrotrascrizione
cDNA
amplificazione della sequenza
di interesse
PCR
amplicone (prodotto di amplificazione)
RETROTRASCRIZIONE
Si basa sull’azione delle Trascittasi inverse, cioè di DNA polimerasi RNA-dipendente,
polimerasi che usano uno stampo di RNA per produrre una copia di DNA.
Le Trascrittasi inverse sono polimerasi che derivano dai retrovirus (virus ad RNA),
che le usano per retrotrascivere il loro genoma.
Essendo DNA polimerasi hanno bisogno di un innesco per iniziare la sintesi di DNA
Nelle reazioni in vitro si usano oligonucleotidi d’innesco sequenza-specifici
oppure una collezione di inneschi con sequenza random (casuale).
Nel caso di inneschi sequenza-specifici viene retrotrascritto solo l’RNA d’interesse
Nel caso della miscela di inneschi random vengono retrotrascritti tutti gli RNA
INNESCHI RAMDOM (CASUALI)
2 NUCLEOTIDI
A T G C
3 NUCLEOTIDI
AA
AT
AG
AC
AAA
AAT
AAG
AAC
ATA
ATG
ATC
AGA
AGT
...
TT
TA
TG
TC
GG
GA
GT
GC
TTT
TTA
TTG
TTC
TAT
TGT
TCT
TGG
TAC
...
CC
CA
CT
CG
...
...
In genere si utilizzano oligonucleotidi d’innesco di circa 8 nucleotidi (pdn8)
Innesco specifico
RNA d’interesse
5’-auucacccaaggcguguucagacuugggccacacguaccccaaauuaaaaaaaaaaa-3’
3’-tgcatggggttta-5’
Inneschi random
Inneschi random
Miscela RNA
retrotrascrizione
PCR con inneschi
sequenza-specifici
Miscela di cDNA
PROFILO TEMPORALE DI ESPRESSIONE GENICA
Zebrafish (Danio rerio)
BLASTULA → GASTRULA → SOMITOGENESI → PHARYNGULA (inizio) → PHARYNGULA (fine) → LARVA
ESTRAZIONE RNA
RETROTRASCRIZIONE
cDNA
PCR con inneschi gene-specifici
CORSA ELETTROFORETICA SU GEL DI AGAROSIO
Donizetti et al., 2008








![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
