CARATTERISTICHE VETTORE PLASMIDICO DI CLONAGGIO
- Origine autonoma di replicazione (ORI)
- Geni per la selezione: resistenza ad uno o due antibiotici
- Altri geni per la selezione del plasmide ricombinante
- Siti multiplo di clonaggio
oppure
elettroporazione
T. A. Brown, BIOTECNOLOGIE MOLECOLARI, Zanichelli editore S.p.A Copyright © 2007
Piastramento
su terreno con antibiotico
T. A. Brown, BIOTECNOLOGIE MOLECOLARI, Zanichelli editore S.p.A Copyright © 2007
Selezione per la presenza
del vettore: resistenza ad
antibiotico
T. A. Brown, BIOTECNOLOGIE MOLECOLARI, Zanichelli editore S.p.A Copyright © 2007
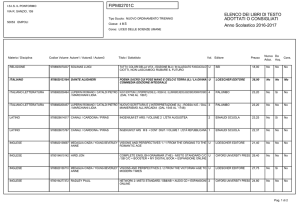
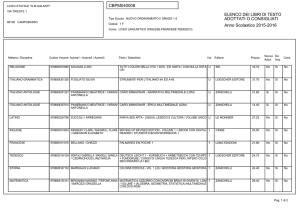
VETTORI DI CLONAGGIO PER E. coli
Tipo
Dimensioni Dimensioni Sistema
inserto (kb) selezione
(kb)
Metodo
introduzione
Plasmidi
pBR322
4,3
6
pUC
2,1
8
Batteriofago lambda
25
6,4
3
Cosmidi
5-6
35-50
7
Amp + Lac Z
20-25
Fago filamentoso M13
Cromosomi artificiali
BAC
2 Antibiotici
fino a 300
Trasformazione
Trasformazione
Infezione
Lac Z
Antibiotico
Trasfezione
Infezione
Amp (CM) + Lac Z Elettroporazione
Plasmide pBR322
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
pBR322: screening delle
colonie ricombinanti
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Vettori plasmidici (pUC18)
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Il gene LacZ codifica per il polipeptide
β-galattosidasi (1021 a.a.)
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
•Ceppo di E.coli: gene Lac Z
mutante: peptide parziale
Complementano
•Vettore pUC: gene LacZ
mutante: peptide parziale
•Ceppo di E.coli Lac Z mutante
•Vettore LacZ interrotto dal
clonaggio
Non c’è
complementazione
β-galattosidasi
ATTIVA
Metabolizzato
Terreno + X-Gal
β-galattosidasi
Non metabolizzato
INATTIVA
β-galattosidasi
ATTIVA
β-galattosidasi
INATTIVA
Colonie
Blu
Colonie
bianche
X-Gal: 5-Bromo-4-Cloroindolil-β-galattoside (incolore)
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
(induttore)
Metodi di inserimento dei vettori ricombinanti nelle
cellule batteriche
Trasformazione: plasmidi (DNA) trasformano cellule procariotiche rese
competenti
Infezione: fagi (DNA con capsidi) che lisano il batterio
Trasduzione: cosmidi (DNA con capsidi) che nel batterio si comportano
come plasmidi
Trasfezione: come una trasformazione ma con DNA fagico senza capsidi
per es: per introdurre la forma replicativa di M13 a doppio filamento
BATTERIOFAGO LAMBDA COME VETTORE DI CLONAGGIO
Lambda w.t. = 50 Kb
Estremità “cos” di 12 nt
per circolarizzazione
Infezione di E. coli
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Batteriofago lambda
Batteriofago lambda
Lambda w.t. = 50 Kb
Lambda di sostituzione: eliminati geni ciclo lisogenico, inserti fino a 23 Kb
Clonaggio di frammenti DNA esogeno e packaging in vitro
Infezione
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Creati siti unici per
clonaggio
Formazione di catenani
T. A. Brown, BIOTECNOLOGIE MOLECOLARI, Zanichelli editore S.p.A Copyright © 2007
Fago difettivo (siti cos alterati)
non può replicarsi ma produce
proteine per i capsidi che possono
essere purificate
Fagi difettivi che da soli non
producono capsidi.
Le singole proteine possono
essere purificate
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
CLONAGGIO IN BATTERIOFAGI
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Screening placche di lisi
Infezione fagica
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Fago filamentoso
La cellula
batterica non
viene lisata!
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Fago filamentoso
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Clonaggio in cosmidi
Formazione di catenani
Formazione di colonie
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
CROMOSOMI ARTIFICIALI DI BATTERIO (BAC)
Fattore di fertilità F di E. coli ha 3 geni (parA, parB, repE) per la
replicazione e per mantenere basso il numero di copie del fattore
(1- 2 per cellula)
Gene per la resistenza al cloramfenicolo (per selezione trasformanti)
Siti di clonaggio nel gene LacZ (identificazione ricombinanti per
complementazione)
Inserto tra 150 e 350 Kb
Integrazione batterica per elettroporazione
Promotori per la trascrizione (T7 e SP6)
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
VETTORI DI CLONAGGIO IN LIEVITO:
YAC (YEAST ARTIFICIAL CHROMOSOME)
Saccharomyces cerevisiae: 16 cromosomi da 250 kb a 2 Mb
Inserto: 300 kb – 1 Mb
Sferoplasting: liticasi
Griffiths et al., GENETICA 6/E, Zanichelli Editore S.p.A. Copyright © 2006
SUP 4
CEN = Centromero
ARS = sequenze autonome di replicazione
TRP1 e URA3 per selezione di molecole con entrambi i bracci
URA 3 = gene per un enzima della sintesi dei nucleotidi pirimidinici
TRP1 = gene per un enzima della sintesi del triptofano
SUP 4 = gene per il tRNA per la tirosina che sopprime la mutazione non
senso “ochre” nel gene ADE2 del ceppo di lievito
ADE2 = gene per la sintesi di adenina: un precursore (fosforibosil amminoimidazolo) che
sta a monte nella via biosintetica si accumula e dà colonie rosse
S. cerevisiae (w.t): URA (-), TRP (-), ADE2 (-) per mutazione “ochre”.
Nel codone UAU (tyr) nel gene ADE2 si verifica la mutazione “ochre” UAA “non
senso” e diventa ADE2(-): l’adenina non viene sintetizzata e si accumula come
premetabolita rosso
S. cerevisiae (w.t): crescono se nel in terreno c’è uracile e triptofano
S. cerevisiae (w.t): URA (-), TRP (-), ADE2 (-) per mutazione “ochre”
accumulo di premetabolita dell’adenina
colonie rosse
YAC con gene SUP 4 = gene per il tRNA con UAA Tyr*
SUP 4 integro sopprime la mutazione non senso “ochre” nel gene ADE2 del
ceppo di lievito: viene sintetizzata adenina, non c’è accumulo di
premetabolita rosso
colonie bianche
S. cerevisiae (terreno minimo) + YAC senza inserto (SUP attivo)
colonie bianche
colonie bianche
S. cerevisiae (terreno minimo) + YAC con inserto (SUP inattivo)
colonie rosse
VANTAGGI E SVANTAGGI DEL CLONAGGIO IN YAC:
- Chimerismo (co-saldatura di più regioni nello stesso YAC)
- Instabilità (soprattutto a livello di regioni ripetute)
- Co-trasformazione (più di uno YAC per cellula di lievito)
Distribuzione dei cloni in griglie
Produzione di filtri con i singoli cloni da utilizzare in screening
Allineamento dei cloni in contigui
ALLINEAMENTO DI YAC PER CONTENUTO DI STS