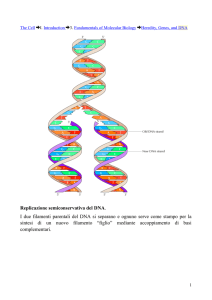
LEZIONE III
DUPLICAZIONE DEL DNA
Dott. Paolo Cascio
DUPLICAZIONE DEL DNA NEI PROCARIOTI
LA DUPLICAZIONE DEL CROMOSOMA BATTERICO CIRCOLARE INIZIA IN
UN UNICO PUNTO E PROCEDE BIDIREZIONALMENTE FINO A QUANDO
TUTTO IL CROMOSOMA NON E’ STATO COPIATO. LA ZONA IN CUI LA
PARTE NEOSINTETIZZATA INCONTRA QUELLA NON DUPLICATA E’
DEFINITA FORCELLA DI DUPLICAZIONE.
LA REPLICAZIONE INIZIA IN UN PUNTO DI ORIGINE FISSO E PROCEDE IN
DIREZIONI OPPOSTE FINO A CHE LE FORCELLE DI REPLICAZIONE SI
INCONTRANO.
IN E. COLI SONO STATI SCOPERTI TRE ENZIMI CHE POSSONO
CATALIZZARE LA SINTESI SU STAMPO DEL DNA (DNA POLIMERASI I,
DNA POLIMERASI II, DNA POLIMERASI III).
DNA POLIMERASI I:
RIPARA LE LESIONI SUBITE DALLA CATENA DI DNA.
DNA POLIMERASI II:
IL SUO RUOLO IN VIVO NON E’ ANCORA NOTO.
DNA POLIMERASI III:
E’ RESPONSABILE DELL’ALLUNGAMENTO DELLA CATENA DEL DNA
DURANTE LA DUPLICAZIONE.
LA DNA POLIMERASI III ESISTE IN UNA FORMA PIU’ SEMPLICE (CON
ATTIVITA’ MINIMA) FORMATA DA TRE SUBUNITA’ (α, ε, θ) E IN UNA
FORMA MOLTO PIU’ ATTIVA (NOTA COME OLOENZIMA) FORMATA
DALL’ASSOCIAZIONE
DI
DUE
COMPLESSI
MULTIMERICI
DI
13
SUBUNITA’ CIASCUNO.
L’ENZIMA SINTETIZZA IL DNA AGGIUNGENDO UN NUCLEOTIDE PER
VOLTA ALL’ESTREMITA 3’ DELLA CATENA IN ACCRESCIMENTO. IL
NUCLEOTIDE AGGIUNTO DERIVA DA UN DESOSSIRIBONUCLEOSIDE-5’TRIFOSFATO (dNTP). PER QUESTA REAZIONE E’, INOLTRE, NECESSARIO
Mg++, POICHE’ I dNTP PER POTER ESSERE UTILIZZATI DEVONO ESSERE
COMPLESSATI CON IL MAGNESIO.
LO SCHELETRO ZUCCHERO-FOSFATO SI FORMA PER TRASFERIMENTO
DI UN GRUPPO NUCLEOTIDICO DA UN dTNP AL GRUPPO OSSIDRILICO 3’
DEL RESIDUO NUCLEOTIDICO TERMINALE DELLA CATENA DI DNA IN
ALLUNGAMENTO. L’IDROLISI DEL PIROFOSTATO (PP) AD OPERA DI UNA
PIROFOSFATASI FORNISCE L’ENERGIA NECESSARIA ALLA REAZIONE.
L’ENZIMA OPERA AD UNA VELOCITA’ DI CIRCA 1000 RESIDUI
NUCLEOTIDICI AL SECONDO.
TEORICAMENTE LA DNA-POLIMERASI III POTREBBE AGIRE CON DUE
MECCANISMI DIVERSI:
1) DOPO CHE UN NUOVO RESIDUO E’ STATO AGGIUNTO ALLA CATENA,
L’ENZIMA POTREBBE STACCARSI E LEGARSI A CASO SU DI UN’ALTRA
CATENA INCOMPLETA (PROCESSO DI SINTESI DEFINITO DISTRIBUTIVO).
OPPURE
2) UNA VOLTA CHE LA POLIMERASI HA INIZIATO LA SINTESI DEL DNA
SU UN FILAMENTO CHE FUNGE DA STAMPO, RIMANE LEGATA AD ESSO
FINCHE’
TALE
FILAMENTO
NON
VIENE
COMPLETAMENTE
REDUPLICATO (PROCESSO DEFINITO PROGRESSIVO).
SPERIMENTALMENTE SI E’ POTUTO DIMOSTRARE CHE IL MECCANISMO
D’AZIONE DELLA DNA-POLIMERASI III (VEDI ESPERIMENTO) E’
PROGRESSIVO. QUANDO IN VIVO L’ENZIMA SI ASSOCIA AD UN
CROMOSOMA PROBABILMENTE NON SE NE DISSOCIA FINO A QUANDO
L’INTERO CROMOSOMA NON E’ STATO REDUPLICATO.
LA
PROGRESSIVITA’
DELLA
DNA-POLIMERSAI
III
E’
DOVUTA
ALL’AZIONE CONCOMITANTE DI VARIE SUBUNITA’ DELL’OLOENZIMA:
1) LA SUBUNITA’ α CATALIZZA IL TRASFERIMENTO DI UN GRUPPO
NUCLEOTIDICO IN DIREZIONE 5’ → 3’.
2) LA SUBUNITA’ β AGGANCIA
SALDAMENTE L’OLOENZIMA ALLA
CATENA DI DNA.
ALTRE PROTEINE COLLABORANO, INOLTRE, CON LA DNA-POLIMERASI
III.
1) LE
ELICASI
CATALIZZANO
LO SVOLGIMENTO DELL’α-ELICA A
LIVELLO DELLE FORCELLE DI DUPLICAZIONE.
2) LA PROTEINA CHE SI LEGA AD UN SINGOLO FILAMENTO (SSB)
IMPEDISCE AI FILAMENTI DI DNA DENATURATI DI RIFORMARE L’αELICA O DELLE ANSE A FORCINA (CHE ARRESTEREBBERO L’AZIONE
DELLA POLIMERASI).
LA DNA-POLIMERSAI III E’, INOLTRE, CAPACE DI CORREGGERE GLI
ERRORI SULLA CATENA DI DNA IN FORMAZIONE CAUSATI DA UN NON
CORRETTO ACCOPPIAMENTO DELLE BASI. QUESTO E’ POSSIBILE
PERCHE’ LA SUBUNITA’ ε POSSIEDE UN’ATTIVITA’ ESONUCLEASICA 3’
→ 5’ CHE IDROLIZZA IL LEGAME FOSFODIESTERE TRA IL RESIDUO
TERMINALE E IL RESTO DELLA CATENA. L’OLOENZIMA INCORPORA
UNA BASE SBAGLIATA CIRCA UNA VOLTA OGNI 10.000 REAZIONI DI
ALLUNGAMENTO (TASSO DI ERRORE 10-4). QUESTI ERRORI VENGONO
CORRETTI DALL’ATTIVITA’ ESONUCLEASICA DELLA SUBUNITA ε CHE,
A SUA VOLTA, HA UN TASSO DI ERRORE DI 10-3. LA COMBINAZIONE DI
QUESTE
DUE
REAZIONI
SEQUENZIALI
PRODUCE
UN
TASSO
COMPLESSIVO DI ERRORE DI 10-7 (UNO DEI PIU’ BASSI MAI
RISCONTRATI PER UN ENZIMA). QUINDI ENORMI MOLECOLE DI DNA
VENGONO REDUPLICATE CON POCHISSIMI ERRORI.
LA
DNA-POLIMERSAI
III
CATALIZZA
L’ALLUNGAMENTO
DELLA
CATENA DI DNA SOLO IN DIREZIONE 5’ → 3’. TUTTAVIA, UN ESAME
DELLA FORCELLA DI DUPLICAZIONE RIVELA CHE LA SINTESI 5’ → 3’
PUO’ ESSERE CONTINUA SOLO SU DI UN FILAMENTO. NELL’ALTRO
FILAMENTO, CHE HA UNA POLARITA’ OPPOSTA, LA SINTESI 5’ → 3’
PROCEDE IN DIREZIONE OPPOSTA RISPETTO ALLA FORCELLA
DI
DUPLICAZIONE.
IL NUOVO FILAMENTO FORMATOSI PER POLIMERIZZAZIONE 5’ → 3’
NELLA DIREZIONE DELLA FORCELLA E’ CHIAMATO FILAMENTO GUIDA.
L’ALTRO FILAMENTO, FORMATOSI PER POLIMERIZZAZIONE 5’ → 3’ IN
DIREZIONE OPPOSTA ALLA FORCELLA E’ CHIAMATO FILAMENTO
LENTO.
IL
FILAMENTO
SINTETIZZATO
LENTO
IN
DEVE
FRAMMENTI,
NECESSARIAMENTE
CIASCUNO
DEI
ESSERE
QUALI
VIENE
POLIMERIZZATO IN DIREZIONE 5’ → 3’. SOLO IN UN SECONDO
MOMENTO I FRAMMENTI NEOSINTETIZZATI VENGONO LEGATI TRA
LORO A FORMARE UN FILAMENTO COMPLETO (SINTESI DISCONTINUA
DEL DNA).
LA PROVA DELLA SINTESI DISCONTINUA DEL DNA E’ STATA OTTENUTA
MARCANDO
IL
ESAMINANDO,
DNA
POI,
NEOSINTETIZZATO
LA
STRUTTURA
CON
3H-TIMIDINA
ED
DEGLI
INTERMEDI
DI
DUPLICAZIONE.
LA TIMIDINA TRIZIATA VIENE SOMMINISTRATA PER UN BREVE
PERIODO ALLE CELLULE DI E. COLI IN FASE DI DUPLICAZIONE.
LA SINTESI DEL DNA VIENE BLOCCATA A DIVERSE RIPRESE E IL DNA
NEOSINTETIZZATO
VIENE
ISOLATO.
LE
MOLECOLE
DI
DNA
NEOSINTETIZZATO SONO DI DUE TIPI: MOLECOLE DI DNA MOLTO
GRANDI (CONTENETI CIRCA META’ DELLA RADIOATTIVITA’ TOTALE) E
FRAMMENTI DI 1000-2000 NUCLEOTIDI (CONTENETI L’ALTRA META’
DELLA RADIOATTIVITA’).
LE MOLECOLE GRANDI DI DNA DERIVANO DALLA SINTESI CONTINUA
DEL FILAMENTO GUIDA. I FRAMMENTI PIU’ PICCOLI, INVECE,
DERIVANO DALLA SINTESI DISCONTINUA DEL FILAMENTO LENTO.
QUESTI PICCOLI FRAMMENTI DI DNA SONO CHIAMATI FRAMMENTI DI
OKAZAKI.
ESISTE, PERO’, UN ULTERIORE PROBLEMA. NESSUNA DNA-POLIMERASI
NOTA E’ IN GRADO DI INIZIARE LA POLIMERIZZAZIONE DEL DNA EX
NOVO, MA TUTTE RICHIEDONO PER POTER AGIRE LA PRESENZA DEL
GRUPPO OSSIDRILICO 3’ DI UN CORTO RNA INNESCO. PERCIO’ LA
SINTESI DI TUTTI FRAMMENTI DI OKAZAKI (COSI’ COME DEL
FILAMENTO GUIDA) COMINCIA CON QUELLA DI UN RNA INNESCO 5’ →
3’ AL QUALE LA DNA-POLIMERASI III PUO’, POI, AGGIUNGERE
DESOSSIRIBONUCLEOTIDI.
L’RNA INNESCO VIENE SINTETIZZATO DA UN ENZIMA DETTO PRIMASI
(O dnaG) CHE SINTETIZZA UN RNA INNESCO DI 1-3 NUCLEOTIDI AL
SECONDO. POICHE’ LA FORCELLA DI DUPLICAZIONE SI MUOVE AD UNA
VELOCITA’ DI CIRCA 1000 NUCLEOTIDI AL SECONDO, LA PRIMASI
PRODUCE UN INNESCO OGNI 1000 NUCLEOTIDI.
A MANO A MANO CHE ALTRO DNA A FILAMENTO SINGOLO COMPARE
DIETRO AD ESSA, LA PRIMASI AVANZA CON LA FORCELLA DI
DUPLICAZIONE E SINTETIZZA NUOVI INNESCHI PER NUOVI FRAMMENTI
DI OKAZAKI.
LA PRIMASI E’ UN COMPONENTE DEL PRIMOSOMA, CHE E’ UN
COMPLESSO MULTIMERICO CHE OPERA A LIVELLO DELLA FORCELLA
DI DUPLICAZIONE. IL PRIMOSOMA CONTIENE ALMENO 17 POLIPEPTIDI
DIFFERENTI, TRA I QUALI (OLTRE ALLA PRIMASI) 6 MOLECOLE DI dnaB
E 6 MOLECOLE DI dnaC. QUESTI DUE ENZIMI SONO DELLE ELICASI CHE
SROTOLANO E SEPARANO I DUE FILAMENTI DELL’α-ELICA.
UNA VOLTA CHE I FRAMMENTI DI OKAZAKI SONO STATI SINTETIZZATI,
GLI RNA INNESCO VENGONO IDROLIZZATI SIA DALLA RNASI H CHE
DALLA COMPONENTE ESONUCLEASICA DELLA DNA-POLIMERASI I. A
QUESTO PUNTO IL FILAMENTO LENTO E’ COSTITUITO DA MOLTI
FRAMMENTI DI OKAZAKI SEPARATI DA DISCONTINUITA’ DOVUTE
ALL’ASSENZA DI ALCUNI NUCLEOTIDI (DOVE PRIMA ERANO PRESENTI
GLI RNA INNESCO).
LA DNA-POLIMERASI I POSSIEDE DIVERSE ATTIVITA’:
ATTIVITA’ ESONUCLEASICA 5’ → 3’ RIMUOVE L’RNA INNESCO
ALL’ESTREMITA’ DI CIASCUN FRAMMENTO DI OKAZAKI (ESITE ANCHE
UN’ATTIVITA’ ESONUCLEASICA 3’ → 5’ CHE CORREGGE IL FILAMENTO
DI DNA QUALORA VENGANO INSERITI NUCLEOTIDI ERRONEAMENTE
APPAIATI).
ATTIVITA’ POLIMERASICA 5’ → 3’ RIEMPIE LE LACUNE TRA I
FRAMMENTI DI OKAZAKI RIMASTE DOPO L’IDROLISI DEGLI RNA
INNESCO.
IL PROCESSO MEDIANTE IL QUALE LA DNA-POLIMERASI I SOSTITUISCE
GLI RNA INNESCO CON DNA VIENE DEFINITO TRASLAZIONE DELLE
DISCONTINUITA’.
STRUTTURA CRISTALLINA DI UN FRAMMENTO DI DNA-POLIMERASI I.
L’ENZIMA POSSIEDE UNA FENDITURA LARGA 2 nm E RIVESTITA DA
CATENE LATERALI DI AMINO ACIDI CON CARICA POSITIVA CHE
POSSONO LEGARE IL DNA. IL FILAMENTO STAMPO VIENE COSI’ A
TROVARSI COMPLETAMENTE INSERITO ALL’INTERNO DI QUESTA
FENDITURA.
LO STADIO FINALE NELLA MATURAZIONE DEL FILAMENTO LENTO DI
DNA APPENA SINTETIZZATO E’ LA SALDATURA DEI FRAMMENTI DI
OKAZAKI DA PARTE DELL’ENZIMA DNA-LIGASI. QUESTO ENZIMA
CATALIZZA LA FORMAZIONE DI UN LEGAME FOSFODIESTERE TRA IL
GRUPPO OSSIDRILICO 3’ DELL’ESTREMITA’ DI UN FRAMMENTO DI
OKAZAKI E IL GRUPPO FOSFATO 5’ DEL FRAMMENTO DI OKAZAKI
ADIACENTE.
IN VIVO LA SINTESI DEL FILAMENTO GUIDA E DI QUELLO LENTO
AVVENGONO
APPROSSIMATIVAMENTE
ALLA
STESSA
VELOCITA’,
POICHE’ I DUE PROCESSI DI REDUPLICAZIONE SONO FISICAMENTE
ACCOPPIATI. LE PROTEINE COINVOLTE NELLA SINTESI DEI DUE
FILAMENTI
SI
ASSOCIANO,
INFATTI,
A
FORMARE
UN
UNICO
COMPLESSO PROTEICO, IL REPLISOMA.
UN REPLISOMA PRESENTE NELLA FORCELLA DI DUPLICAZIONE
RISULTA PERCIO’ COSTITUITO DA:
2 MOLECOLE DI DNA-POLIMERASI III (UNA PER IL FILAMENTO GUIDA E
UNA PER QUELLO LENTO).
1 PRIMOSOMA COSTITUITO DALLA PRIMASI (CHE SINTETIZZA GLI RNA
INNESCO) E DALLE ELICASI (dnaB E dnaC).
1 PROTEINA REP CHE SVOLGE LA STESSA FUNZIONE DI dnaB (CIOE’
SROTOLARE L’α-ELICA) MA PER IL FILAMENTO GUIDA.
4 PROTEINE SSB CHE SI LEGANO AL FILAMENTO SINGOLO CHE FA DA
STAMPO PER IL FILAMENTO LENTO E GLI IMPEDISCONO DI RIFORMARE
L’α-ELICA O ANSE A FORCINA.
VARIE DNA-TOPOISOMERASI CHE ALLENTANO LA TENSIONE CHE SI
ACCUMULA A MONTE DELLA FORCELLA PER LO SROTOLAMENTO
DELL’α-ELICA
UN REPLISOMA PRESENTE NELLA FORCELLA DI DUPLICAZIONE
RISULTA PERCIO’ COSTITUITO DA:
2 MOLECOLE DI DNA-POLIMERASI III (UNA PER IL FILAMENTO GUIDA E
UNA PER QUELLO LENTO).
1 PRIMOSOMA COSTITUITO DALLA PRIMASI (CHE SINTETIZZA GLI RNA
INNESCO) E DALLE ELICASI (dnaB E dnaC).
1 PROTEINA REP CHE SVOLGE LA STESSA FUNZIONE DI dnaB (CIOE’
SROTOLARE L’α-ELICA) MA PER IL FILAMENTO GUIDA.
4 PROTEINE SSB CHE SI LEGANO AL FILAMENTO SINGOLO CHE FA DA
STAMPO PER IL FILAMENTO LENTO E GLI IMPEDISCONO DI RIFORMARE
L’α-ELICA O ANSE A FORCINA.
VARIE DNA-TOPOISOMERASI CHE ALLENTANO LA TENSIONE CHE SI
ACCUMULA A MONTE DELLA FORCELLA PER LO SROTOLAMENTO
DELL’α-ELICA
LA DUPLICAZIONE DEL DNA BATTERICO HA INIZIO IN UN SITO UNICO
DETTO SITO DI ORIGINE DELLA DUPLICAZIONE (oriC IN E. COLI). oriC
CONSISTE IN UNA SEQUENZA DI 245 NUCLEOTIDI ALTAMENTE
CONSERVATI IN QUASI TUTTI I BATTERI STUDIATI (QUINDI QUESTO
SITO SI E’ CONSERVATO DURANTE L’EVOLUZIONE).
oriC PRESENTA QUATTRO SITI DI LEGAME CHE ALL’INIZIO DEL
PROCESSO DI REDUPLICAZIONE DEL DNA INTERAGISCONO CON dnaA
CHE
E’
UNA
PROTEINA
TETRAMERICA
ASSOLUTAMENTE
INDISPENSABILE PER L’AVVIO DELLA REDUPLICAZIONE.
UNA VOLTA CHE LE PRIME 4 dnaA SI SONO LEGATE AD oriC, MOLTE
ALTRE SE NE AGGIUNGONO. SI VIENE COSI’ A FORMARE UNA
STRUTTURA PARTICOLARE CON UN NUCLEO CENTRALE PROTEICO
ATTORNO AL QUALE SI AVVOLGONO DELLE SPIRE DI DNA CHE
DETERMINA UNA PICCOLA DENATURAZIONE DEL DNA.
IN
QUESTA
“BOLLA”
DI
DNA
DENATURATO
ASSEMBLARSI I COMPONENTI DEL REPLISOMA.
COMINCIANO
AD
PER PRIMA COSA LE PRIMASI SINTETIZZANO L’RNA INNESCO PER IL
FILAMENTO GUIDA DELLA FORCELLA OPPOSTA.
IN UN SECONDO MOMENTO LA PRIMASI COMINCIA A SINTETIZZARE
ANCHE L’RNA INNESCO PER IL FILAMENTO LENTO. IN QUESTO MODO
LA REDUPLICAZIONE PROCEDE PER AMBEDUE I FILAMENTI STAMPO IN
ENTRAMBE LE DIREZIONI (QUINDI SONO NECESSARIE 4 MOLECOLE DI
DNA-POLIMERASI III).


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)