Struttura complessa (230 x 300 nm)
Genoma: 1 molecola di DNA ds (100-200 x 106 d)
Virioni molto stabili
(mesi a 4 °C, anni a -20 °C)
Inattivati da solventi dei lipidi
Per vedere questa immagine
occorre QuickTime™ e un
decompressore Photo - JPEG.
Generi:
AVIPOXVIRUS
ENTOMOPOXVIRUS
LEPORIPOXVIRUS
ORTHOPOXVIRUS -Virus del vaiolo
- Virus vaccino*
Fibrille proteiche
1
*modello di studio
EEV
IUV
nucleoide o
esterna
Per membrana
vedere questa
immagine
occorre QuickTime™ e un
decompressorePer
Photo
- JPE
G.
vedere
questa
immagine
occorre QuickTime™ e un
decompressore Photo - JPE G.
ds DNA (
•* Legami fosfodiesterici all’estremita’ di sequenze terminali ripetute invertite
•scissi da endonucleasi virali durante l’infezione
Proteine del core(
Strutturali : proteine basiche associate al DNA
Enzimatiche: RNA polimerasi, Poliadenilato polimerasi, metil-transferasi,
guanilil-transferasi.
Trascritti senza introni. Assenza di splicing
2
Early proteins
DNA polymerase
transcriptional factors
RNA polymerase
Early mRNA
Late mRNA
Late proteins
structural proteins
enzymes
3
Hepadnaviridae
virus dell’Epatite B (HBV)
Per vedere questa immagine
occorre QuickTime™ e un
decompressore Photo - JPEG.
virus pleiomorfo
particella di Dane
(42 nm)
4
Involucro della particella di Dane
Antigene di superficie:
Antigene Australia
HBsAg
5
HEPADNAVIRUS: DNAds circolare incompleto
Filamento - = 3.2 kbp
Filamento + = 50-80% del
filamento (-)
DNA Polimerasi (RT/RNasi H) è contenuta
nei virioni (legata covalentemente al
5’ del filamento di DNA(-))
6
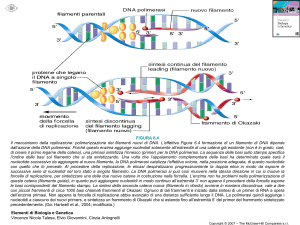
Replicazione di HBV
Replicazione nucleare
e citoplasmatica
RNA < 3.2 kb = mRNA
RNA 3.5 kb = pregenoma
RNA pregenomico
Trascrizione inversa
DNA (-)
7
Particella con involucro (35-50 nm)
Genoma: ssRNA 1.7 kb
Codifica per la proteina del capside (antigene d)
QuickTime™ and a
Photo - JPEG decompressor
are needed to see this picture.
virus satellite di HBV
8