Tecniche analitiche per
l’identificazione/tracciabilità di
specie/varietà di frumento in sfarinati
e prodotti finiti
1
INDICE
PARTE I METODI GENETICO-MOLECOLARI
METODI MOLECOLARI PER L’IDENTIFICAZIONE GENETICA DEI FRUMENTI NELLE FILIRE
AGRO-ALIMENTARI
Premessa ........................................................................................................................................................................... 5
La certificazione .............................................................................................................................................................. 6
Un criterio gerarchico per l’identificazione genetica ............................................................................................... 7
Discriminazione di varietà moderne da varietà antiche ........................................................................................ 8
Discriminazione di varietà a statura bassa da quelle a statura alta ...................................................................... 9
Materiali necessari ..................................................................................................................................................... 10
Pre-requisiti per l'analisi ........................................................................................................................................... 10
Tipo di discriminazione ............................................................................................................................................ 11
Isolamento di marcatori genoma specifici ............................................................................................................... 11
Metodi classici ............................................................................................................................................................ 12
Isolamento di un set di marcatori diagnostici genotipo-specifici........................................................................ 12
L’analisi genomica: aspetti procedurali.................................................................................................................... 14
Metodi di estrazione del DNA................................................................................................................................. 14
Strategie per una determinazione specifica ........................................................................................................... 16
PCR .............................................................................................................................................................................. 16
Amplificazione esponenziale ............................................................................................................................... 17
RAPD........................................................................................................................................................................... 18
AFLP............................................................................................................................................................................ 19
SNP .............................................................................................................................................................................. 20
Microsatelliti (SSR) .................................................................................................................................................... 21
Determinazioni quantitative ...................................................................................................................................... 22
Quantificazione relativa di una varietà contaminante una matrice alimentare................................................ 22
Amplificazione dell’intero genoma........................................................................................................................... 24
REPLI-g ....................................................................................................................................................................... 24
Microchip .................................................................................................................................................................... 25
La tecnologia LiquiChip............................................................................................................................................ 26
Reazione Padlock....................................................................................................................................................... 27
Bibliografia .................................................................................................................................................................... 28
ALLEGATO 1 ................................................................................................................................................................ 29
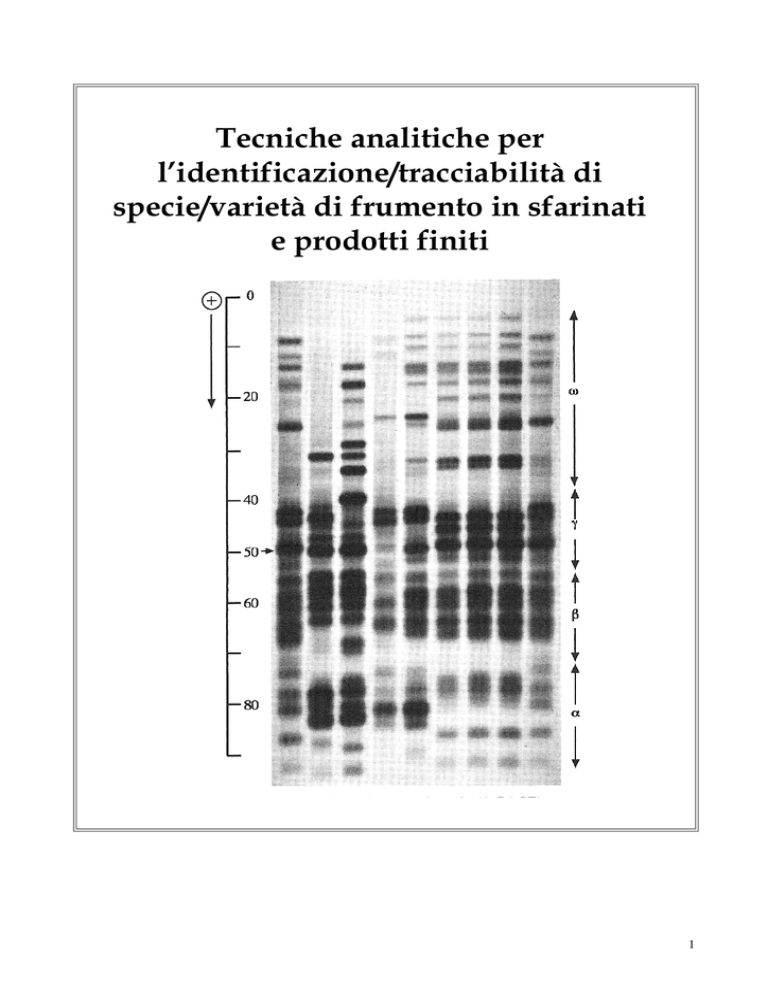
ANALISI DELLE PROTEINE DI RISERVA DEL FRUMENTO DURO ............................................................ 29
ALLEGATO 2 ................................................................................................................................................................ 39
ESTRAZIONE DI DNA DA TESSUTI VEGETALI ............................................................................................... 39
VALUTAZIONE DELLA CONCENTRAZIONE DEL DNA GENOMICO ....................................................... 41
ALLEGATO 3 ................................................................................................................................................................ 43
2
PROTOCOLLO PER L’AMPLIFICAZIONE DI MARCATORI MICROSATELLITI...................................... 43
PROTOCOLLO PER L’AMPLIFICAZIONE DI RAPDs ....................................................................................... 44
ELETTROFORESI ORIZZONTALE SU GEL DI AGAROSIO............................................................................ 45
ELETTROFORESI VERTICALE SU GEL DI ACRILAMMIDE PER SSR DI FRUMENTO DURO ............. 47
ANALISI AFLP ............................................................................................................................................................. 51
CORSA ELETTROFORETICA AL SEQUENZIATORE………………………………………. ………………...55
PARTE II METODI ELETTROFORETICI E IMMUNO CHIMICI
METODI ELETTROFORETICI ED IMMUNO CHIMICI PER IL RICONOSCIMENTO DI SFARINATI
DI FRUMENTO TENERO IN SEMOLE E PASTA DI SEMOLA DI FRUMENTO DURO
Premessa..................................................................................................................................................................... 58
Metodo elettroforetico di Resmini ....................................................................................................................... 59
Metodo elettroforetico per focalizzazione ionica (Metodo Resmini-DeBernardi)...................................... 60
Metodo immunochimico di Cantagalli-Piazzi.................................................................................................... 65
Metodo Kit immunochimico Durotest................................................................................................................. 68
Analisi della frazione sterolica............................................................................................................................... 69
Bibliografia................................................................................................................................................................ 71
ALLEGATO 1............................................................................................................................................................ 72
METODO RESMINI-DE BERNARDI.................................................................................................................. 72
ALLEGATO 2 ........................................................................................................................................................... 73
METODO CANTAGALLI-PIAZZI....................................................................................................................... 73
3
PARTE I
METODI GENETICO-MOLECOLARI
4
METODI MOLECOLARI PER L’IDENTIFICAZIONE GENETICA DEI FRUMENTI
NELLE FILIERE AGRO-ALIMENTARI
Premessa
La selezione massale (in tempi storici) ed il
miglioramento genetico basato sugli incroci
artificiali (ultimo secolo) hanno contribuito alla
formazione di centinaia di varietà di frumento.
Queste varietà si distinguono prevalentemente
in frumenti tetraploidi - tra cui il frumento
duro (Triticum turgidum spp. durum) ed il farro
dicocco (Triticum turgidum spp. dicoccon) - ed in
frumenti esaploidi - frumento tenero (Triticum
aestivum spp. aestivum) e spelta (Triticum
aestivum
spp.
spelta).
Un
rappresentante
importante tra i frumenti diploidi è il farro
monococco
(Triticum
monococcum
spp.
monococcum) (Fig. 1 e 2).
Una varietà coltivata (cultivar) di frumento è
Figura 1. Spighe di differenti varietà di
frumenti teneri (in alto) e duri (in basso)
pertanto una entità tassonomica intraspecifica caratterizzata da un elevato grado di
omozigosi, soprattutto per i geni che controllano i caratteri per i quali è stata effettuata la
selezione. Ciascuna varietà presenta individui uniformi per caratteri morfologici e
produttivi. Ciò nonostante è stato osservato che possono esistere differenze all’interno
della stessa varietà per caratteri biochimici controllati geneticamente (es. componenti delle
proteine di riserva) (Peruffo et al. 1985). Queste varianti sono state definite “biotipi”, per
poter sancire un ordine tassonomico inferiore al rango varietale. La presenza di
eterogeneità intravarietale potrebbe complicare l’identificazione genetica di una varietà
rispetto ad altre se si usasse solo una tipologia di caratteristiche ereditarie.
Lo sviluppo dei marcatori del DNA ha permesso di studiare un numero molto elevato di
loci1 e, tra questi, è stato possibile identificare un sottoinsieme in grado di rintracciare
geneticamente ciascuna varietà coltivata (loci diagnostici) (Donini et al. 1998). Tra questi
1
siti cromosomici in cui è localizzata la sequenza di DNA
5
marcatori i microsatelliti (SSRs2) rappresentano lo strumento più affidabile, robusto e
conveniente oggi a disposizione (Roder et al. 2002).
La certificazione
La certificazione dei prodotti a base di cereali può prevedere che
le singole unità tassonomiche (specie e/o varietà) vengano
distinte mediante analisi standard di laboratorio all’interno dei
prodotti di post raccolta di prima e seconda trasformazione. La
rintracciabilità genetica prevede che in ogni passaggio della
catena di trasformazione dei materiali di base sia possibile
dimostrare l'autenticità della matrice alimentare.
I frammenti di DNA dell'individuo o degli individui che
costituiscono la matrice alimentare rappresentano il bersaglio
per l'identificazione genetica. Ciò nonostante, la distinguibilità
prevede non solo l’identificazione genetica del prodotto
commerciale così come definito su etichetta o su altri tipi di
documenti, ma anche la verifica di eventuali contaminazioni e
misture con altri cereali. Nelle grandi filiere le misture più
probabili sono quelle con prodotti derivanti dalle varietà più
comunemente ed ampiamente coltivate di frumento duro e
tenero. Questo principio vale anche per i cereali di nicchia come
il Kamut, filogeneticamente molto affine al frumento duro, i farri
(monococco, dicocco e spelta) (Fig. 2) avena, orzo e triticale.
Le tecniche più diffuse di laboratorio3 si distinguono in analisi di
marcatori molecolari ed in analisi genetiche di marcatori
biochimici costituiti principalmente dalle proteine di riserva del
seme (gliadine e glutenine).
I marcatori genetici molecolari
consistono nell’evidenza di laboratorio di mutazioni e variazioni
a carico della sequenza del DNA.
2
Simple Sequence Repeats
3
quelle più robuste e maggiormente utilizzate sono descritte in allegato
Figura 2. Varietà di farri:
dicocco (in alto), monococco
(centro) e spelta (in basso)
6
Un criterio gerarchico per l’identificazione genetica
Come accennato, l’identificazione genetica può avvenire a livello di specie, di varietà entro
specie e di biotipo entro varietà (Fig. 3). L’identificazione a livello di specie è relativamente
semplice quando si analizzano i caratteri morfologici perché alcuni sono dei descrittori
specie-specifici. Ad esempio il frumento tenero è prevalentemente mutico mentre il
frumento duro è aristato; così come il farro presenta cariosside vestita rispetto ai frumenti
tenero e duro che presentano cariossidi
nude. Numerosi sono, inoltre, i marcatori
morfologici presi in considerazione dal
sistema di registrazione varietale ufficiale.
Genere
Specie a, Specie b, ecc.
Per quanto riguarda i caratteri genetici
biochimici, le proteine di riserva della
cariosside (gliadine e glutenine) sono state e
Varietà 1, Varietà 2, Varietà 3, ..
continuano ad essere utilizzate con successo
per identificare varietà entro specie e per
Biotipo1, 2, 3, ecc
differenziare individui con diverso grado di Figura 3. Ripartizione gerarchica delle differenze
ploidia (es. misture di semi di frumento genetiche entro e tra unità tassonomiche
duro con frumento tenero) (Lafiandra e Kasarda 1985; Peruffo et al. ??). Purtroppo quest'
analisi è molto efficiente se condotta su singoli chicchi di frumento. Diventa poco efficiente
su prodotti trasformati (es. paste) a causa della denaturazione delle proteine durante le
fasi di trasformazione. La granella rappresenta, quindi, un efficace materiale di partenza
per l’identificazione delle varietà di frumento tramite l’analisi dei profili elettroforetici
delle gliadine e glutenine4.
Come prevedibile, il problema diagnostico si complica quando la granella, ridotta in
sfarinato entra nei processi di trasformazione e produzione di prodotti commerciali (paste,
biscotti, ecc.). In questi casi è necessario isolare una quantità sufficiente di DNA che
diventerà bersaglio di appropriate analisi molecolari. In questi ultimi casi, negli ultimi
anni, sono state sviluppate tecnologie più sensibili, mirate alla identificazione di
4
Le proteine di riserva si distinguono in due grandi gruppi in funzione del tipo di solubilità: Gliadine e Glutenine. Le prime sono a loro volta
frazionate elettroforeticamente in componenti alfa, beta e gamma. Ciascuna di queste componenti è inoltre costituità da singole sub-unità controllate
geneticamente. In modo del tutto simile, le Glutenine si frazionano in due grandi gruppi (gliadine ad alto ed a basso peso molecolare.)
7
frammenti di DNA unici o in mistura, che si possono isolare anche in campioni di modeste
dimensioni (analisi quantitative tramite RT-PCR e Microchip-technology).
In linea del tutto generale, l’analisi molecolare dei frumenti, finalizzata all’identificazione
genetica si basa sul polimorfismo interspecifico dovuto alla presenza di genomi diversi e
sul polimorfismo intraspecifico che si basa sulla variazione della sequenza del DNA
indotta naturalmente. La variazione nella sequenza del DNA è causata da mutazioni
spontanee il cui effetto fenotipico può essere visibile oppure mancare del tutto. A titolo di
esempio, tra le mutazioni con effetto fenotipico visibile si cita quella che ha nanizzato la
statura della pianta, ritenuta tanto utile dal breeder, da essere introdotta tramite gli incroci
in quasi tutte le varietà moderne. Il gene nanizzante (inattivo perché mutato) è raro nelle
antiche varietà e nelle razze locali ma comune nelle moderne varietà. Altre mutazioni a
carico del DNA, che non si esprimono fenotipicamente, sono ad esempio quelle che fanno
variare il numero di unità ripetute di nucleotidi (Simple Sequence Repeats) che prendono il
nome di sequenze microsatelliti (Roder et al. 1995). Queste mutazioni, quasi tutte note e
mappate sui cromosomi del frumento, sono utilizzate anche per scopi di identificazione
varietale e rintracciabilità genomica (Figliuolo e Perrino, 2005).
Discriminazione di varietà moderne da varietà antiche
Disporre di uno strumento in grado di discriminare le varietà moderne da quelle antiche
implica la possibilità di poter identificare la presenza di evidenze di varietà moderne
all'interno di prodotti di nicchia ottenuti da farri e razze locali di frumento duro e tenero.
E' necessario pertanto scegliere sequenze diagnostiche che marcano lo stesso genoma
oppure genomi differenti5. L’integrazione di alcune analisi molecolari dovrebbe rendersi
necessaria
per
diagnosticare
la
presenza
di
una
o
più
specie
bersaglio
e,
contemporaneamente, la presenza/assenza di eventuali varietà moderne contaminanti. In
quest’ultimo caso, con tecniche più moderne, sarà possibile anche quantificare il grado di
contaminazione.
Le varietà moderne di frumento, “comunemente ed ampiamente” coltivate, appartengono
in primis al frumento tenero T. aestivum (2n=6X=42; genoma AABBDD) e secondariamente
al frumento duro T. durum (2n=4X=28; genoma AABB). Queste varietà sono tutte a statura
5
L’insieme dei cromosomi contenuto nel nucleo cellulare è chiamato genoma. Il genoma dei frumenti coltivati, descritto sinteticamente con lettere
maiuscole, appartiene al tipo A nel farro monococco al tipo AB nel frumento duro e farro dicocco ed al tipo ABD nel frumento tenero e nello spelta. Il
tipo di lettera fa riferimento al genoma dei progenitori selvatici ancestrali alle forme coltivate.
8
bassa (minore di 70 cm) o medio-bassa (tra 70 e 90 cm) a causa della presenza di geni
nanizzanti con effetto maggiore sul fenotipo.
Le varietà antiche, anche se moderne dal punto di vista della valorizzazione, sono
principalmente rappresentate dal farro, dalle razze locali e dal frumento Kamut.
Il farro “comunemente e localmente” coltivato è costituito dalle seguenti specie (Fig. 2):
T. monococcum (2n=2X=14; genoma AA)
T. dicoccon (2n=4X=28; genomi AABB)
T. spelta (2n=6X=42; genomi AABBDD).
Tutte le specie di farro presentano, anche dopo la raccolta, la cariosside saldamente
protetta dalle glume (cariosside vestita)
Il Kamut, un altro cereale di nicchia, dovrebbe essere una selezione del T. turgidum,
filogeneticamente molto simile al frumento duro (T. durum; genomi AABB). Questo
frumento similmente ad altre forme coltivate di frumento duro e tenero presenta la
cariosside nuda, cioè non protetta dalle glume e di dimensioni abbastanza elevate.
I farri, il Kamut, e le varietà selezionate da razze locali nelle filiere corte, dal punto di vista
filogenetico sono “antichi” rispetto alle varietà moderne di frumenti teneri e duri. Questo
“carattere” è descritto fenotipicamente non solo dalla presenza di glume saldamente
associate alla cariosside (nel caso dei farri), ma anche dalla statura, che è alta in tutte e
quattro le unità tassonomiche oggetto di indagine. Quest’ultimo carattere condiviso, se
“marcato” da una differenza genetica chiara può rappresentare il primo stadio per una
possibile differenziazione diagnostica.
I metodi di seguito proposti non sono da considerarsi alternativi. In funzione del tipo di
matrice possono essere applicati singolarmente oppure, in casi più complessi (misture di
specie diverse), si prevede una loro integrazione gerarchica.
Discriminazione di varietà a statura bassa da quelle a statura alta
Questo approccio si basa sulla identificazione di mutazioni recenti. Se i frumenti antichi
sono tutti a statura alta (superiore ad 1 m), quelli moderni, per effetto del trasferimento del
gene Rht dal Norin 10 (una varietà giapponese non più in uso) sono tutti a statura bassa.
Rintracciare questa mutazione puntiforme tramite la messa a punto di un metodo PCR
(Polymerase Chain Reaction) serve a poter identificare nei prodotti di post-raccolta la
9
presenza di contaminazioni da varietà moderne tramite una semplice reazione6. Premesso
che questa mutazione marca sia il genoma B (locus Rht-B1b) che il genoma D (locus RhtD1b) per condurre l’analisi sono necessari i materiali che seguono.
Materiali necessari
- Per il cromosoma 4B il primer forward BF (5’ –ggtagggaggcgagaggcgag-3’) combinato con
il primer reverse WR1 (5’-catccccatggccatctcgagctg-3’) produce una reazione positiva
(banda di circa 250 bp) per piante alte e quindi per i farri, il kamut e le varietà da razze
locali. Quando il primer forward BF viene combinato con il reverse MR1 (5’catccccatggccatctcgagcta-3’), il genoma delle piante a bassa statura (varietà moderne di
frumento duro e tenero) sarà rintracciato con una singola reazione (Ellis et al. 2002).
- Per il cromosoma 4D il primer forward DF2 (5’-ggcaagcaaaagcttcgcg-3’) oppure DF (5’cgcgcaattattggccagagatag-3’) in combinazione con WR2 (5’-ggccatctcgagctgcac-3’)
produce una reazione positiva (banda di circa 300 bp) per piante alte e quindi per i farri
il kamut e varietà sviluppate da razze locali. Quando il primer forward DF2 oppure DF
viene combinato con il reverse MR2 (5’-ccccatggccatctcgagctgcta-3’) il genoma delle
piante a bassa statura (varietà moderne di frumento tenero) sarà rintracciato con una
singola reazione (Ellis et al. 2002).
Pre-requisiti per l'analisi
-
Presenza di DNA nel prodotto da essere analizzato.
-
Estraibilità del DNA, dal prodotto da essere analizzato, che dovrà essere di quantità,
purezza e lunghezza dei frammenti sufficiente.
-
Controllo: DNA da varietà note delle tre specie di farro, del kamut e di varietà locali e
commerciali di frumento duro e tenero.
Questo protocollo di rintracciabilità mediante PCR merita di essere messo a punto in
relazione alla matrice di estrazione per quanto riguarda la purificazione del DNA ed in
relazione alla tecnica di amplificazione per quanto riguarda la reazione PCR. Per
quest’ultimo aspetto, essendo il polimorfismo dovuto alla variazione di un singolo
nucleotide, sarà necessario fare in modo che l’annealing nei primi cicli di amplificazione sia
6
Il metodo PCR si basa sull’attività dell’enzima DNA-polimerasi. Questo enzima, in presenza di un piccolo frammento di DNA innesco (primer) che
si associa ad un filamento bersaglio di DNA, in presenza di nucleotidi e magnesio, catalizza la sintesi di un nuovo filamento di DNA secondo un
modello semiconservativo.
10
molto specifico, ricorrendo ad esempio a concentrazioni di cloruro di magnesio ottimali ed
a cicli touchdown. Tramite la Real Time PCR, trattata più avanti nel documento, il test
genetico diventa molto più efficiente
Tipo di discriminazione
Questa reazione dovrà discriminare dai cereali di interesse economico (farri e kamut,
varietà da razze locali) le varietà moderne di frumento duro e tenero (che entrambe
condividono il genoma B tramite le combinazioni di primer che marcano il genoma B.
Una volta distinte (frumenti antichi vs frumenti moderni) queste due grandi categorie sarà
necessario distinguere le tre differenti specie di farro.
Isolamento di marcatori genoma specifici
Marcatori specifici per il genoma A, B e D rispettivamente, sono importanti per effettuare
uno screening di base al fine di differenziare specie che mancano di uno oppure due di
questi genomi.
Con questi marcatori ad esempio è possibile distinguere all’interno dei farri:
a) T. monococcum da T. dicoccon;
b) T. monococcum da T. spelta
c) T. dicoccon da T. spelta.
Gli stessi marcatori possono distinguere:
a) T. monococcum e T. dococcon dal frumento tenero
b) T. monococcum da frumento duro
Ma quest’ultima tappa è stata già superata mediante lo screening della mutazione Rht.
Il criterio di uso di questi marcatori si basa sul principio della presenza/assenza. Un
marcatore genoma specifico è presente in concomitanza al contributo genomico della
specie o varietà indesiderata o contaminante.
All’interno del gruppo di cereali di interesse economico è possibile ad esempio utilizzare
anche sequenze Dgas44 tramite PCR (Bryan et al. 1998). Questo metodo è abbastanza
robusto perché le sequenze Dgas44 sono ripetute e, possono essere identificate anche in
matrici come pasta, sfarinati e prodotti da forno e pasticceria. Questa sequenza permette
anche la identificazione di bassi livelli di contaminazione da parte del genoma D (specifico
di farro spelta e frumento tenero). E’ pertanto evidente come questo marcatore possa
11
essere utile a determinare l’autenticità della pasta italiana che per definizione deve essere
di “grano duro”.
Con questo tipo di sequenze è possibile con tecniche diagnostiche più mirate (Real Time
PCR) anche valutare il grado di contaminazione di un genoma rispetto al contenuto di
genomi desiderati (percentuale di mistura di varietà diverse).
Metodi classici
Si prevede la costituzione di una mistura artificiale di DNA fogliare dall’1% al 20 % di
contaminazione. La validazione del messaggio molecolare avverrà con DNA da
equivalenti misture di sfarinato di coppie di specie (specie commerciale più specie
contaminante). Con questo criterio sarà:
a) efficiente la possibilità di discriminare il farro dicocco dal farro spelta (mistura molto
diffusa a livello commerciale) mediante marcatori a copia ripetuta, specifici del genoma D.
b) anche possibile con una sequenza ripetuta, opportunamente identificata sul genoma A
oppure B, rintracciare contaminazioni di T. monococcum oppure T. dicoccon (misture meno
diffuse a livello commerciale).
Isolamento di un set di marcatori diagnostici genotipo-specifici
Nel caso in cui sarà necessario identificare una particolare varietà all’interno della specie,
allora si dovrà identificare geneticamente questa varietà rispetto ad altre della stessa
specie. Questo metodo vale in modo particolare per il frumento Kamut che, data la sua
similarità genetica con il frumento duro ed altre forme di frumenti tetraploidi, necessita di
essere rintracciato mediante mutazioni genotipo specifiche. Ciò nonostante il pre-screening
mediante la mutazione Rht permette una prima differenziazione da tutte le varietà
moderne di frumento duro. Sarà pertanto necessario identificare lo stesso Kamut da altri
cereali tetraploidi a statura alta che pur attualmente non coltivati in occidente potrebbero
essere utilizzati in modo fraudolente.
Per distinguere con efficienza i cereali che condividono gli stessi genomi, sarà necessario
isolare alleli rari e specifici per la varietà che si vorrà rintracciare (Donini et al. 1998). Gli
alleli rari possono risultare non specifici perché dipendenti da frequenza. In piccole
popolazioni o campioni possono risultare specifici per la varietà di interesse economico
12
però, se aumenta la dimensione della popolazione, lo stesso marcatore può apparire anche
in altre varietà. In questo caso si ricade nella metodologia prevista per l’identificazione
varietale. Pertanto con le distanze genetiche minime sarà possibile identificare
geneticamente la varietà di interesse commerciale (Figliuolo e
Perrino, 2004).
Campione
(tessuto, cibo)
Come accennato, è necessario isolare un set di marcatori
diagnostici (es microsatelliti) che manifestano alleli rari nei
materiali di interesse economico e procedere alla valutazione
genomica delle matrici oggetto di diagnosi solo con l’uso di questi
Estrazione DNA
marcatori. Questo criterio permette l’identificazione genetica
delle varietà, li dove è possibile separarle in matrici “misture di
genotipi” (es semi distinguibili nella stessa confezione). Questo
metodo risulta meno sensibile se l’obiettivo è quello di
identificare possibili miscele e contaminazioni.
Strategie per una analisi
specifica
Un aspetto importante in analisi di identificazione varietale è
rappresentato
dall’accuratezza
del
campionamento.
Il
campionamento dovrà riguardare da un lato il genoma (quantità
e distribuzione genomica dei loci polimorfici) e da un altro lato la
popolazione di varietà che rappresenta il pool genico della varietà
Determinazione
qualitativa e quantitativa
che si dovrà identificare. I due tipi di campionamento sono Figura 4. Diagramma
delle procedure finalizzate
correlati. All'aumentare del campionamento genomico si può all’analisi genomica
ridurre il campionamento della popolazione di germoplasma e viceversa. Generalmente,
risulta che sia più efficiente aumentare la dimensione del campione di marcatori piuttosto
che quella degli individui nello stimare con precisione l'identità di una varietà in termini
di distanze genetiche
Per tutelare i diritti del “proprietario/consumatore” bisogna stimare alcune statistiche che
permetteranno di decidere in caso di contenzioso. In particolare chi dovrà risolvere il
contenzioso vorrà sapere la probabilità che una varietà scelta a caso abbia lo stesso
genotipo di una data varietà commerciale oppure la probabilità relativa che una varietà
possieda un genotipo identico a quello della varietà commerciale. Questo tipo di
applicazione ricade negli studi di identità/non identità di due o più genotipi e, l'approccio
di calcolo seguito è, come accennato, di tipo probabilistico (Evett e Weir 1998).
13
Questo set di marcatori potrà essere utile a rintracciare una varietà (es.il frumento kamut o
il Creso) rispetto ai suoi geneticamente simili. Durante questo processo di identificazione
di mutazioni rare potrebbe realizzarsi anche l’identificazione di un unico marcatore
diagnostico.
L’analisi genomica: aspetti procedurali
Il laboratorio di analisi genomica, dopo aver acquisito il campione, attribuisce un codice di
identificazione e procede al processamento finalizzato all’analisi genetica. Il primo
passaggio consiste nell’estrazione del DNA (Fig. 4). Esistono diversi kit commerciali le cui
soluzioni sono già predisposte per l’uso. Il tipo di kit utilizzato dipende dal tipo di
campione che dovrà essere analizzato. Il principio di base dell’estrazione degli acidi
nucleici consiste nel purificare il DNA contenuto in una determinata matrice (prodotto
trasformato, cariosside, tessuti fogliari). Spesso è più conveniente preparare le soluzioni e i
solventi di base direttamente in laboratorio. A tal fine esistono numerosi protocolli pratici
di estrazione e purificazione del DNA (Maniatis, 1989). Negli ultimi anni lo sviluppo delle
tecnologie chimiche e robotiche hanno contribuito alla diffusione di estrattori
automatizzati in grado di processare numerosi campioni (workstation) tramite l’uso di kit
commerciali standardizzati per estrazioni manuali o automatizzate.
I marcatori molecolari, come già accennato, si basano sul polimorfismo della sequenza del
DNA e prevedono l’utilizzo di specifiche tecniche per evidenziare, visualizzare e
quantificate le mutazioni del DNA. Le principali tecniche usate per evidenziare i
polimorfismi del DNA sono offerete dai seguenti marcatori:
RAPDs (Random Amplified Polymorphic DNA), SCAR (Sequence Characterised Amplified
Region) AFLPs (Amplified Fragment Length Polymorphism), SNPs (Single Nucleotide
Polymorphism) e microsatelliti SSRs (Simple Sequence Repeats). Tutti questi marcatori
prevedono l’utilizzo del metodo PCR e real-time PCR.
Metodi di estrazione del DNA
Pre-trattamento dei campioni
bead mill, es TissueLyser (QIAGEN)
Purificazione manuale del DNA
14
DNeasy Tissue Kit (QIAGEN)
Colonnine con membrane al silicio
Purificazione automatizzata del DNA
Con BioRobot M48 (QIAGEN)
Particelle magnetiche
Protocollo “MagAttract”
BioSprint 15
Magnetic particles
Protocol “Tissue”
Con 6100 Nucleic Acid PrepStation (Applied Biosystems)
Applica il protocollo “TransPrep chemistry”
L’omogenizzazione del campione avviene utilizzando un mulino (bead mill) che prevede
l’utilizzo di provette che contengono il campione, una sfera metallica e tampone di
estrazione. Il bead mill esercita una agitazione ad elevata frequenza che determina la
frantumazione in provetta del campione.
Il DNeasy Tissue Kit utilizza una tecnologia centrata sull’utilizzo di membrana di gel di
silice per un isolamento del DNA cellulare totale rapido ed efficiente, senza che si usi
fenolo e cloroformio (come solventi organici) per la purificazione ed etanolo per la
precipitazione. I campioni possono essere meccanicamente frantumati in una prima fase e,
successivamente, lisati enzimaticamente tramite la Proteinasi K. Il sistema tampone è
ottimizzato per permettere il legame selettivo del DNA alla membrana DNaeasy. I lisati
sono “caricati” sulle colonnine Dneasy-spin e, in seguito ad un breve spin, il DNA
selettivamente si lega alla membrana di silica-gel mentre le sostanze contaminanti
attraversano la membrana e vengono eluite. I contaminanti residui e gli inibitori
enzimatici saranno rimossi in due lavaggi consecutivi. Il DNA purificato, per un pronto
utilizzo, è successivamente eluito in acqua oppure in un tampone a basso contenuto salino
(Tris-EDTA 1X).
La workstation BioRobot M48 rilascia il DNA purificato mediante un sistema
automatizzato capace di processare un ampio numero di campioni (anche 48 campioni
possono essere processati in un ciclo). La testa della pipetta contiene 6 siringhe ad alta
precisione che operano simultaneamente per permettere l’aspirazione o dispensare piccoli
volumi di liquido (25-1000 µl) mediante puntali muniti di filtro. La tecnologia
15
“MagAttract” combina la velocità e l’efficienza della purificazione basata sulle membrane
al silica-gel con l’utilizzo di particelle magnetiche.
La workstation BioSprint 15 è un sistema aperto che impegna poco spazio e si gestisce
agevolmente tramite una piccola tastiera. E’ utilizzata per la purificazione di DNA da
differenti tipologie di campioni dopo che questi sono stati lisati. La workstation processa
da 1 a 15 campioni per corsa. Essa controlla un array di ganci magnetici che attraggono o
respingono particelle magnetiche trasferendole da una provetta ad un’altra. Il
trasferimento sequenziale delle particelle magnetiche permette una rapida purificazione,
di molecole bersaglio di DNA genomico, fino al lavaggio delle particelle ed alla eluizione
delle molecole bersaglio pure.
La PrepStation 6100 Nucleic Acid processa contemporaneamente fino a 96 campioni,
utilizzando diverse chimiche in funzione del tipo di materiale di partenza da cui estrarre
acidi nucleici. La workstation utilizza un sistema pneumatico di aspirazione dei fluidi
all’interno di una micropiastra provvista di tubi con filtro.
Strategie per una determinazione specifica
Alla base dello sviluppo di gran parte dei marcatori molecolari oggi più utilizzati c’è la
reazione a catena della polimerasi (PCR=Polymerase Chain Reaction). I primi marcatori
molecolari erano rappresentati dagli RFLP (Restriction Fragment Length Polymorphisms)
basati sul polimorfismo dei siti di restrizione del DNA operato da specifici enzimi7. Questa
tecnica è in parte tramontata perché prevedeva l’uso di sostanze marcate con radionuclidi
ed anche perché è stata integrata da marcatori più recenti come gli AFLP basati sulla
reazione a catena della polimerasi.
PCR
La PCR (Polymerase Chain Reaction) è utilizzata per amplificare porzioni brevi di sequenza
di DNA. La tecnica permette ad un piccolo ammontare di molecole di DNA iniziali (DNA
bersaglio) di essere amplificato molte volte in modo esponenziale, in presenza di due
primers (determinano l’inizio e la fine della regione che necessita di essere amplificata) di
Taq polimerasi (enzima che catalizza il processo di amplificazione) dei singoli nucleotidi
7
Gli enzimi di restrizione effettuano un taglio della doppia elica dopo aver riconosciuto una specifica sequenza. In funzione del tipo di enzima, il
taglio può essere “netto” o “sfalsato” mentre, la sequenza riconosciuta può essere lunga quattro o sei paia di basi.
16
(base per la sintesi del DNA) di un tampone che ricostituisce l’ambiente chimico
appropriato per la Taq polimerasi. Il prodotto PCR può essere identificato come quantità e
dimensione tramite gel elettroforesi oppure tramite elettroforesi capillare.
Ciclo 1
Ciclo 2
Ciclo 3
Ciclo 4
Figura 5. Incremento esponenziale del frammento di DNA originario in seguito alla reazione a
catena della polimerasi
Amplificazione esponenziale
L’amplificazione esponenziale è dovuta alla replicazione semiconservativa del DNA (Fig.
5). Ciò nonostante dopo 30-35 cicli di attività polimerasica l’enzima Taq tende ad esaurire
il potere catalitico. Di seguito si riporta il calcolo del numero di copie di doppia elica
rappresentato in Fig. 5.
21= 2 copie
22= 4 copie
23= 8 copie
24= 16 copie
235= 34 miliardi copie
Formula: 2n (n = numero di cicli)
17
RAPD
I marcatori RAPD sono prodotti di amplificazione di sequenze di DNA ignote utilizzando
come innesco oligonucleotidi (primer) brevi ed a sequenza arbitraria, che, pertanto, non
richiedono alcuna conoscenza della sequenza del DNA. Il basso costo, l’efficienza nello
sviluppare un elevata quantità di marcatori del DNA in poco tempo e la richiesta di
attrezzature poco sofisticate hanno reso i
RAPD una tecnica molto comune sebbene la
riproducibilità dei profili è ancora al centro
del dibattito scientifico. Generalmente per
questa tecnica si utilizzano come primer un Figura 6. Profilo di marcatori RAPD
singolo breve (10 basi) oligo sintetizzato, a sequenza casuale, per amplificare piccole
quantità di DNA genomico totale (ordine di ng) con basse temperature di annealing
durante le reazioni PCR. La variazione nella sequenza tra DNA bersaglio da differenti
varietà darà origine ad un polimorfismo di presenza e assenza di bande, a causa di
cambiamenti dei siti di innesco del primer (Fig. 6). I RAPD sono uno strumento molto utile
per determinare le relazioni genetiche entro e tra specie. I RAPD polimorfici possono
essere trasformati in SCAR.
18
AFLP
Questi marcatori
integrano
la
tecnologia degli
1. Digestione del DNA con una coppia di enzimi
Sito di restrizione per
EcoRI
MsEI
RFLP a quella
RAPD. Essi sono
in
grado
di
determinare
i
polimorfismi dei
2. Ligation degli adattatori Eco/Mse
siti di restrizione
senza conoscere
la
sequenza
3. Pre-amplificazione selettiva con un nucleotide selettivo
mediante la PCR
e
quindi
l’amplificazione
di frammenti di
restrizione.
In
questo caso lo
stampo per la
PCR è il
4. Amplificazione selettiva (con tre nucleotidi selettivi)
5. Elettroforesi di frammenti marcati in gel di acrilammide o
tramite elettroforesi capillare in un sequenziatore automatico
DNA genomico
digerito
enzimi
con
di
restrizione.
I
primer
contengono
sito
il
6. Analisi dell’impronta digitale genetica e identificazione della
varietà di interesse
di
riconoscimento
Figura 7 Schema per la generazione di marcatori AFLP
dell’enzima di restrizione così come nucleotidi arbitrari addizionati che si estendono oltre
il sito di restrizione. La porzione fissata conferisce stabilità al primer mentre la porzione
casuale permette di individuare solo alcuni loci. I prodotti amplificati possono essere
19
risolti in un gel di poliacrilammide o tramite elettroforesi capillare. I frammenti
polimorfici possono essere convertiti in marcatori STS
SNP
I SNP (Single Nucleotide Polymorphisms) sono variazioni nella sequenza del DNA che si
osservano quando un singolo nucleotide (A, T, C, o G) differisce tra i membri di una
specie. La determinazione avviene mediante sequenziamento diretto o indiretto del DNA
(Fig.7).
Figura 7. Sequenza di un frammento di DNA così come evidenziata in seguito ad elettroforesi
capillare
20
Microsatelliti (SSR)
Un microsatellite è un breve blocco di sequenza di DNA che consiste di una unità di sequenza
ripetuta in tandem spesso lunga meno di 150 coppie di basi. In un microsatellite la sequenza ripetuta
consiste di due, tre o quattro nucleotidi (di-, tri-, e tetranucleotide repeats rispettivamente), e può
essere ripetuta da 10 a 100 volte (Fig. 8). Il numero di unità ripetute (repeats) ad un particolare
locus è ipervariabile (altamente polimorfico) tra individui della stessa specie. Pertanto le sequenze
microsatelliti possono essere usate per il fingerprinting genetico ed i test di paternità.
Figura 8. Rappresentazione di sequenza microsatellite in un eterozigote (alto) e rappresentazione
della tecnica
21
Determinazioni quantitative
La tecnologia offerta dalla Real
1. Denaturazione (95 °C)
2. Annealing di primer e sonda (60 °C)
time PCR permette non solo una
determinazione
(presenza
o
assoluta
assenza)
di
un
genoma o contaminante in una
data matrice, ma anche una
3. Polimerizzazion e rimpiazzo della sonda (60 °C)
quantificazione percentuale del
genoma contaminante rispetto a
quello totale. La RT-PCR si basa
sull’utilizzo di una polimerasi
speciale denominata TaqMan e
sulla presenza addizionale in una
reazione
di
amplificazione
4. Attività esonucleasica (60 °C)
classica di una sonda marcata con
fluorocromi
specifica
alla
sequenza di DNA amplificata.
Quest’ultima emette fluorescenza
ogni volta che la nuova sequenza
viene
polimerizzata
(vedere
figura). In tempo reale è possibile
osservare
fluorescenza,
l’aumento
di
correlato
alla
quantità di DNA contaminante,
in
relazione
ai
cicli
di
amplificazione.
Figura 9. Real-time-PCR / TaqManTM-technology
Quantificazione relativa di una
22
varietà contaminante una matrice alimentare
Questa procedura è basata su almeno due curve standard:
o
o
La prima per la varietà nota
La seconda per la varietà ricercata o contaminante
In questo tipo di analisi si utilizzano, inoltre, appropriati controlli genetici.
L’ammontare relativo (%) è calcolato dividendo l’ammontare della varietà contaminante
con il totale e moltiplicando tutto per 100.
Figura 10. Metodo di analisi e calcolo previsto dalla RT-PCR
Nella RT-PCR l'amplificazione esponenziale del DNA bersaglio specifico è misurato
tramite le sonde marcate con fluorocromi (tecnologia TaqMan). Se la specie contaminante
o la varietà-specifica è presente nel materiale oggetto di investigazione, viene trasmesso un
segnale fluorescente. Questa tecnica è particolarmente vantagiosa per analisi di routine. E'
importante rimarcare che l'amplificazione e la determinazione analitica con questa tecnica
23
avviene in provette chiuse senza alcuna manipolazione post-PCR, riducendo in tal modo
potenziali contaminazioni incrociate. In secondo luogo, il test non è lungagginoso poichè
la successiva verifica elettroforetica dei prodotti PCR non è necessaria. Terzo, una grande
quantità di campioni alimentari si possono analizzare in una singola reazione. Questa
tecnica permette la messa a punto di saggi quantitativi e pertanto riserva grande
flessibilità applicativa.
Amplificazione dell’intero genoma
REPLI-g
L’analisi della sequenza di DNA dei campioni di prodotti alimentari può essere limitata da
piccole quantità di DNA disponibile. Il Repli-g (Quiagen) kit permette l’uniforme
amplificazione del genoma totale da campioni di piccole dimensioni.
Figura 11 Schema di azione della polimerasi Phi29
Il REPLI-g è un metodo di “Multiple displacement amplification” (MDA) che realizza
l’amplificazione isotermica del genoma utilizzando solo una DNA polimerasi processiva
capace di replicare 100 Kb senza mai dissociarsi dal DNA genomico stampo. Questa DNA
polimerasi presenta una attività esonucleasica 3’-5’ di lettura della bozza che consente di
mantenere una elevata fedeltà di replicazione ed è utilizzata in presenza di primers
resistenti all’attività esonucleasica per ottenere rese elevate di DNA. Phi29 è una DNA
polimerasi isolata dal batteriofago phi29 che può essere utilizzata per una amplificazione
dall’elevato livello di fedeltà del DNA genomico bersaglio. Proprietà importanti della
polimerasi phi29 sono rappresentate dalla forte capacità di dislocazione dei filamenti di
DNA, dall' attività di proof reading (correzione delle bozze) e da una elevata processività. A
causa dell’attività di dislocazione dei filamenti, dopo una iniziale fase di denaturazione, la
24
polimerasi phi29 non richiede ulteriore ciclizzazione per innescare la sintesi del filamento
nascente.
Microchip
Per la determinazione specifica di sequenze multiple di DNA in una singola analisi, il
microchip è ormai un metodo consigliato. Esso prevede che siano disponibili per la(le)
specie oggetto di indagine vetrini (microchip) con migliaia di sonde8 fissate specifiche (per
una data specie/varietà). Di conseguenza centinaia o anche migliaia di specie/varietà
possono essere determinate su un microchip di dimensioni pari ad 1 centimetro quadrato.
La determinazione è funzione di una specifica fluorescenza associata al DNA (estratto o
amplificato) proveniente da campioni con genoma ignoto, che si associa alle sonde (a
funzione o sequenza nota) del microchip.
Figura 12. Posizione su vetrino di microchip con presenza di sonde associate e non associate ai
frammenti di acidi nucleici (possono essere anche RNA)
8
In biologia molecolare per sonda si intende una molecola in grado di rilevare secondo una relazione di specificità una
molecola omologa.
25
La tecnologia LiquiChip
Esistono diverse varianti al microchip. Per gli scopi di questa descrizione come esempio si
riporta l’integrazione di un sistema genere-specifico su un Liquidchip (Fig. 13). In questo
caso la determinazione del segnale ancora richiede un passaggio iniziale di amplificazione.
Al fine di evitare il passaggio di PCR selettiva prevista dal Whole Genome Amplification
(WGA) questo approccio, pur non essendo molto utilizzato si rivela promettente.
Figura 13. Rappresentazione schematica della liquidchip technology
La rilevazione dell’analita si basa sulla simultanea determinazione e classificazione del
codice di fluorescenza e sulla fluorescenza reporter. Lo strumento registra solo la
fluorescenza reporter che è associata con un valido segnale di classificazione.
Il LiquiChip assay è basato sulla tecnologia xMAP e coinvolge l’interazione di molecole
“cattura” immobilizzate su sfere con la reazione con un partner (analita) in soluzione. Una
molecola reporter, specifica per l’analita è utilizzata per quantificare l’interazione. Sono
disponibili differenti set di sfere, ciascuno contenente una definita mistura di due coloranti
fluorescenti. Accoppiando differenti molecole di cattura a differenti set di sfere si possono
effettuare saggi in multiplex.
Differenti set di sfere con differenti codici di colore e
molecole di cattura sono aggiunte ad un sistema dove esse reagiscono con differenti
analiti. Il codice di colore unico per ciascun set di sfere permette la discreta quantificazione
di ciascun analita nel lettore del LiquiChip. Questo tipo di analisi permette di ottenere una
buona informazione anche utilizzando piccoli quantitativi di campione. Nel lettore del
liquid chip ciascuna reazione (set di sfere o bead set) è identificata dalla propria firma
spettrale in seguito all’irraggiamento di un laser di classificazione rosso. Il segnale reporter
26
da ciascuna reazione è simultaneamente quantificato dalla fluorescenza generata dal laser
reporter verde.
Reazione Padlock
Un’altra strategia d’uso dei microchip è la reazione Padlock che combina la ligation
(ligazione) di una molecola circolare con una specifica strategia di identificazione.
Figura 14. Schema di azione di una reazione padlock
Questo sistema, dal punto di vista molecolare, molto più complicato dei precedenti,
prevede l’uso di sonde “invertite”, di primer senso ed antisenso, e di amplificazioni PCR.
Nello schema di Fig. 14 l’amplificato padlock invertito è ibridizzato ad un microchip che
contiene i cosiddetti cZIP (ZIPcode oligonucleotides) complementari.
27
Bibliografia
Bryan G. J., Dixon A., Gale M. D. e G. Wiseman (1998) A PCR-based Method for the
Detection of Hexaploid Bread Wheat Adulteration of Durum Wheat and
Pasta. J. Cereal Sci. 28:135-145.
Donini P, Stephenson P, Bryan GJ e Koebner RMD (1998). The potential microsatellites
for high throughput genetic diversity assessment in wheat and barley.
Genetic Resources and Crop Evolution 45: 415-421.
Ellis M. Spielmeyer W. Gale K. Rebetzke G. e R. Richards (2002)."Perfect" markers for the
Rht-B1b and Rht-D1b dwarfing genes in wheat. Tehor. Appl. Genet. 105:
1038-1042.
Evett IW e BS. Weir (1998). Interpreting DNA evidence. Sinauer, Sunderland, Mass.
Figliuolo G. e P. Perrino (2004). Genetic diversity and intra-specific phylogeny of
Triticum turgidum L. subsp. dicoccon (Schrank) Thell. Revealed by RFLPs
and SSRs. Genetic Resources and Crop Evolution 51(5): 519-527.
Lafiandra D. e DD Kasarda (1985). One and two dimensional (two pH) polyacrilamide
gel electrophoresis in a single gel: separation of heat proteins. Cereal Chem.
62:314–319.
Maniatis E F (1989). Molecular Cloning: A Laboratory Manual. 2nd ed., Cold Spring
Harbor Laboratory, Cold Spring Harbor, NY.
Peruffo A. D. B., Pogna N. E., Pallavicini C. e E. Pegoraro (1985). Diagrammi
elettroforetici delle gliadine e chiave di identificazione delle varietà di
grano tenero iscritte nel registro delle varietà. Agricoltura e Ricerca 11:7581.
Prasad M, Varshney RK, Roy JK, Balyan HS e PK. Gupta (2000). The use of microsatellites
for detecting DNA polymorphism genotype identification and genetic
diversity in wheat. Theor Appl Genet. 100: 584-592.
Röder M, Plaschke J, Koenig SU, Borner A, Sorrells ME, Tanksley SD e MW.Ganal (1995).
Abundance, variability and chromosomal location of microsatellites in
wheat. Mol. Gen. Genet. 246: 327-450.
Röder MS, Wendehake K, Korzun V, Bredemeijer G, Laborie D, Bertrand L, Isaac P,
Rendell S, Jackson J, Cooke RJ, Vosman B e MW. Ganal (2002). Construction
and analysis of a microsatellite-based database of European wheat varieties.
Theor Appl Genet. 106: 67-73.
28
ALLEGATO 1
ANALISI DELLE PROTEINE DI RISERVA DEL FRUMENTO DURO
Gliadine (Metodo Acid-PAGE)
1) Preparare un elenco dei campioni (registro delle gliadine) di cui si vuol estrarre le proteine prevedendo
delle colonne in cui riportare il peso di ciascuna cariosside (mg) e la quantità di soluzione di estrazione
(µl) che dovrà essere utilizzata;
2) Preparare una serie di provette (eppendorf) da 1,5ml con il numero di ciascun campione;
3) Selezionare una cariosside di dimensioni medie per ciascun campione ponendola nella provetta
corrispondente;
4) Pesare la cariosside su bilancia di precisione riportando il peso (mg) sul registro;
5) Macinare in un mortaio la cariosside, facendo pressione con un pestello;
6) Versare lo sfarinato nella relativa provetta servendosi di una spatolina;
Nota: Prima di porre un’altra cariosside nel mortaio, pulire quest’ultimo ed il pestello con della carta, in
modo che non restino tracce della farina precedente.
7) Dare un’agitata su votex e poi uno spin;
8) Aggiungere alla farina ottenuta una soluzione di N,N dimetil-formammide (soluzione di estrazione)
dosata secondo un rapporto 1:5 peso/vol (cioè per ogni mg di peso della cariosside, si utilizzano 5 µl
della soluzione di estrazione) che si lascia agire per un notte;
Precauzione: operare sotto cappa.
9) Centrifugare quindi la miscela per 10-15’ a circa 10.000 giri a 4°C e recuperare il surnatante (cambiando il
puntale per ogni campione) e ponendolo, con una micropipetta, in una nuova provetta da 1,5 ml,
precedentemente numerata, mentre, la provetta con il pellet può essere conservata in busta ben chiusa in
congelatore per l’eventuale successiva estrazione delle glutenine.
10) Si procede in questo modo per ogni campione.
Separazione delle gliadine
Avviene in gel continuo di poliacrilammide al 6%, con elettroforesi verticale (camere elettroforetiche SE 600 Pharmacia Biotech), in lattato di alluminio 1X (pH 3,1). Si preparano gel a sandwich con vetri di dimensioni
16cm x 18cm x 0,3cm separati da appositi spaziatori (spessorini) in PVC dello spessore di 0,15cm e con
pettini in PVC dello spessore di 0,15cm.
Preparazione dei vetri e delle relative basi
1) Lavare i vetri con etanolo per togliere le impurità e far aderire meglio gli spessorini;
2) Prevedere due vetri per ciascun gel che si vuol preparare;
3) Allineare i vetri poggiandoli sul piano di lavoro e farli combaciare perfettamente nella loro superficie e
lungo ciascun lato;
4) Inserire tra i vetri due spessorini (uno lungo ciascun lato verticale) e risistemare i vetri in modo che
combacino perfettamente;
5) Inserire due sistemi di fissaggio laterali dotati di viti lungo i due lati verticali del sandwich ottenuto,
stringendo completamente le viti solo dopo essersi accertati della perfetta sistemazione dei vetri e degli
spessorini;
6) Inserire la parte inferiore del sandwich nella base orizzontale (dopo aver inserito in quest’ultima una
guarnizione di silicone) e incastrarla perfettamente;
7) Inserire infine, ai due lati e negli appositi alloggi, due fissatori (pomellini neri) e ruotarli fino ad
assumere una posizione verticale in modo da stringere perfettamente il sandwich e non consentire la
fuoriuscita del gel.
Nota: ricordare che ciascuna base orizzontale ha due alloggi per i gel e che anche le camere verticali
prevedono la corsa di due gel contemporaneamente, per cui occorre preparare due gel per base e/o camera,
oppure, se si vuol preparare un solo gel, utilizzare l’apposito spessore in PVC trasparente che simula il
secondo gel.
8) Prevedere un pettine dello spessore di 0,15cm per ciascun gel.
29
Preparazione dei gel e condizioni di corsa
1) Preparare sotto cappa tante beutine da 100ml quanti sono i gel;
2) Versare in ciascuna beutina la quantità prevista dello stock di acrilammide-bis acrilammide
precedentemente preparato (vedi tabella soluzioni);
3) Immediatamente prima di versare il contenuto della beuta tra i vetri, aggiungere i due polimerizzanti
(solfato ferroso e perossido d’idrogeno) precedentemente preparati (vedi tabella soluzioni) ed agitare
rapidamente;
4) Versare altrettanto rapidamente la soluzione tra i vetri montati fino ad arrivare a circa 1 cm dal bordo
superiore dei vetri;
5) Inserire quindi il pettine diagonalmente e lentamente per non creare bolle, immergendolo nel gel per
circa 1,5cm;
6) Attendere quindi circa 1ora per la polimerizzazione, senza muovere o spostare il gel;
7) A polimerizzazione avvenuta, sfilare delicatamente il pettine;
8) Procedere con la pulizia dei pozzetti riempiendoli con tampone di corsa con una pipetta pasteur e
aspirando lo stesso tampone con una siringa con ago spuntato, facendo attenzione a non bucare i
pozzetti;
9) Ripetere l’operazione di pulizia fino a quando non ci sono più residui di acrilammide non polimerizzati
e i pozzetti appaiono ad angolo retto;
10) Sfilare i sandwich dalla base togliendo i due fissatori (neri) ed immergerli nella camera verticale.
Nota: assicurarsi che nella camera ci sia un adeguato volume di tampone di corsa (lattato di alluminio 1X)
pari a circa i 2/3 del volume della camera;
11) Occorre effettuare prima una pre-corsa.
12) Riempire i pozzetti con tampone di corsa, sistemare la vaschetta superiore dopo aver inserito nella sua
parte inferiore una guarnizione di silicone e fissarla con gli stessi fissatori (neri) utilizzati per la base
inferiore;
13) Riempire la vaschetta superiore con la soluzione di corsa fino alla completa immersione dell’elettrodo
metallico, chiudere la camera con il coperchio superiore, collegare gli spinotti (il filo rosso al polo
positivo (+) e il filo nero/azzurro al polo negativo (–)) ed accendere il refrigeratore (settato a 10°C) (LKB)
e l’alimentatore (LKB);
Precauzioni: accendere l’alimentatore solo dopo essersi assicurati di aver connesso tutti gli elementi con la
giusta polarità. In caso di riversamento della soluzione di corsa sul banco, prima spegnere l’alimentatore e
poi procedere alla rimozione del liquido.
14) La pre-corsa procede per circa 1h a 50mA per camera;
15) Procedere quindi al caricamento dei gel (5µl per campione), usando, come riferimento del fronte di
corsa, violetto di metile;
16) Caricare i gel “a serpente” (da sinistra a destra il I davanti, da destra a sinistra il II dietro), caricando il
violetto di metile nel I pozzetto (5-10µl);
17) La corsa procede per circa 2,5-3 h a 100 mA, se vengono caricate due camere elettroforetiche (4 gel).
Nota: per la corsa delle gliadine occorre invertire la polarità degli spinotti, inserendo lo spinotto (+) in
corrispondenza del polo (–) dell’alimentatore e viceversa;
18) A fine corsa, spegnere l’alimentatore e il refrigeratore, sfilare la vaschetta superiore versando il tampone
in essa contenuto e sfilare i gel dalla camera;
19) Per recuperare il gel dai vetri svitare le viti e sfilare i fissatori laterali, con l’aiuto di una spatolina
spingere all’esterno uno spessorino e, facendo leva con l’altro spessorino, aprire i vetri in modo che il gel
resti su uno dei due vetri;
20) Poggiare il lato inferiore del vetro con il gel nella vaschetta contenente il colorante e, sollevando un
angolo del gel con la spatolina, far scivolare delicatamente il gel nel colorante;
21) La colorazione si realizza in vaschette con una soluzione di acido tricloroacetico (TCA) al 12% e Blue di
Coomassie all’1% (vedi tabella soluzioni) che si lascia agire per una notte in lenta agitazione su un
agitatore rotante;
22) Il lavaggio si esegue prima con acqua, e poi con TCA al 12%, ripetendolo più volte a distanza di ore
sempre in lenta agitazione. Nota: dopo il lavaggio, prendere il gel con guanti (piegandolo “a panino”);
Precauzioni: preparare il TCA sotto cappa con cautela ed usando guanti di protezione poiché è altamente
corrosivo. Preparare il Blue di Coomassie con cautela per non disperdere le polveri nell’ambiente.
23) Porre il gel su un transilluminatore a luce bianca e pulirlo spennellandolo;
30
24) Fotografare il gel con macchina Polaroid (GelCam) e pellicole 665-667 Polaroid su luce bianca, utilizzando
il filtro di colore giallo specifico per il Blue di Coomassie;
25) Bagnare con acqua una pellicola trasparente da dialisi, poggiarla e stenderla bene (in modo da eliminare
tutta l’aria) su un vetro;
26) Sistemare il gel sul vetro e ricoprirlo con la pellicola eliminando l’aria;
27) Porre intorno al gel 4 bacchette di PVC trasparente in modo che aderiscano bene ai bordi del gel e
fissarle con clips;
28) Lasciare asciugare ed essiccare il gel 4°C;
29) Togliere infine le bacchette e conservare i gel dopo aver eliminato la pellicola in eccesso.
31
SOLUZIONI PER METODO ACID-PAGE (GLIADINE)
SOLUZIONI
PREPARAZIONE
COMMENTI/PRECAUZIONI
N,N dimetil-formammide (soluzione di estrazione)
Sciogliere 1,1g di N,N dimetil-formammide L’ N,N dimetil-formammide è tossico ed irritante per
in 10 ml di acqua distillata
inalazione e per contatto.
Operare sotto cappa indossando guanti e mascherina di
protezione.
Stock Bis-acrilammide/Acrilammide
Per 1 litro di soluzione: sciogliere 3g
di
Bis-acrilammide,
60g
di
Acrilammide e 240mg di Acido
Ascorbico. Portare a volume con
lattato di alluminio 1X. Filtrare con
carta da filtro (Whatman N.1) in una
bottiglia da 1 litro scura o coperta
con carta di alluminio, porre nel
liofilizzatore e degassare per 5-10
minuti.
L'acrilammide è neurotossica. Allo stato solido può essere
inalata, mentre allo stato liquido può essere assorbita per
via cutanea. Pesare sempre sotto cappa indossando
camice, guanti e mascherina.
Per assicurare la riproducibilità dei dati, occorre
ripreparare la soluzione dopo 1-2 mesi.
Lattato di alluminio 50X a pH 3
Per 500ml di soluzione: sciogliere 31,25g di
Lattato di Alluminio e 50ml Acido Lattico.
Portare a volume con acqua distillata.
Aggiustare il pH.
In un pallone da 5 litri sciogliere 100ml di
lattato di alluminio 50X.
Sciogliere 1g Blue di Coomassie in 100 ml di
etanolo al 100%.
Il lattato di alluminio è altamente polverulento. Pesare
sotto cappa usando una mascherina anti-polvere.
Conservare la soluzione al buio o coprire la bottiglia con
carta di alluminio. Conservare a 4°C.
Conservare la soluzione al buio o coprire il pallone con
carta di alluminio. Conservare a 4°C.
Preparare il Blue di Coomassie con cautela per non
disperdere le polveri nell’ambiente.
Sciogliere 40mg in 1ml di soluzione
tampone (lattato di alluminio 1X)
E’ disponibile tal quale.
Sciogliere 0,5g di violetto di metile in 25ml
di acqua distillata (1:50)
Va preparato al momento e conservato a 4°C e al buio.
Lattato di alluminio 1X
Blue di Coomassie (1%)
Solfato ferroso (polimerizzante del gel)
Perossido d’idrogeno (acqua ossigenta) (3%)
Violetto di metile
Acido Tricloroacetico (TCA) al 100%
Va conservata a 4°C e al buio.
Preparare il Violetto di metile con cautela per non
disperdere le polveri nell’ambiente.
Ad una bottiglia contenente 500g di TCA, Preparare il TCA sotto cappa con cautela ed usando
aggiungere 227ml di acqua distillata. guanti di protezione poiché è altamente corrosivo.
Agitare più volte per favorire lo
32
Acido Tricloroacetico (TCA) al 12%
Gel di poliacrilammide
Colorante per i gel
scioglimento. La soluzione risultante
conterrà TCA al 100% peso/vol.
Aliquotare 120ml di TCA al 100% e portare Preparare il TCA sotto cappa con cautela ed usando
il volume ad 1 litro con acqua distillata. guanti di protezione poiché è altamente corrosivo.
Agitare.
In una beutina per ogni gel versare: 45-50ml Usare sempre guanti di protezione
di stock di Acrilammide, 10µl di Solfato
ferroso (FeSO4), 50µl Perossido d’idrogeno
(H2O2) al 3%.
Per ciascun gel aliquotare 180ml di TCA al
12% e 10ml di Blue di Coomassie. Agitare.
33
Glutenine (Metodo SDS-PAGE)
L’estrazione delle glutenine può essere realizzata a partire dal pellet da cui sono state in precedenza estratte le gliadine, oppure selezionando e macinando nuove
cariossidi (quest’ultima soluzione è preferibile).
1) Preparare un elenco dei campioni (registro delle glutenine) di cui si vuol estrarre le proteine prevedendo delle colonne in cui riportare il peso di ciascuna
cariosside (mg) e la quantità di soluzione di estrazione (µl) che dovrà essere utilizzata;
2) Preparare una serie di provette (eppendorf) da 1,5ml con il numero di ciascun campione;
3) Selezionare una cariosside di dimensioni medie per ciascun campione ponendola nella provetta corrispondente;
4) Pesare la cariosside su bilancia di precisione riportando il peso (mg) sul registro;
5) Macinare in un mortaio la cariosside, facendo pressione con un pestello;
6) Versare lo sfarinato nella relativa provetta servendosi di una spatolina;
Nota: prima di porre un’altra cariosside nel mortaio, pulire quest’ultimo ed il pestello con della carta, in modo che non restino tracce della farina precedente.
7) Dare un’agitata su votex e poi uno spin;
8) Aggiungere alla farina una quantità di liquido estrattore nel rapporto di 1:10 (peso/vol), ossia, per ogni mg di peso della cariosside, utilizzare 10 µl di
soluzione di estrazione che si lascia agire per una notte;
11) Centrifugare a10.000 giri a 4°C per 10-15’e recuperare il surnatante (cambiando il puntale per ogni campione) e ponendolo, con una micropipetta, in una
nuova provetta da 1,5 ml, precedentemente numerata;
12) Si procede in questo modo per ogni campione.
Separazione delle glutenine
Avviene con elettroforesi verticale (camere elettroforetiche SE 600 - Pharmacia Biotech) in 2 gel sovrapposti di poliacrilammide costituiti da un “bottom gel” (o
“running gel”) (inferiore) e da uno “stacking gel” (superiore). Si preparano gel a sandwich con vetri di dimensioni 16cm x 18cm x 0,3cm separati da appositi
spaziatori (spessorini) in PVC dello spessore di 0,15cm e con pettini in PVC dello spessore di 0,15cm.
Preparazione dei vetri e delle relative basi
1) Lavare i vetri con etanolo per togliere le impurità e far aderire meglio gli spessorini;
2) Prevedere due vetri per ciascun gel che si vuol preparare;
3) Allineare i vetri poggiandoli sul piano di lavoro e farli combaciare perfettamente nella loro superficie e lungo ciascun lato;
4) Inserire tra i vetri due spessorini (uno lungo ciascun lato verticale) e risistemare i vetri in modo che combacino perfettamente;
5) Inserire due sistemi di fissaggio laterali dotati di viti lungo i due lati verticali del sandwich ottenuto, stringendo completamente le viti solo dopo essersi
accertati della perfetta sistemazione dei vetri e degli spessorini;
6) Inserire la parte inferiore del sandwich nella base orizzontale (dopo aver inserito in quest’ultima una guarnizione di silicone) e incastrarla perfettamente;
7) Inserire infine, ai due lati e negli appositi alloggi, due fissatori (pomellini neri) e ruotarli fino ad assumere una posizione verticale in modo da stringere
perfettamente il sandwich e non consentire la fuoriuscita del gel.
Nota: ricordare che ciascuna base orizzontale ha due alloggi per i gel e che anche le camere verticali prevedono la corsa di due gel contemporaneamente, per cui
occorre preparare due gel per base e/o camera, oppure, se si vuol preparare un solo gel, utilizzare l’apposito spessore in PVC trasparente che simula il secondo
gel.
8) Prevedere un pettine dello spessore di 0,15cm per ciascun gel.
34
Preparazione dei gel e condizioni di corsa
1) Si preparano prima i bottom gel;
2) Preparare sotto cappa tante beutine da 100ml quanti sono i gel;
3) Versare in ciascuna beutina la quantità prevista di stock di acrilammide-bis acrilammide, tris-HCl pH 8,8 e SDS 10% precedentemente preparati (vedi tabella
soluzioni);
4) Immediatamente prima di versare il contenuto della beuta tra i vetri, aggiungere i due polimerizzanti (Ammonio persolfato e TEMED) precedentemente
preparati (vedi tabella soluzioni) ed agitare rapidamente;
5) Versare altrettanto rapidamente la soluzione tra i vetri montati fino ad arrivare a circa 3 cm dal bordo superiore dei vetri;
6) Versare alcune gocce di butanolo lungo il bordo superiore del bottom gel in modo da appianare la superficie;
7) Lasciare polimerizzare il gel per circa 45’;
8) A polimerizzazione avvenuta, prima di versare lo stacking gel, lavare la parte alta dei vetri per eliminare il butanolo con acqua distillata e asciugare con carta
bibula;
9) Preparare gli stacking gel;
10) Preparare sotto cappa tante beutine da 100ml quanti sono i gel;
11) Versare in ciascuna beutina la quantità prevista di acqua distillata, stock di acrilammide-bis acrilammide, tris-HCl pH 6,8 e SDS 10% precedentemente
preparati (vedi tabella soluzioni);
12) Immediatamente prima di versare il contenuto della beuta tra i vetri, aggiungere i due polimerizzanti (Ammonio persolfato e TEMED) precedentemente
preparati (vedi tabella soluzioni) ed agitare rapidamente;
13) Versare altrettanto rapidamente la soluzione tra gli stessi vetri in cui è già polimerizzato il bottom gel fino ad arrivare a circa 1 cm dal bordo superiore dei
vetri;
14) Inserire quindi il pettine diagonalmente e lentamente per non creare bolle, immergendolo nel gel per circa 1,5cm;
15) Il secondo gel richiede circa 20’ per la polimerizzazione;
16) A polimerizzazione avvenuta, sfilare delicatamente il pettine facendo attenzione a non danneggiare i pozzetti per la consistenza molto molle del gel
superiore;
17) Procedere con la pulizia dei pozzetti riempiendoli con tampone di corsa (Tris-Glicina-SDS) con una pipetta pasteur e aspirando lo stesso tampone con una
siringa con ago spuntato, facendo attenzione a non bucare i pozzetti;
18) Ripetere l’operazione di pulizia fino a quando non ci sono più residui di acrilammide non polimerizzati e i pozzetti appaiono ad angolo retto;
19) Procedere al caricamento dei campioni (circa 20µl), senza bisogno di un riferimento per la corsa, essendo essi già colorati;
20) Caricare i gel “a serpente” (da sinistra a destra il I davanti, da destra a sinistra il II dietro);
21) Sfilare i sandwich dalla base togliendo i due fissatori (neri) ed immergerli nella camera verticale. Nota: Assicurarsi che nella camera ci sia un adeguato
volume di tampone di corsa pari a circa i 2/3 del volume della camera;
22) Sistemare la vaschetta superiore dopo aver inserito nella sua parte inferiore una guarnizione di silicone e fissarla con gli stessi fissatori (neri) utilizzati per la
base inferiore;
23) Riempire la vaschetta superiore con la soluzione di corsa fino alla completa immersione dell’elettrodo metallico, chiudere la camera con il coperchio
superiore, collegare gli spinotti (il filo rosso al polo positivo (+) e il filo nero/azzurro al polo negativo (–)) ed accendere il refrigeratore (settato a 10°C) (LKB) e
l’alimentatore (LKB);
35
Precauzioni: accendere l’alimentatore solo dopo essersi assicurati di aver connesso tutti gli elementi con la giusta polarità. In caso di riversamento della soluzione
di corsa sul banco, prima spegnere l’alimentatore e poi procedere alla rimozione del liquido.
24) La corsa procede per circa 5 h a 60 mA per ciascuna camera (2 gel). Nota: Per le glutenine non è necessaria una pre-corsa e non bisogna invertire la polarità
degli spinotti;
25) A fine corsa, spegnere l’alimentatore e il refrigeratore, sfilare la vaschetta superiore versando il tampone in essa contenuto e sfilare i gel dalla camera;
26) Per recuperare il gel dai vetri svitare le viti e sfilare i fissatori laterali, con l’aiuto di una spatolina spingere all’esterno uno spessorino e, facendo leva con
l’altro spessorino, aprire i vetri in modo che il gel resti su uno dei due vetri. Nota: E’ conveniente eliminare ciò che resta dello stacking gel;
27) Poggiare il lato inferiore del vetro con il gel nella vaschetta contenente il colorante e, sollevando un angolo del gel con la spatolina, far scivolare
delicatamente il gel nel colorante;
28) La colorazione si realizza in vaschette con una soluzione di acido tricloroacetico (TCA) al 10%, Blue di Coomassie all’1% ed acido acetico glaciale (vedi tabella
soluzioni) che si lascia agire per una notte in lenta agitazione su un agitatore rotante;
30) Il lavaggio si esegue prima con acqua, e poi con TCA al 10%, ripetendolo più volte a distanza di ore sempre in lenta agitazione. Nota: Dopo il lavaggio, il gel
può essere preso a mani nude in modo da avere una migliore capacità prensile ed evitarne la rottura (piegandolo “a panino”);
Precauzioni: preparare il TCA sotto cappa con cautela ed usando guanti di protezione poiché è altamente corrosivo. Preparare il Blue di Coomassie con cautela
per non disperdere le polveri nell’ambiente.
31) Porre il gel su un transilluminatore a luce bianca e pulirlo spennellandolo;
32) Fotografare il gel con macchina Polaroid (GelCam) e pellicole 665-667 Polaroid su luce bianca, utilizzando il filtro di colore giallo specifico per il Blue di
Coomassie;
33) Bagnare con acqua una pellicola trasparente da dialisi, poggiarla e stenderla bene (in modo da eliminare tutta l’aria) su un vetro;
34) Sistemare il gel sul vetro e ricoprirlo con la pellicola eliminando l’aria;
35) Porre intorno al gel 4 bacchette di PVC trasparente in modo che aderiscano bene ai bordi del gel e fissarle con clips;
36) Lasciare asciugare ed essiccare il gel 4°C;
Togliere infine le bacchette e conservare i gel dopo aver eliminato la pellicola in eccesso.
Precauzione: usare guanti di protezione
36
SOLUZIONI PER METODO SDS-PAGE (GLUTENINE)
SOLUZIONI
PREPARAZIONE
Tris-HCl 1M
Sciogliere 121,1g di Tris base in 800ml di acqua distillata.
Aggiustare il pH fino al valore desiderato aggiungendo HCl
concentarato tenendo il beker su un agitatore. Considerare che:
pH=7.4
70ml HCl
pH=7.6
60ml HCl
pH=8.0
42ml HCl
Portare a volume con acqua distillata. Autoclavare.
Sample Buffer
Soluzione di estrazione
Stock di Bis-acrilammide/Acrilammide
COMMENTI/PRECAUZIONI
Se la soluzione 1M è di colore giallo, non utilizzarla
perché il tris è di qualità scadente.
Il pH della soluzione tris è funzione della
temperatura e si riduce approssimativamente di 0,03
unità di pH per ciascun grado di incremento della
temperatura. Per esempio, a 0,005 M la soluzione ha
pH di 9,5, 8,9 e 8,6 a 5°C, 25°C e 37°C
rispettivamente.
Aliquotare in un beker: 2g di SDS, 15 mg di Blu-bromofenolo, 6,25 Pesare le sostanze sotto cappa.
ml di Tris-HCl 1M pH 6.8, 12,05 ml di acqua distillata e 10 ml Indossare sempre una maschera quando si prepara
glicerolo.
l'SDS e pulire bene la bilancia ed il piano di pesata
poiché i cristalli sono molto leggeri e si disperdono
facilmente.
Aliquotare: 4,5 ml di Sample Buffer, 2,5 ml di Dimetil- Aliquotare le sostanze sotto cappa.
formammide, 1,5 ml di β-Mercaptoetanolo e 4,25 ml di acqua Il β-Mercaptoetanolo emana un odore penetrante
distillata.
che irrita le mucose nasali. Va connservato a 4°C in
una bottiglia scura.
Per 600 ml di soluzione: sciogliere 1,356g di Bis- L'acrilammide è neurotossica. Allo stato solido può
acrilammide e 105g di Acrilammide. Portare a volume essere inalata, mentre allo stato liquido può essere
assorbita per via cutanea. Pesare sempre sotto cappa
con acqua distillata. Filtrare con carta da filtro indossando camice, guanti e mascherina.
(Whatman N.1) in una bottiglia da 1 litro scura o
coperta con carta di alluminio, porre nel liofilizzatore e
37
degassare per 5-10 minuti.
Ammonio persolfato (APS) 1%
SDS 10%
Bottom gel
Stacking gel
Sciogliere 0,1g (100mg) in 10ml di acqua distillata
Va preparato al momento e conservato a 4°C e al
buio.
Sciogliere 100g di SDS in 900ml di acqua distillata. Riscaldare a Indossare sempre una maschera quando si prepara
68°C per favorire lo scioglimento. Aggiustare il pH fino al valore l'SDS e pulire bene la bilancia ed il piano di pesata
di 7,2 con gocce di HCl concentrato. Portare a volume di 1 litro.
poiché i cristalli sono molto leggeri e si disperdono
facilmente.
In una beutina per ciascun gel aliquotare:26 ml di stock di Aliquotare le sostanze sotto cappa.
acrilammide, 17 ml di Tris-HCl 1M pH 8.8, 4,6 ml di APS 1%, 0,92
ml di SDS 10%, 89µl di TEMED.
In una beutina per ciascun gel aliquotare:7 ml di acqua Aliquotare le sostanze sotto cappa.
distillata, 1,25 ml di Tris-HCl 1M pH 6.8, 2,6 ml di
stock di acrilammide, 0,1 ml di SDS 10%, 1,7 ml di APS
1%, 50µl di TEMED.
Tris-Glicina-SDS
Blue di Coomassie (0,5%)
Acido Tricloroacetico (TCA) al 100%
Acido Tricloroacetico (TCA) al 10%
Colorante per i gel
Per 5 litri di soluzione: Pesare 70,5g di Glicina, 15g di Trizma base Pesare le sostanze sotto cappa.
e5g di SDS.
Indossare sempre una maschera quando si prepara
l'SDS e pulire bene la bilancia ed il piano di pesata
poiché i cristalli sono molto leggeri e si disperdono
facilmente.
Sciogliere 1g di Blue di Coomassie in 200 ml di metanolo.
Preparare il Blue di Coomassie con cautela per non
disperdere le polveri nell’ambiente.
Ad una bottiglia contenente 500g di TCA, aggiungere 227ml di
acqua distillata. Agitare più volte per favorire lo scioglimento. La
soluzione risultante conterrà TCA al 100% peso/vol.
Aliquotare 100ml di TCA al 100% e portare il volume ad 1 litro
con acqua distillata. Agitare.
Per 800ml di soluzione: aggiungere ai 200ml di Blue di Coomassie,
500 ml di TCA 10% e 70 ml di Acido acetico glaciale. Portare a
volume con acqua distillata ed agitare.
Preparare il TCA sotto cappa con cautela ed usando
guanti di protezione poiché è altamente corrosivo.
Preparare il TCA sotto cappa con cautela ed usando
guanti di protezione poiché è altamente corrosivo.
Aliquotare le sostanze sotto cappa.
38
ALLEGATO 2
ESTRAZIONE DI DNA DA TESSUTI VEGETALI
(metodo pratico molto appropriato per le graminacee)
1) Accendere il bagnetto termostatato e settarlo a 65°C;
2) Preparare il tampone di estrazione senza aggiungere bisolfito di sodio e riscaldare a 65°C;
3) Immediatamente prima della sua utilizzazione, aggiungere 0,38g di Bisolfito di sodio per ogni 100 ml di tampone di estrazione, precedentemente preparato, e
aggiustare il pH a 7,8-8;
4) Macinare in un mortaio tramite pressione con pestello, in presenza di un’aliquota del tampone di estrazione, circa 1g di tessuto vegetale per campione e
versare la poltiglia in falcon da 50 ml aiutandosi con una spatolina. Portare il volume con il tampone di estrazione a circa 5ml. Nota: Lavorare sotto cappa.
5) Sotto cappa, aggiungere quindi ai campioni una quantità della miscela Cloroformio/Alcool Isoamilico (nel rapporto di 24ml : 1ml) ed agitare dolcemente con
le mani., fino al raggiungimento di un volume di circa 40 ml.
Precauzione: la miscela va preparata sotto cappa, evitando il contatto con la pelle poiché il cloroformio è altamente corrosivo.
6)
7)
8)
9)
10)
11)
12)
Porre le provette ben chiuse in bagnomaria a 65°C per 15’;
Centrifugare a 2.800 giri per 15’ e a temperatura ambiente, recuperare il surnatante;
Versare il surnatante in nuove provette falcon;
Aggiungere 2 volumi (fino ad arrivare a circa 50 ml) di etanolo al 95% (mantenuto a –20°C);
Uncinare quindi il DNA con Pasteur di vetro e lavarlo con etanolo al 70% per almeno tre volte utilizzando provette eppendorf da 1,5ml;
Lasciare asciugare il pellet e porlo in provette da 1,5 ml con circa 1 ml di TE 1X sterile;
Facilitare il dissolvimento del DNA, capovolgendo le provette più volte, tramite agitatore orbitale.
N.B.Nel caso in cui si effettua una seconda estrazione con cloroformio mediante aggiunta sotto cappa di 0,5 ml di cloroformio/alcol isoamilico, agitare
dolcemente con mani o vortex e centrifugare a 2500 giri per 10’. Recuperare il surnatante che contiene il DNA e conservarlo in nuove provette da 1,5 ml.
Precauzione: tutti i solventi organici (es. cloroformio) vanno versati sotto cappa nell’apposito contenitore per lo smaltimento dei tossici speciali.
39
SOLUZIONI PER L’ESTRAZIONE DEGLI ACIDI NUCLEICI
SOLUZIONI
PREPARAZIONE
Tampone di estrazione
Per 100 ml di soluzione: Aliquotare:10 ml di NaCl
5M,, 10 ml di Tris-HCl 1M pH 8.0, 10 ml di EDTA
0,5M, 6,25 ml di SDS 10%
Cloruro di sodio (NaCl) 5M
sciogliere 292,2g di NaCl in 1 litro di acqua distillata
Tris-HCl pH 8 1M
Sciogliere 121,1g di Tris base in 800ml di acqua
distillata. Aggiustare il pH fino al valore desiderato
aggiungendo HCl concentarato tenendo il beker su
un agitatore. Considerare che:
pH=7.4
70ml HCl
pH=7.6
60ml HCl
pH=8.0
42ml HCl
Portare a volume con acqua distillata. Autoclavare.
EDTA 0,5M
SDS 10%
Ammonio Acetato 5M
Miscela Cloroformio/alcool isoamilico
sciogliere 186,1g di EDTA in polvere in 800ml di
acqua distillata. Aggiustare il pH fino a portarlo al
valore di 8 con NaOH (circa 20g di NaOH in perle).
Autoclavare
sciogliere 100g di SDS in 900ml di acqua distillata.
Riscaldare a 68°C per favorire lo scioglimento.
Aggiustare il pH fino al valore di 7,2 con gocce di
HCl concentrato. Portare a volume di 1 litro
COMMENTI/PRECAUZIONI
Aggiungere 0,38g di Bisolfito di sodio per ogni 100
ml di tampone di estrazione al momento della sua
utilizzazione
L’autoclavaggio è facoltativo
Se la soluzione 1M è di colore giallo, non utilizzarla
perché il tris è di qualità scadente.
Il pH della soluzione tris è funzione della
temperatura e si riduce approssimativamente di 0,03
unità di pH per ciascun grado di incremento della
temperatura. Per esempio, a 0,005 M la soluzione ha
pH di 9,5, 8,9 e 8,6 a 5°C, 25°C e 37°C
rispettivamente.
Questo sale non si scioglierà a meno che il pH della
soluzione non sia circa 8 mediante aggiunta di
idrossido di sodio.
Indossare una mascherina durante la pesatura di
questa sostanza e pulire la superficie su cui si
effettua la pesatura poiché i cristalli di SDS essendo
molto piccoli e volatili si disperdono facilmente.
Non è necessario autoclavare questa soluzione.
Sciogliere 385g di acetato d'ammonio in 800ml di
acqua distillata. Portare a volume di 1 littro.
Sterilizzare mediante filtrazione
Per 1 litro: 975ml di cloroformio+25ml di alcool Operare sotto cappa aggiungendo in un cilindro
isoamilico
prima il cloroformio e poi l’alcool. Non agitare ed
usare guanti e camice di protezione.
40
VALUTAZIONE DELLA CONCENTRAZIONE DEL DNA GENOMICO
E’ possibile valutare la concentrazione del DNA genomico in due diversi modi:
A) Quantificazione UV (usando spettrofotometro Beckman);
B) Quantificazione con marcatore a concentrazione nota in gel di agarosio 0.8%.
A) Quantificazione UV (usando spettrofotometro Beckman)
1) I campioni di DNA da valutare vanno diluiti nel rapporto 1:50 ( 20uL di DNA stock: 980uL di
acqua dd).
2) Accendere lo spettrofotometro ed inserire il modulo di lettura per gli ac. Nucleici, quindi
accendere la stampante.
3) Dopo che gli UV si sono accesi (uv →UV) procedere alla lettura di ciascun campione, posto
nell’apposita cuvetta di quarzo, alla lunghezza di 260 nm (λ) secondo le istruzioni riportate
dal manuale.
NB Lo strumento deve essere sempre tarato prima di procedere alla lettura, utilizzando come
bianco l’acqua dd.
OD 260x 50 (fattore di diluizione) x50 ug/mL
Concentrazione DNA (ug/uL)= -----------------------------------------------------------2x1000
OD260 = lettura spettrofotometrica di λ a 260nm
50= fattore di diluizione del campione
50 ug/mL= quantità di DNA corrispondente ad una unità di OD (assorbanza)
2= fattore di correzione
NB Per determinare il grado di purezza del campione occorre valutare il rapporto OD260/OD280:
• se questo ha valore compreso tra 1,8-2.0 l’assorbanza è dovuta ad ac. Nucleici
• se questo rapporto è < a 1,8 il campione è sporco ( presenza di proteine o sostanze estranee)
• se questo rapporto è > di 2.0 il campione è contaminato da cloroformio, fenolo e va
riprecipitato con etanolo.
B) Quantificazione con marcatore a concentrazione nota in gel di agarosio 0.8%
1) Una volta estratti, i campioni di DNA vengono fatti correre per 1 ora alla tensione di 80V e
all’intensità di corrente di 60mA su gel di agarosio allo 0.8% in camere elettroforetiche
orizzontali con buffer di corsa TBE 0.5X utilizzando come marker di riferimento il DNA
digerito del fago λ (Gene RulerTM 100bp DNA ladder 0.5ng/µl) a diverse concentrazioni (50,
100, 200 ng/µl).
2) In ogni pozzetto sono stati caricati 2µl del DNA del campione da analizzare, 4µl di colorante
LB (loading buffer) 1X per appesantire il campione e 6µl di acqua bi-distillata. I campioni
sono stati visualizzati utilizzando etidio bromuro come rivelatore (10mg/mL).
3) La determinazione della concentrazione consiste nel confronto “visivo” tra intensità della
banda del campione del DNA e l’intensità delle bande del marker (DNA λ). Questo metodo
permette anche di valutare la qualità del DNA estratto che è maggiore quanto più la banda
vicino al pozzetto è netta e priva di altri segnali di background dovuti alla presenza di RNA o
altri prodotti di degradazione.
4) Per le successive analisi, il DNA è stato diluito alla concentrazione di lavoro.
41
SOLUZIONI STOCK PER LA QUANTIFICAZIONE DEL DNA
SOLUZIONE
METODO DI PREPARAZIONE
TBE 5x
Aggiungere 54g di Tris base + 27g di acido borico +
20ml di EDTA 0,5M pH 8. Portare a 1litro.
Agarosio
40 g in 500 mL di TBE 0.5X
100bp DNA ladder 0.5ng/µl (Gene RulerTM, Genenco ) Diluito con acqua e LB 6X (fornito dalla Genenco)
(1:1:3)
Bromuro di etidio (10 mg/ml)
Aggiungere 1 g di bromuro di etidio a 100 ml di
acqua. Agitare con magnetino per diverse ore
assicurandosi che tutta la sostanza si sia dissolta.
Coprire il contenitore con alluminio oppure
trasferire la soluzione in un contenitore scuro.
PRECAUZIONI E COMMENTI
Usare guanti perché soatanze irritanti
Usare guanti
Usare guanti
L’etidio bromuro è un potente mutageno ed è
moderatamente tossico.
I guanti dovrebbero essere indossati ogni volta che
si operi con soluzioni o gel che contengono questa
sostanza.
La mascherina deve usarsi quando si prepara la
soluzione base alla concentrazione di 10 g/ml.
Dopo l’uso, le soluzioni che contengono anche
traccie di questa sostanza devono essere
decontaminate con il carbone attivo.
42
ALLEGATO 3
PROTOCOLLO PER L’AMPLIFICAZIONE DI MARCATORI MICROSATELLITI
AMPLIFICAZIONE SSR DI TRITICUM DURUM
•
•
•
Il DNA di frumento, dopo essere stato quantificato, viene diluito alla concentrazione di 20ng/µl, per la sua amplificazione.
Ogni reazione di amplificazione è fatta utilizzando piastre microtiter a 96 pozzetti sterili in cui sono stati caricati, in ciascun pozzetto, 20µl di Master
Mix e 5µl di DNA di ciascun campione da amplificare per un volume finale di 25µl per campione, utilizzando il termociclizzatore della Biorad
iCycler.
La mix di reazione contiene i seguenti reagenti e le seguenti quantità e concentrazioni (nell’esempio sono stati considerati 100 campioni):
REAGENTI
STOCK
25ul
Q/CAMP.(25ul)
Buffer
MIX
dNTP
(A+C+T+G)
Taq
(enzima
polimerasi)
MgCl2 (cloruro di
magnesio)
MIX-PRIMERS
(F+R)
10 x
10 mM
1X
0,25mM
2,5 ul
0,625 ul
x
x
N° CAMPIONI
n° variabile*
100
=
100
=
5U/ul
1U/ul
0,2 ul
x
100
25 mM
2,5mM
2,5 ul
x
5uM
0,2uM
1ul
x
TOTALE
250 ul
62,5 ul
+
+
=
20ul
+
100
=
250ul
+
100
=
100ul
+
x 100
=
1317,5 ul =
Tot. 6,825 ul
H2O dd sterile
20ul-6,825=13,175
2000ul tot.mix
*Preparare la mix di reazione per un numero di campioni aumentati dell'1% rispetto al necessario per superare eventuali errori di
pipettamento.
• Per ogni primers c’è una temp. di “annealing” diversa quindi un diverso programma della macchina PCR.
•
•
Un programma di amplificazione classico per il DNA di frumento impostato nel termociclizzatore della durata di circa 3 h 20', prevede: da 3 a 7' a 95°C (a
seconda della Taq utilizzata), 3 fasi (1' a 94°C, 1' alle temperature di annealing da 47 a 61°C - a seconda dei primers - ed 1' a 72°C) ripetute per 45 cicli, 5' a 72°C
ed infine 4°C per un tempo indeterminato.
Una volta amplificati,i campioni possono essere visualizzati in gel orizzontali di agarosio o in gel verticali di acrilammide (in quest’ultimo caso la
separazione delle bande è migliore).
43
PROTOCOLLO PER L’AMPLIFICAZIONE DI RAPDs
•
•
•
Il DNA di tartufo, dopo essere stato estratto e quantificato, viene diluito alla concentrazione di 2ng/µl, per la sua amplificazione.
Ogni reazione di amplificazione è fatta utilizzando piastre microtiter a 96 pozzetti sterili in cui sono stati caricati, in ciascun pozzetto, 15µl di Master
Mix e 10µl di DNA di ciascun campione da amplificare per un volume finale di 25µl per campione, utilizzando il termociclizzatore della Biorad
iCycler.
La mix di reazione contiene i seguenti reagenti e le seguenti quantità e concentrazioni (nell’esempio sono stati considerati 100 campioni):
REAGENTI
STOCK
25ul
Q/CAMP.(25ul)
Buffer
MIX
dNTP
(A+C+T+G)
Taq
(enzima
polimerasi)
MgCl2 (cloruro di
magnesio)
PRIMER
10 x
10 mM
1X
0,25mM
2,5 ul
0,625 ul
x
x
N° CAMPIONI
n° variabile*
100
=
100
=
5U/ul
1U/ul
0,2 ul
x
100
50 mM
4 mM
2 ul
5uM
0,2uM
1ul
x
x
TOTALE
250 ul
62,5 ul
+
+
=
20ul
+
100
=
200ul
+
100
=
100ul
+
x 100
=
867,5 ul =
Tot. 6,325 ul
H2 O dd sterile
15ul-6,325=8,675
1500ul tot.mix
*Preparare la mix di reazione per un numero di campioni aumentati dell'1% rispetto al necessario per superare eventuali errori di
pipettamento.
•
•
•
Per ogni primers c’è una temp. di “annealing” diversa quindi un diverso programma della macchina PCR.
Un programma di amplificazione classico per il DNA di tartufo impostato nel termociclizzatore della durata di circa 3 h 20', prevede: 3’ a 95°C, 3 fasi (1' a
94°C, 1' alla temperatura di annealing da 45°C ed 1' a 72°C) ripetute per 45 cicli, 5' a 72°C ed infine 4°C per un tempo indeterminato.
Una volta amplificati,i campioni possono essere visualizzati in gel orizzontali di agarosio al 2% (5g agarosio in 250ml TBE 0,5X) ed essere incamerati su
computer per mezzo di un sistema di acquisizione di immagini (KODAK ID) e salvati come file d’immagine, estensione JPEG.
44
ELETTROFORESI ORIZZONTALE SU GEL DI AGAROSIO
(cassette Pharmacia)
1.
Preparare la slitta, che supporta il gel, sigillando il lato superiore ed inferiore con scotch cartaceo, onde evitare che l’agarosio liquido fuoriesca, e posizionare
il pettine nella posizione indicata dalle camere.
2. Verificare se la cassetta elettroforetica è a livello col piano di lavoro.
3. Preparare il gel di agarosio : es. per un gel alla concentrazione dell’0.8 pesare in una beuta a collo largo 4 g di agarosio in polvere a cui vanno aggiunti circa
500 ml di TBE 0,5x.
Nota: La concentrazione dello 0,8% consente la quantificazione di DNA genomico, concentrazioni tra 1,5 e 2% consentono la separazione dei frammenti
amplificati con la PCR.
4. Dopo aver mescolato la soluzione, porre la beuta nel fornetto a microonde fino a completo scioglimento dell’agarosio (circa 3 min), farlo raffreddare sotto un
getto d’acqua fredda ruotando la beuta tra le mani.
5
Aggiungere 5ul di Bromuro d’Etidio (preparato alla conc. di 10mg/ml).
Precauzione: operare sotto cappa usando guanti e maschera per non respirare i vapori di etidio che è un potente mutageno.
6) Versare il gel nella slitta (avendo cura di non far fare bolle) ed attendere almeno 1 ora prima che si solidifichi.
7)Togliere lo schotch e mettere la slitta nella cassetta elettroforetica, riempirla di TBE 0,5x fino a coprire con un strato sottile il gel (circa 2 litri per camera).
8) Caricare i campioni nel gel, mettere al 1° posto il lambda digerito con HIND III (3-5ul) o un altro marker di riferimento.
9) Chiudere la cassetta e collegare il filo rosso al polo positivo (+) e il filo nero/azzurro al polo negativo (–)
10) Accendere l’alimentatore (voltaggio max 100-110V) per almeno 2-3 ore.
11) Spegnere quando i campioni hanno percorso il gel fino ad una distanza max di 2 cm dal bordo inferiore.
12) Porre il gel sul transilluminatore UV, abbassare il coperchio e visualizzare la corsa, dopo aver accesso il bottone degli UV.
13) Fotografare indossando la maschera di protezione da raggi UV (pellicole Polaroid 665/667), utilizzando il filtro di colore arancione specifico per il bromuro
di etidio;
Precauzione: usare occhiali protettivi.
14) I gel possono essere incamerati su computer per mezzo di un sistema di acquisizione di immagini (KODAK ID) e salvati come file d’immagine, estensione
JPEG.
45
SOLUZIONI STOCK ELETTROFORESI ORIZZONTALE
SOLUZIONE
METODO
DI MODALITA’
DI
PREPARAZIONE
CONSERVAZIONE
TE 100x
Sciogliere 121,1g di Tris e Conservare a temp. Ambiente
37,2g di EDTA in 800ml di H2
O.Raggiungere il pH 8,0 con
HCl e portare a 1 litro con H2
O distillata. Autoclavare.
Bromuro di etidio (10 Aggiungere 1 g di bromuro di Conservare a
mg/ml)
etidio a 100 ml di acqua. temperatura ambiente.
Agitare con magnetino per
diverse ore assicurandosi che
tutta la sostanza si sia dissolta.
Coprire il contenitore con
alluminio oppure trasferire la
soluzione in un contenitore
scuro.
a
temper.
TBE 5x
Pesare 54g di Tris base, 27g di Conservare
acido borico e 20ml di EDTA Ambiente
0,5M pH 8. Portare a
1litro.Autocl.
PRECAUZIONI
Usare guanti perché Trizma ed EDTA sono irritanti. Quando si
aggiunge HCl lo si deve fare sotto cappa indossando la
mascherina.
L’etidio bromuro è un potente mutageno ed è moderatamente
tossico.
I guanti dovrebbero essere indossati ogni volta che si operi con
soluzioni o gel che contengono questa sostanza.
La mascherina deve usarsi quando si prepara la soluzione base
alla concentrazione di 10 g/ml. Dopo l’uso, le soluzioni che
contengono anche traccie di questa sostanza devono essere
decontaminate con il carbone attivo.
Usare guanti perché le sostanze sono irritanti.
46
ELETTROFORESI VERTICALE SU GEL DI ACRILAMMIDE PER SSR DI FRUMENTO DURO
La separazione degli amplificati SSR di frumento duro avviene in un gel di acrilammide non denaturante all’8% con elettroforesi
verticale (camere elettroforetiche SE 600 - Pharmacia Biotech), in TBE 1X. Si preparano gel a sandwich con vetri di dimensioni 16cm x
18cm x 0,3cm separati da appositi spaziatori (spessorini) in PVC dello spessore di 0,3 cm e con pettini in PVC dello spessore di 0,3 cm.
Preparazione dei vetri e delle relative basi
1) Lavare i vetri con etanolo per togliere le impurità e far aderire meglio gli spessorini;
2) Prevedere due vetri per ciascun gel che si vuol preparare;
3) Allineare i vetri poggiandoli sul piano di lavoro e farli combaciare perfettamente nella loro superficie e lungo ciascun lato;
4) Inserire tra i vetri due spessorini (uno lungo ciascun lato verticale) e risistemare i vetri in modo che combacino perfettamente;
5) Inserire due sistemi di fissaggio laterali dotati di viti lungo i due lati verticali del sandwich ottenuto, stringendo completamente le viti solo dopo essersi
accertati della perfetta sistemazione dei vetri e degli spessorini;
6) Inserire la parte inferiore del sandwich nella base orizzontale (dopo aver inserito in quest’ultima una guarnizione di silicone) e incastrarla perfettamente;
7) Inserire infine, ai due lati e negli appositi alloggi, due fissatori (pomellini neri) e ruotarli fino ad assumere una posizione verticale in modo da stringere
perfettamente il sandwich e non consentire la fuoriuscita del gel.
Nota: ricordare che ciascuna base orizzontale ha due alloggi per i gel e che anche le camere verticali prevedono la corsa di due gel contemporaneamente, per cui
occorre preparare due gel per base e/o camera, oppure, se si vuol preparare un solo gel, utilizzare l’apposito spessore in PVC trasparente che simula il secondo
gel.
8) Prevedere un pettine dello spessore di 0,3cm per ciascun gel.
Preparazione dei gel e condizioni di corsa
1) Preparare sotto cappa tante beutine da 100ml quanti sono i gel;
2) Versare in ciascuna beutina la quantità prevista di acqua, di stock di acrilammide-bis acrilammide e TBE5X precedentemente preparati (vedi tabella
soluzioni);
3) Immediatamente prima di versare il contenuto della beuta tra i vetri, aggiungere i due polimerizzanti (Ammonio persolfato e TEMED) precedentemente
preparati (vedi tabella soluzioni) ed agitare rapidamente;
4) Versare altrettanto rapidamente la soluzione tra i vetri montati fino ad arrivare a circa 1 cm dal bordo superiore dei vetri;
5) Inserire quindi il pettine diagonalmente e lentamente per non creare bolle, immergendolo nel gel per circa 1,5cm;
6) Attendere quindi circa 45 minuti per la polimerizzazione, senza muovere o spostare il gel;
7) A polimerizzazione avvenuta, sfilare delicatamente il pettine;
8) Procedere con la pulizia dei pozzetti riempiendoli con tampone di corsa (TBE 1X) con una pipetta pasteur e aspirando lo stesso tampone con una siringa con
ago spuntato, facendo attenzione a non bucare i pozzetti;
9) Ripetere l’operazione di pulizia fino a quando non ci sono più residui di acrilammide non polimerizzati e i pozzetti appaiono ad angolo retto;
10) Colorare i campioni con 8µl/campione di Loading Buffer 12X;
11) Procedere al caricamento dei campioni (circa 20µl);
47
12) Caricare i gel “a serpente” (da sinistra a destra il I davanti, da destra a sinistra il II dietro), caricando nei primi pozzetti i riferimenti. Come standard di
riferimento per gli SSR possono essere usati il “DNA Lambda" Pst I Digest” (29 frammenti: 15-11.497 bp) (SIGMA) ed il “PCR 100 bp Low Ladder” (10
frammenti: 100-1000 bp) (SIGMA);
13) Sfilare i sandwich dalla base togliendo i due fissatori (neri) ed immergerli nella camera verticale.
Nota: assicurarsi che nella camera ci sia un adeguato volume di tampone di corsa pari a circa i 2/3 del volume della camera;
14) Sistemare la vaschetta superiore dopo aver inserito nella sua parte inferiore una guarnizione di silicone e fissarla con gli stessi fissatori (neri) utilizzati per la
base inferiore;
15) Riempire la vaschetta superiore con la soluzione di corsa fino alla completa immersione dell’elettrodo metallico, chiudere la camera con il coperchio
superiore, collegare gli spinotti (il filo rosso al polo positivo (+) e il filo nero/azzurro al polo negativo (–)) ed accendere il refrigeratore (settato a 4°C) (LKB) e
l’alimentatore (LKB);
Precauzioni: accendere l’alimentatore solo dopo essersi assicurati di aver connesso tutti gli elementi con la giusta polarità. In caso di riversamento della soluzione
di corsa sul banco, prima spegnere l’alimentatore e poi procedere alla rimozione del liquido.
16) La corsa procede per 200-250V per circa 3h;
17) A fine corsa, spegnere l’alimentatore e il refrigeratore, sfilare la vaschetta superiore versando il tampone in essa contenuto e sfilare i gel dalla camera;
18) Per recuperare il gel dai vetri svitare le viti e sfilare i fissatori laterali, con l’aiuto di una spatolina spingere all’esterno uno spessorino e, facendo leva con
l’altro spessorino, aprire i vetri in modo che il gel resti su uno dei due vetri;
19) Poggiare il lato inferiore del vetro con il gel nella vaschetta contenente il colorante e, sollevando un angolo del gel con la spatolina, far scivolare
delicatamente il gel nel colorante;
20) La colorazione si realizza in vaschette di plastica contenenti una soluzione acquosa (10µl in 200ml di acqua) di bromuro di etidio (10mg/ml);
21) Coprire le vaschette con carta di alluminio e porle per 45' su agitatore rotante;
22) Fotografare i gel con pellicole Polaroid 665-667 su transilluminatore a raggi UV, utilizzando il filtro di colore arancione specifico per il bromuro di etidio;
Precauzioni: La colorazione va realizzata sotto cappa. Per la fotografia sul transilluminatore indossare occhiali o maschera protettiva.
23) I gel possono essere incamerati su computer per mezzo di un sistema di acquisizione di immagini (KODAK ID) e salvati come file d’immagine, estensione
JPEG.
48
SOLUZIONI STOCK PER ELETTROFORESI VERTICALE CON ACRILAMMIDE
(SSR FRUMENTO DURO)
SOLUZIONE
METODO DI PREPARAZIONE
MODALITA’
CONSERVAZIONE
DI PRECAUZIONI
Stock acrilammide/
bis-acrilammide al 30%
Per 1 litro di soluzione, pesare 290g Conservare a 4-6 °C in bottiglia Usare guanti, mascherina e lavorare sotto cappa
di Acrilammide e 10g di Bis- scura
acrilammide. Si aggiungono prima
tanti ml di acqua dd sterile quanti
bastano
per
consentire
lo
scioglimentodelle sostanze, lasciando
agitare la soluzione. Successivamente
si porta a volume. Si filtra con carta da
filtro in una bottiglia da 1 litro e si
pone nel liofilizzatore a degassare per
5-10 minuti.
Gel di poliacrilammide
Per un singolo gel aliquotare: 36,8 ml
di acqua sterile, 18,62 ml della
soluzione
acrilammide/bisacrilammide, 14 ml di TBE 5X, 500ul
di Ammonio Persolfato al 10% e 30µl
di TEMED.
Bromuro di etidio (10 mg/ml)
Aggiungere 1 g di bromuro di etidio a Conservare
100 ml di acqua. Agitare con ambiente.
magnetino
per
diverse
ore
assicurandosi che tutta la sostanza si
sia dissolta. Coprire il contenitore con
alluminio
oppure
trasferire
la
soluzione in un contenitore scuro.
Ammonio persolfato
TEMED
1g in 10 mL di volume finale
Loading Buffer 12X
Per un volume di 25ml pesare 0,062g
Aliquotare le sostanze sotto cappa.
Si usa fresco
a
temperatura L’etidio bromuro è un potente mutageno ed è
moderatamente tossico.
I guanti dovrebbero essere indossati ogni volta che
si operi con soluzioni o gel che contengono questa
sostanza.
La mascherina deve usarsi quando si prepara la
soluzione base alla concentrazione di 10 g/ml.
Dopo l’uso, le soluzioni che contengono anche
traccie di questa sostanza devono essere
decontaminate con il carbone attivo.
Usare guanti perché corrosivo
Usare guanti e lavorare sotto cappa perché
irritante per le mucose nasali
Usare guanti
49
TBE 5x
di Blu di Bromofenolo allo 0,25%,
0,062g di Xilene Cianolo allo 0,25%,
7,5g di Ficoll 400 al 30%.
Pesare 54g di Tris base, 27g di acido Conservare a temper. Ambiente Usare guanti perché sostanze irritanti
borico e 20ml di EDTA 0,5M pH 8.
Portare a 1litro.Autocl.
50
ANALISI AFLP
Protocollo per la determinazione di frammenti AFLP utilizzando DNA di frumento (modifica del protocollo di Vos et al., 1995),
• Il DNA di frumento, dopo essere stato quantificato visivamente su gel di agarosio all’0.8%, viene diluito alla concentrazione di 15-20 ng/µl, per la sua
amplificazione.
L’analisi degli AFLP comprende le seguenti fasi:
Digestione
250ng/uL of DNA sono digeriti con 2,5U di EcoRI e 2,5U MseI in un volume di reazione di 25 uL
(per ciascun campione x N° campioni)
DNA working
20 uL
Buffer 10X OPA (Pharmacia)
2,5 uL
Eco Ri (20U/uL)
0.125 uL
MseI (10U/uL)
0.25 uL
Acqua distillata
2.125 uL
La digestione viene fatta per circa 2h a 37°C
Dopo aver verificato su gel di agarosio all’1.5% la digestione (viene fatta foto che sarà conservata in formato jpeg); si procede alla ligazione.
1. Ligazione degli adattatori
x ciascun campione
x N° campioni
DNA digerito
20 uL
Buffer 10X OPA (Pharmacia)
0.5uL
10mM di ATP (NEB)
0.5 uL
Eco-adattatore 2.5uM
0.5 uL
Mase-adattatore 25uM
0.5 uL
T4 ligasi 1U (Pharmacia)
0.01 uL
Acqua distillata
2.99 uL
La mistura “restriction ligation” viene incubata per almeno 12 h at 37°C .
2. Amplificazione pre-selettiva
(per ciascun campione x N° campioni)
DNA prodotto dalla ligazione
2 uL
2xCoremix* (Applera)
10 uL
Taq DNA polimerasi (0.4U) (Applera)
0.08 uL
51
Primer preselettivi 30ng/uL (Pharmacia)
Acqua
6.92uL
1.0 uL
2x core mix (20mM Tri-HCl pH 8.3, 3mM MgCl2, 100mM KCl, 0.2mM dNTPs)
3.1 PCR pre-selettiva:
Attivazione della polimerasi a 95°C x 10 min
denaturazione: 94°C, 30 sec;
annealing: 56°C, 30 sec.
Estensione: 72°C, 1 min,
cicli ripetuti per 35 volte
Verifica amplificazione pre-selettiva su gel di agarosio 1.5% (conservazione della foto del gel in formato jpeg).
Diluizione degli amplificati con TE0.1 (1:9).
3.2 Amplificazione selettiva
(per ciascun campione x N° campioni)
DNA diluito
2.5 uL
2xCoremix* (Applera)
5 uL
Taq DNA polimerasi (0.4U)
0.04 uL
Primer selettivo Eco-XXX 1uM
0.4 uL
Primer selettivi Mse-XXX 5uM
0.6 uL
Acqua
1.5 uL
2x core mix (20mM Tri-HCl pH 8.3, 3mM MgCl2, 100mM KCl, 0.2mM dNTPs)
4.1 PCR selettiva:
Attivazione della polimerasi a 95°C x 10 min
denaturazione: 94°C, 30 sec;
annealing: 65°C*, 30 sec.
Estensione: 72°C, 1 min,
cicli ripetuti per 12 cicli
ad ogni ciclo la temperatura diminuisce di 0.7°C
denaturazione: 94°C, 30 sec;
annealing: 56°C, 30 sec.
Estensione: 72°C, 1 min,
cicli ripetuti per 19 cicli
52
Precauzioni: usare guanti e camice
Per quanto riguarda la corsa elettroforetica degli SSR e degli AFLP vedere protocollo del sequenziatore ABI PrismTM 377 o di versioni più recenti come AB3100
53
SOLUZIONI STOCK Analisi AFLP
Soluzione
Metodo di preparazione
TE0.1
0.5mLTrisHCl 1M
0.01mL EDTA
per un volume finale di 50mL
Commento e precauzione
Usare guanti e mascherina perché nocivo per ingestione
(EDTA)
Usare guanti e lavorare sotto cappa perché TRIZMA è nocivi
per ingestione e per gli occhi.
Portare a pH sotto cappa perché i vapori di HCl sono irritanti
per le mucose nasali
54
CORSA ELETTROFORETICA AL SEQUENZIATORE
Per l’automazione nell’analisi di frammenti di DNA (AFLP e/o SSR etc) è utilizzato il Sequenziatore ABI PrismTM 377.
Modalità d’uso
1) Accensione apparecchio
2) Assemblaggio coppia di vetri
3) Preparazione del gel
4) Preparazione campioni
5) Corsa elettroforetica.
6) Manutensione dello strumento e pulizia vetri
1-2) Dopo aver acceso lo strumento si procede all’assemblaggio della coppia di vetri “a sandwich” manipolando gli stessi con cura.
Precauzioni: evitare che i vetri si scheggino o si rompino perché ciò può provocare tagli o ferite.
3) Il gel si ottiene mescolando 30 mL di ReproGelTM 377 solution ,costituito dalle soluzioni A e B, che vengono usate nel rapporto 1:1 un gel di 42 cm di lunghezza e
0.3 mm di spessore. Questa soluzione viene versata tramite una siringa da 50 ml nei due vetri assemblati..
I vetri supportati dal casting sono posti sotto un sistema di polimerizzazione ad UV (ReproSetTM dell’Amersharm Pharmacia BiotecTM) che ha la funzione di
accelerare la polimerizzazione (circa 10 minuti) trascorsi i quali il gel viene pulito da eventuali impurità.
Precauzione: utilizzare guanti di protezione poichè l’acrilammide è neurotossica e allo stato liquido può essere assorbita per via cutanea.
Assicurarsi che durante la polimerizzazione il coperchio del Reproset sia abbassato (no esposizione a raggi UV)
4) I campioni amplificati e da caricare per la corsa elettroforetica sono denaturati per 5 min a 95°C (nel termociclizzatore) con una mix di denaturazione nel
rapporto 1: 4 (DNA:mix )
5) Prima di caricare i campioni e dar inizio alla corsa elettroforetica occorre preparare il buffer di corsa TBE 1X (da uno stock TBE10X) che va versato nella
vaschetta superiore ed inferiore..
Precauzione: utilizzare guanti di protezione perché il TBE è irritante.
La corsa elettroforetica consiste nelle seguenti fasi:
1. Plate check: la macchina si configura in funzione del gel;
2. Pre-run:
3. Run: la fase di corsa elettroforetica vera e propria
Precauzione: occorre controllare il sistema di raffreddamento dello strumento
7) Periodicamente i vetri del sequenziatore sono lavati usando 1) una soluzione di HCl 0.5M per 30 min e poi 2) una soluzione di NaOH 0.5 M.
Precauzione: usare guanti, perché entrambe le soluzioni sono corrosive, lavorare sotto cappa ed usare una mascherina nel caso dell’acido cloridrico perché
irritante per le mucose aeree.
55
SOLUZIONI STOCK PER SEQUENZIAMENTO FRAMMENTI DI DNA
SOLUZIONE
METODO DI PREPARAZIONE
Reprogel 377
Amersham
Biotech
Soluzione A+ Soluzione B
Pharmacia (1:1)
COMMENTO E PRECAUZIONE
Soluz. A
(Acrilammide +bis 19:1)
Neurotossico: usare guanti
Soluz B
TBE 1X +Agente denaturante+iniziatore di UV
(brevetto no US 5873991).
LB 6X +formammide 100% + GS 400 HD o 500XL ROX size LB (0.1% blubromofenolo, 10 g Ficoll400 8mL
Mix denaturante
0.5EDTA, 2mL SDS20%, 30 mL H2O)
(tuuti i composti sono standard
comprati
già
pronti
formammide 100%
dall’Applera Italia)
Rox standard
TBE 10X
TBE 1X
Usare guanti: irritante
Ambion
HCl 0.5 M
Acqua distillata:HCl 37%
Usare guanti e maschera: irritante e corrosivo
1:1
NaOH 0.5M
20g NaOH x 1L soluzione
Usare guanti e maschera: irritante e corrosivo
56
PARTE II
METODI ELETTROFORETICI E IMMUNO
CHIMICI
57
METODI ELETTROFORETICI, BIOCHIMICI ED IMMUNO CHIMICI PER IL
RICONOSCIMENTO DI SFARINATI DI FRUMENTO TENERO IN SEMOLE E
PASTA DI SEMOLA DI FRUMENTO DURO
Premessa
Il frumento è una graminacea del genere Triticum, di cui sono note numerose specie.
Le più comuni e le più importanti dal punto di vista merceologico sono il Triticum
aestivum o vulgare, comunemente detto grano tenero, e il Triticum durum o grano duro.
Il grano tenero si presta a essere coltivato a latitudini e climi molto diversi, caldi
temperati e freddi e dalla sua macinazione si ottiene la “farina di grano tenero” o
“farina” destinata alla produzione del pane e dei dolci.
Il grano duro invece può essere coltivato soltanto in zone a clima caldo; a causa di
questa limitazione il suo costo è più elevato rispetto a quello del grano tenero. Dalla
macinazione del frumento duro si ottiene la farina di grano duro comunemente nota
come semola che è particolarmente adatta alla produzione delle paste alimentari.
Ogni qualvolta si parla di pasta di semola di grano duro si associa istintivamente
l’immagine di questo prodotto all’Italia, dove viene ancora preparata secondo la
tradizionale ricetta che prevede l’impiego esclusivo di semola.
Secondo la legge italiana (DPR n 187 del 2001) sono denominati “pasta di semola di
grano duro” e “pasta di semolato di grano duro”, i prodotti ottenuti dalla trafilazione,
laminazione e conseguente essiccamento di impasti preparati rispettivamente ed
esclusivamente con semola di grano duro.
Il prezzo più basso del grano tenero rispetto a quello del grano duro può indurre i
produttori di pasta all’impiego di miscele nella sua fabbricazione e a riportare
fraudolentemente in etichetta la dizione: “pasta di semola di grano duro”. Di qui
nasce la necessità di andare a rilevare aggiunte fraudolente di farina alla semola a
monte del processo di pastificazione sugli sfarinati di semola tal quali e di verificare
che ciò che viene dichiarato sull’etichetta del prodotto finito sia corrispondente al
vero, conducendo delle analisi mirate sulle paste. In tale ottica, sono stati sviluppati, a
partire dalla metà del secolo scorso, numerosi metodi che mirano a stabilire, da una
parte, la purezza degli sfarinati di semola di grano duro e, dall’altra, la miscelazione
di grano tenero con il grano duro. Negli ultimi anni, inoltre, è diventata sempre più
crescente l’esigenza di attuare i controlli sul prodotto finito (non essendo sempre
58
possibile il controllo a monte) che essendo sottoposto a cicli termici di essiccazione
industriali, ha introdotto delle ulteriori difficoltà analitiche. Attualmente non esiste
una normativa uniforme per la regolamentazione del prodotto pastario in ambito
europeo; infatti, mentre la legislazione italiana (art. 2 del DPR n 187 del 2001)
stabilisce per gli sfarinati e semole di grano una tolleranza della presenza di farina di
grano tenero in misura non superiore al 3 % rilevabile con il metodo elettroforetico di
Resmini-De Bernardi, l’UE fissa tale limite al 7%, rilevabile con il metodo immunochimico di Cantagalli Piazzi.
Sono stati sviluppati diversi metodi per l’individuazione di sfarinati di frumento
tener nel duro e alcuni di questi hanno preso in considerazione la frazione proteica. Il
frumento duro e tenero, pur appartenendo allo stesso genere Triticum presentano un
numero diverso di cromosomi rispettivamente 28 e 42, inoltre nel grano tenero è
presente il genoma D assente invece nel frumento duro.
Essendo le proteine composti ad elevato contenuto di informazioni genetiche, lo
studio della frazione proteica fu intrapreso allo scopo di stabilire se il grano tenero
contenesse qualche componente proteica specifica.
Di seguito sono riportati alcuni metodi utilizzati per il riconoscimento degli sfarinati
di frumento tenero in sfarinati e paste di semola di frumento duro.
Metodo elettroforetico di Resmini
Nel 1968 Resmini pubblicò un metodo, divenuto Metodo Ufficiale, per il
riconoscimento di sfarinati di tenero nelle semole, basato sulla separazione della
frazione albuminica mediante precipitazione con solfato di ammonio e successiva
analisi elettroforetica su gel di poliacrilammide. L’elettroforesi evidenziò la presenza
di bande corrispondenti a due proteine specifiche del grano tenero, aventi mobilità
elettroforetica inferiore rispetto alle proteine comuni alle due specie.
Il procedimento proposto da Resmini nella versione originale del 1968 consisteva in
un’estrazione delle albumine precipitabili nell’intervallo di concentrazione do 0,8-1,2
M di solfato di ammonio ed in una purificazione attraverso successive precipitazioni
e ridissoluzioni. Il precipitato ridisciolto in tampone di glicina al 40% di saccarosio;
viene
sottoposto
a
migrazione
elettroforetica,
mediante
una
colonna
di
poliacrilammide. Al termine dell’analisi elettroforetica, si evidenziano le bande delle
59
varie albumine separate, con un rivelatore a base di sale di Schwarz. La Figura 1
riporta il tracciato elettroforetico ottenuto con questo metodo.
b
a
Figura 1. Tracciato elettroforetico delle albumine del frumento duro (a) e del frumento tenero
(b)
Come si osserva per il grano duro si evidenziano quattro componenti proteiche
indicate con i numeri da 1 a 4, senza la comparsa di nessuna altra banda compresa tra
la zona di start e la banda 1. Il tracciato del frumento tenero evidenzia la presenza di
due bande indicate con A e B corrispondenti a due proteine specifiche caratterizzate
da una velocità elettroforetica inferiore alla banda 1, sono evidenti inoltre altre 4
bande indicate con i numeri da 1 a 4 che presentano velocità elettroforetica simile a
quanto riscontrato per le frazioni del frumento duro.
Sperimentazioni
effettuate
su
larga
scala
confermarono
la
costanza
del
comportamento elettroforetico descritto, sia per i frumenti puri che per miscele di
sfarinati. Per quanto concerne la sensibilità, del metodo, l’aggiunta del 10% di
sfarinati di tenero in sfarinati di frumento duro veniva accertata in maniera
inequivocabile. Il metodo indicato, fu reso ufficiale (GU n° 254 del 30 settembre 1974).
Metodo elettroforetico per focalizzazione ionica (Metodo Resmini -De Bernardi)
Il metodo soprariportato presentava difficoltà per la preparazione dell’estratto e per i
tempi di analisi (circa 48 h). Per questo motivo e allo scopo di aumentare la sensibilità
del metodo, nel 1976 Resmini e De Bernardi proposero un nuovo metodo più rapido,
più semplice e più sensibile.Attualmente è questo il metodo che viene ancora attuato
60
per la ricerca del grano tenero. Il metodo di Resmini-De Bernardi si basa sulla
separazione e sul riconoscimento, mediante elettroforesi su strato sottile (gelfocalizzazione ionica in TLC) delle albumine caratteristiche delle due specie di
frumento come già evidenziate con il primo metodo di Resmini. Tuttavia con questo
metodo vengono semplificate le fasi di estrazione e purificazione del campione prima
dell’analisi elettroforetica. Inoltre il maggior potere di risoluzione dell’elettroforesi in
TLC consente di depositare una miscela di proteine non purificate e di ottenere
ugualmente un tracciato con una buona separazione delle bande caratteristiche del
grano tenero e del frumento duro distanziate e valutabili quantitativamente.
Con questo metodo estraendo opportunamente le albumine dal frumento o sfarinati e
sottoponendo a separazione elettroforetica per la focalizzazione ionica su strato
sottile, si ottengono tracciati diversi a seconda che l’estratto derivi dal Triticum durum
o dal T. aestivum. Il differente comportamento elettroforetico permette il
riconoscimento delle due specie di cereali sia separatamente che in loro eventuali
miscele. In particolare dopo separazione delle componenti proteiche, si pongono su
un gel di poliacrilammide dei rettangolini di carta utilizzati per far adsorbire
l’estratto (Fig. 2).
Figura 2. Schema di sistemazione sulla piastra di poliacrilammide dei rettangoli di deposizione
e dei rettangoli di deposizione e delle strisce agli elettrodi
61
Agli estremi della piastra (zona catodica e zona anionica) vengono sistemate due
strisce di carta leggermente imbibite di acido fosforico 1M per la zona anionica e con
una soluzione di NaOH 1M per la zona catodica. Sulle strisce di carta vengono posti
gli elettrodi ed applicato un potenziale costante. In queste condizioni si verifica la
migrazione delle frazioni proteiche che andranno a posizionarsi sulla piastra in
corrispondenza al loro pH isoelettrico; per la rilevazione delle bande si immerge la
piastra in una soluzione di acido tricloroacetico al 12%
Per la valutazione quanti/qualitativa dei risultati è indispensabile condurre in
parallelo ai campioni e sulla stessa piastra delle corse elettroforetiche di campioni di
riferimento standard. In generale si utilizzano miscele di sfarinati a contenuto noto e
crescente di frumento tenero (0, 2, 7 e 15%).
Nella Figura 3 sono riportati i tracciati relativi a miscele standard di frumento tenero
e duro. La parte del tracciato presa in considerazione è quella compresa fra la zona di
deposizione (rettangoli di carta) e la frazione 1 caratteristica del frumento duro. In
questa zona compaiono le frazioni A e B caratteristiche del frumento tenero, assenti
nel tracciato relativo ai prodotti di solo frumento duro. La presenza nel tracciato di
tali bande è pertanto indice della presenza di Triticum aestivum; per piccole
percentuali di frumento tenero compare solo la banda B mentre per percentuali
maggiori campare anche la banda A.
62
Figura 3. Tracciati elettroforetici di miscele di sfarinati di frumento duro e tenero
Dal confronto dei tracciati con quelli ottenuti secondo il metodo originario di Resmini
si può rilevare che il nuovo metodo presenta una elevata sensibilità, poiché è in grado
di evidenziare fino all’1% di grano tenero, inoltre si ha un aumento del potere di
risoluzione in quanto si ha la perfetta separazione ed evidenziazione delle bande
delle due specie di frumento, sia con basse percentuali di grano tenero (1-3%) che con
quelle elevate.
La determinazione quantitativa del frumento tenero viene effettuata dal rapporto
delle intensità delle frazioni A e B con quella della frazione 1 mediante scansione
densitometrica.
Questa tecnica può essere adottata seguendo due criteri, e cioè o quello a scansione
densitometrica del gel così come ottenuto direttamente in trasmissione, o quello a
scansione densitometrica del negativo fotografico del gel, in riflessione.
Nella Figura 4 sono riportati alcuni tracciati ottenuti mediante scansione
densitometrica effettuata in trasmissione.
63
Figura 4. Tracciato elettroforetico ottenuti mediante scansione densitometrica in trasmissione:
(a) campione di frumento duro grano duro, (b) frumento duro con l’1% di frumento tenero, (c)
frumento duro con il 7% di frumento tenero
Nel caso della semola si nota la totale assenza di frazioni proteiche nella zona
anteriore al picco 1. In presenza dell’1% di sfarinati di frumento tenero si evidenzia il
picco B di cui è possibile il dosaggio quantitativo. Con il 3% di tenero, il picco B, a
parità di altezza del picco 1, ha un’altezza otto volte superiore a quella rilevata nel
caso dell’1%. Con il 7% di tenero, il pico B si avvicina per altezza al picco 1.
Dopo aver calcolato le aree dei picchi di interesse, vengono utilizzate le curve di
taratura per il dosaggio quantitativo (Figura 5). In particolare le curve sono costruite
riportando in ascisse la percentuale di grano tenero e in ordinata il rapporto:
area del picco A + area del picco B
area del picco 1
64
Figura 5. Rette di taratura per il dosaggio del grano tenero (dallo 0% al 30%)
Metodo immunochimico di Cantagalli-Piazzi
Questo metodo immunochimico per la ricerca degli sfarinati di grano tenero nelle
semole e nella pasta alimentare, sfrutta la specificità del meccanismo per cui un
antigene viene riconosciuto da uno specifico anticorpo. L’antigene infatti per
definizione è una qualsiasi sostanza estranea che indotta in un organismo determina
una risposta immunologia specifica con formazione di anticorpi capaci di combinarsi
con l’antigene stesso (reazione di immunoprecipitazione).
Il metodo di immunodiffusione è fondato sulla reazione di immunoprecipitazione in
gel di agarosio che si verifica quando la frazione proteica specifica del frumento
tenero, contenuta nello sfarinato estratto, viene messa a contatto con un siero
specifico anti-frumento tenero. Tale frazione proteica deriva dal genoma AABBDD
caratteristico del T. aestivum.
Gli estratti ottenuti da sfarinati di frumento duro non hanno alcuna reazione di
immunoprecipitazione con il siero anti-frumento tenero.
L’analisi
immunologia
può
essere
condotta
con
il
metodo
della
doppia
immunodiffusione o mediante immunodiffusione radiale. Seguendo il metodo della
doppia immunodiffusione, su un vetrino, dopo deposizione del gel di agarosio,
65
vengono effettuati appositi fori di diametro di 3 e 8 mm così come riportato in Figura
6.
Figura 6. Disposizione dei pozzetti nel metodo della doppia immunodiffusione
Nei due pozzetti centrali si dispone il siero specifico anti-frumento tenero, mentre
negli otto pozzetti piccoli si depositano gli estratti proteici ottenuti dal campione di
sfarinato in esame. In presenza di sfarinati di frumento tenero nei campioni in esame,
nel punto di incontro delle due soluzioni si forma una sottile linea di precipitato
costituita dal complesso antigene-anticorpo, osservabili mediante luce incidente su
fondo scuro. Nel caso di soli sfarinati di frumento duro non si osserva alcuna linea di
precipitazione (Fig. 7).
Figura 7. Linee di precipitazione con il metodo della doppia
immunodiffusione
Ac= siero specifico anti-frumento tenero;
a,b,c,d: campioni di sfarinati standard contenenti rispettivamente il 40, 20, 10 e 5% di frumento tenero;
e,f,g campioni contenenti circa il 15, 30 e 10% di farina di tenero;
h: campione di frumento duro
Con il metodo dell’immunodiffusione radiale che è il sistema più utilizzato, è
possibile ottenere dei dati quantitativi abbastanza precisi. Questa tecnica basata sulla
diffusione dell’antigene attraverso un gel nel quale è stato incorporato l’anticorpo
(siero anti-frumento tenero). In particolare sui vetrini viene depositato uno strato di
gel di agarosio contenente il siero anti-frumento. Si ricavano dei pozzetti del diametro
di 3 mm così come riportato in Figura 8.
66
Figura 8. Disposizione dei pozzetti nello strato di siero-agarosio gelificato con il metodo
dell’immunodiffusione radiale
I pozzetti vengono riempiti con estratti proteici di sfarinati e di estratti di miscele
standard. I vetrini vengono successivamente posti in una camera umida per 24 h per
consentire alle soluzioni di diffondere nello stato di agarosio. Gli estratti ottenuti da
sfarinati di frumento duro non danno nessuna reazione di immunoprecipitazione con
il siero anti frumento tenero. In presenza anche di piccole quantità di frumento tenero
si ottiene intorno al pozzetto di deposizione dell’estratto del campione in esame, un
cerchio di precipitazione il cui quadrato del diametro è direttamente proporzionale,
in un cero intervallo, alla quantità di frumento tenero presente (Fig. 9).
Figura 9. Pozzetti di immunodiffusione radiale di campioni di paste alimentari standard a
concentrazioni note di frumento tenero (A 0%, B 2%, C 7%, D 15%, E, F paste commerciali)
Per l’interpretazione quantitativa è necessario costruire una curva ottenuta
riportando su un sistema di coordinate cartesiane in ascisse il quadrato dei diametri
dei cerchi di precipitazione ed in ordinate le percentuali di frumento tenero (Fig. 10).
La percentuale di frumento tenero nei campioni incogniti viene determinata
misurando i diametri dei rispettivi cerchi di precipitazione e facendo riferimento alla
curva di calibrazione, ottenuta con i campioni standard analizzati nel medesimo
vetrino con i campioni incogniti. Poiché per percentuali di frumento tenero superiori
al 20% non vi è relazione lineare tra il quadrato del diametro dei cerchi di
67
precipitazione
e
la
percentuali
di
frumento
tenero,
è
necessario
diluire
Frumento tenero %
opportunamente il campione.
Figura 10. Curva di calibrazione ottenuta con sfarinati a concentrazione nota di frumento
tenero
Metodo kit immunochimico Durotest
Un altro metodo disponibile per la valutazione del frumento tenero in sfarinati di
duro è il DUROTEST che utilizza un kit monouso. Questo test sfrutta un anticorpo
monoclonale che è specifico esclusivamente per la proteina friabilina, presente solo
nel frumento tenero. In presenza di frumento tenero in sfarinati di duro si forma un
complesso tra un anticorpo e la proteina che in presenza di un reagente cromoforo,
assume una colorazione blu. Il grado di colorazione del campione è proporzionale
alla percentuale di frumento tenero, la quale è stimata per confronto con uno
standard di frumento tenero, fornendo così un dato semi-quantitativo, utile più per
avere un’indicazione sommaria sulla presenza o meno di sfarinati di tenero più che
per una determinazione della sua quantità. Mentre il metodo di Resmini è
sufficientemente accurato, ma richiede apparecchiature specifiche ed un elevato
68
livello di conoscenza del metodo, il Durotest è un sistema abbastanza rapido (2 ore)
ma le determinazioni sono soprattutto di carattere qualitativo.
La Figura 11 riporta in maniera schematica la procedura di utilizzo del kit Durotest.
Figura 11. Procedura di utilizzo del kit Durotest
Analisi della frazione sterolica
I metodi di analisi disponibili in letteratura e attualmente utilizzati per evidenziare
l’aggiunta di frumento tenero nelle semole sono fondati sull’analisi di componenti di
natura proteica, tuttavia tali metodiche non trovano una facile applicazione a causa
della denaturazione di tali composti in seguito ai trattamenti termici cui vengono
oggigiorno sottoposte le paste. L’ attenzione dei ricercatori è stata perciò rivolta
all’analisi della componente di natura lipidica (trigliceridi, steroli ed esteri degli
69
steroli) degli sfarinati. In particolare lo studio della frazione sterolica dei cereali può
fornire delle utili indicazioni sulla presenza di sfarinati di frumento tenero nelle
semole. Gli steroli, componenti minori della frazione lipidica delle sostanze grasse,
sono spesso utilizzati in campo analitico per identificare l’origine botanica della
sostanza grassa, essendo composti specie specifici.
Già negli anni ‘60 alcuni studi evidenziarono l’importanza della valutazione del
sitosteril palmitato per discriminazione il T. aestivum dal T. durum (Gilles e Youngs
1964, Sarwar e McDonald 1993). Studi recenti condotti da Caboni et al., 2005 hanno
dimostrato l’utile applicazione della valutazione degli esteri degli steroli per la
differenziazione dei frumenti. Le analisi condotte mediante tecniche cromatografiche
combinate e altamente efficienti (GC, GC-MS, HPLC, HPLC-MS) hanno evidenziato
la presenza di quantitativi differenti di steroli esterificati con l’acido palmitico nel
frumento tenero e nel frumento duro.
70
Bibliografia
Caboni MF, Iafelice G, Pelillo M, Marconi E. Analysis of fatty steryl esters in
tetraploid and hexaploid wheats: identification and comparison
between chromatographic methods (2005). J. Agric. Food Chem. 53:
7465-7472.
Cantagalli P, Piazzi SE, Sordi-Galli S. (1969). Il controllo della genuinità delle semole
di frumento duro e delle paste alimentari mediante immunologia.
Tecnica Molitoria 4: 79-85.
DPR n 187 del 9 febbraio 2001. Regolamento per la revisione della normativa sulla
produzione e commercializzazione di sfarinati e paste alimentari.
Gilles KA, Youngs VL. Evaluation of durum wheat and durum products. Separation
and identification of sitosterol esters of semolina (1964). Cereal Chem.
41: 502-513.
GU n° 4 del 5 gennaio 1980 Metodi ufficiali di analisi dei cereali.
Resmini P, De Bernardi G. Possibilità di semplificare il metodo elettroforetico Resmini
per la ricerca ed il dosaggio dei prodotti del grano tenero in quelli di
grano duro (1969). Tecnica Molitoria 4: 70-75.
Resmini P, De Bernardi G. Il metodo elettroforetico rapido per il riconoscimento ed il
dosaggio del grano tenero nel grano duro, negli sfarinati e nelle paste
alimentari (1976). Tecnica Molitoria 10: 97-101.
Sarwar M, McDonald CE. Detection of bread wheat farina adulteration in durum
wheat semolina and pasta dried at low, high and ultra high
temperatures (1993). Cereal Chem. 70: 405-411.
71
ALLEGATO 1
PROTOCOLLO PER IL RICONOSCIMENTO E DOSAGGIO DEGLI SFARINATI DI FRUMENTO
MEDIATE FOCALIZZAZIONE IONICA
(Metodo Resmini-De Bernardi, GU n. 4 del 5 gennaio 1980)
Estrazione della proteina specifica
1) Preparare la soluzione di estrazione mescolando 200 ml di una soluzione di solfato di magnesio
0,13M con 73 ml della soluzione di solfato di ammonio 3M modulando il pH finale fino a 4,6.
2) Pesare lo sfarinato (5g), aggiungere 20 ml della soluzione estraente in una beuta e disperdere
uniformemente. In queste condizioni, la miscela di prodotto macinato e la soluzione salina deve
avere un pH compreso tra 4,5 e 4,9. La fase di estrazione deve durare almeno 30 min.
3) Trasferire la miscela (sfarinato e soluzione salina) in provette da 50 ml e centrifugare.
4) Trasferire 10 ml di surnatante in un becker da 25 ml e aggiungere, sotto agitazione, 2,25 ml della
soluzione di solfato di ammonio
5) Lasciare riposare a temperatura ambiente per almeno 15 min.
6) Trasferire in provette da 15 ml e centrifugare.
7) Il precipitato ottenuto è quindi pronto per la separazione elettroforetica
Separazione elettroforetica in focalizzazione ionica
1) Attivare il sistema refrigerante dell’apparecchio elettroforetico impostando una temperatura di
10°C.
2) Cospargere di vaselina la piastra refrigerante dell’apparecchiatura e sistemare su di essa il gel di
poliacrilammide evitando la formazione di bolle d’aria.
3) Solubilizzare il precipitato in 25 µl di soluzione di urea 3M
4) Porre sul gel di acrilammide dei rettangolini di carta Wathman a 2 cm dal bordo catodico della
piastra.
5) Depositare e far assorbire circa 10 µl il precipitato proteico ottenuto come indicato
precedentemente.
6) Agli estremi della piastra (zona catodica e zona anodica) sistemare due strisce di carta, di
dimensioni 25x0,5 cm e 0,2 mm di spessore, leggermente imbibite con una soluzione di acido
fosforico per la zona anodica e con una soluzione di NaOH 1M per la zona catodica.
7) Sistemare gli elettrodi della cella in corrispondenza delle strisce di carta.. Applicare una corrente di
0,75 watt per ogni cm di fronte del gel . In queste condizioni la corsa elettroforetica e la
stabilizzazione delle proteine al loro pH isoelettrico è ultimata in circa 105 min.
Rivelazione delle frazioni proteiche
Asportare con una lametta il gel di poliacrilammide, immergere il gel con la parte superiore (superfice
di deposizione) verso l’alto in una vaschetta contenente una soluzione di acido tricloro acetico. Dopo 25 minuti vengono evidenziate le frazioni proteiche che appaiono bianche nel gel rimasto trasparente ed
incolore.
Interpretazione qualitativa e quantitativa dei risultati
Per la valutazione dei risultati è indispensabile condurre parallelamente ai campioni incogniti e sullo
stesso supporto determinazioni di campioni di riferimento standard. Utilizzare nel caso di analisi di
sfarinati, miscele di sfarinati a contenuto noto e crescente di frumento tenero 0, 2, 7 e 15%
La valutazione quantitativa dei risultati viene condotta confrontando il tracciato del campione
incognito con quelli dei campioni standard evidenziati sullo stesso gel.
72
ALLEGATO 2
PROTOCOLLO PER IL RICONOSCIMENTO E DOSAGGIO DEGLI SFARINATI DI FRUMENTO
MEDIATE METODO IMMUNOCHIMICO
(Metodo Cantagalli-Piazzi,G.U. n. 4 del 5 gennaio 1980)
Estrazioni della proteina specifica
1) Miscelare accuratamente 4 g di pasta con 5 ml di soluzione di cloruro di sodio 0,15M all’1°/°° si
sodio azide. Per gli sfarinati miscelare 2 g di prodotto con 4 ml della soluzione di cloruro di sodio.
2) Dopo riposo da 6 a 15 ore a temperatura ambiene centrifugare la sospesione per 15 min a 4000xg.
3) Recuperae il surnatante ed effettuare l’analisi di immunodiffusione.
Preparazione dei vetrini per l’analisi
1) Depositare sui vetrini di vetro, accuratamente puliti, un sottile strato di agarosio preventivamente
fluidificato a caldo.
2) Essiccare i vetrini in stufa a 80°C quindi lasciare raffreddare a temperatura ambiente.
3) Aggiungere a 1 ml di siero specifico anti-frumento tenero, 2,2 ml di agarosio tamponato Versare
rapidamente questa miscela sul vetrino in modo che si distribuisca uniformemente.
4) Lasciare raffreddare lentamente a temperatura ambiente i vetrini così stratificati in modo da
ottenere la gelificazione dello strato siero-agarosio.
5) Incidere con un foratappi lo strato di siero-agarosio gelificato in modo da avere 8 pozzetti.
6) Asportare l’agarosio inciso in modo da avere la formazione di pozzetti nello strato di agarosio.
Analisi di immunodiffusione radiale
1) Utilizzare gli 8 pozzetti per l’analisi in doppio di due campioni di pasta o sfarinato e in singolo,
dei quattro standard di confronto.
2) Deporre 10 µl degli estratti proteici nel corrispondente pozzetto. Trasferire i vetrini in camera
umida e lasciare almeno 24 ore per consentire alle soluzioni di diffondere nello strato di agarosio.
3) Sottoporre i vetrini ad una serie di lavaggi con una soluzione di cloruro di sodio, cambiando la
soluzione di lavaggio 3 volte al giorno.
4) Procedere al fissaggio dei precipitati immergendo per 30 min i vetrini in una soluzione di acido
tannico. Trasferire i vetrini in acqua distillata per 2-3 ore ed essiccarli in stufa a 37°C.
5) Decolorare i preparati immergendoli nella soluzione decolorante e seccare i vetrini a temperatura
ambiente.
Interpretazione qualitativa e quantitativa dei risultati
Per la valutazione quantitativa dei risultati è indispensabile condurre parallelamente ai campioni
incogniti e sullo stesso vetrino l’analisi di campioni di riferimento standard.
La comparsa attorno ai pozzetti di deposizione, di un anello di precipitazione è indice della presenza
nel campione analizzato, di sfarinati di frumento tenero.
Per l’interpretazione quantitativa è necessario tracciare una retta di taratura che correli il quadrato del
diametro dei cerchi di precipitazione con la percentuale di frumento tenero.
1) Misurare con uno strumento in grado di apprezzare 1/10 di mm il diametro dei cerchi di
precipitazione ottenuti con i campioni standard.
2) Costruire su un sistema di coordinate cartesiane, dove in ascisse sono posti i quadrati dei diametri
ed in ordinate le percentuali di frumento tenero, una retta di taratura.
3) Determinare la percentuale di frumento tenero nei campioni incogniti misurando i diametri dei
rispettivi cerchi di precipitazione e facendo riferimento alla retta di taratura ottenuta con in
campioni standard utilizzati sul medesimo vetrino con i campioni incogniti.
Poiché per percentuali di frumento tenero superiori al 20% non vi è relazione lineare tra il quadrato del
diametro del cerchio di precipitazione e la percentuale di frumento tenero, per campioni che abbiano
rivelato un contenuto di frumento tenero superiore al 20% sarà necessario diluire opportunamente
l’estratto con la soluzione di cloruro di sodio e sottoporlo nuovamente all’analisi di immunodiffusione
radiale.
73












