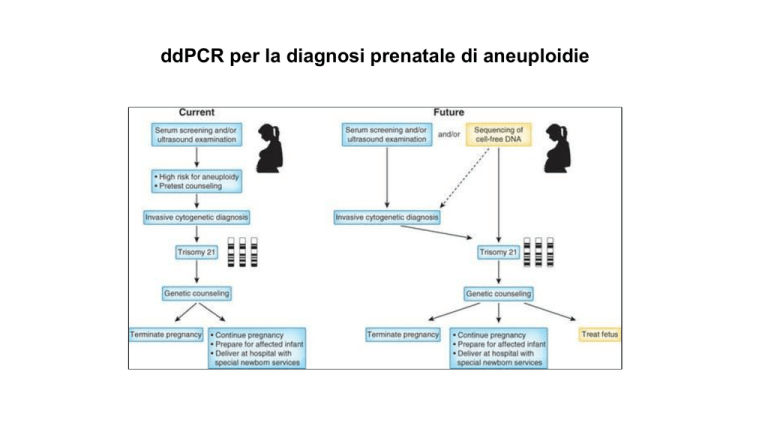
ddPCR per la diagnosi prenatale di aneuploidie
Rilevazione della presenza di trisomia 21 per la diagnosi prenatale
Il sangue materno
contiene circa il
10% di DNA
fetale
Il grado di sovra-espressione dipende
dalla concentrazione di DNA fetale
frazionata nel cfDNA (DNA fetale
circolante,
delle cellule fetali). Ad
esempio, la percentuale teorica di chr21
nel genoma del feto con trisomia 21 (T21)
dovrebbe essere 0,6. Quando si analizza
un campione di plasma materno
contenente almeno 3% DNA fetale.
Per valutare la presenza di altre
aneuploidie fetali relative ai cromosomi
18, 13 ed ai cromosomi sessuali (X e Y).
50 (chr21 positive)
Ratio=
= 0,5
100 (50 chr1 + 50 chr21 positive)
75 (chr21 positive)
= 0,6
Ratio=
125 (50 chr1 + 75 chr21 positive)
Limiti della metodica: uno è rappresentato dalla presenza di mosaicismi a bassa percentuale e dalla possibilità che la quantità di DNA fetale circolante
non sia sufficiente ad ottenere un risultato
Tipizzazione allelica di varianti genetiche e mutazioni geniche rare:
il gene CFTR (Cystic fibrosis transmembrane conductance regulator)
CFTR
Applicazioni della droplet digital PCR
Quantificazione assoluta: studi di biomarcatori nel cancro e variazione del numero di copie (droplet
digitale PCR su fluidi biologici e biopsie)
Individuazione di mutazioni rare: misura vari gradi di mutazioni tumorali, rilevare bersagli di DNA rari,
evidenzia le variazioni del numero di copie con sensibilità e risoluzione superiore ad altre tecniche.
Presenza di varianti genetiche per malattie rare ereditarie come CFTR, beta-talassemia, etc…
(haplotyping)
Le analisi di linkage
Analisi di espressione genica: per ottenere misure affidabili e riproducibili su mRNA poco abbondanti
e miRNA.
Rilevamento di agenti infettanti poco rappresentati, diluiti: per quantificare piccole variazioni di
molecole di DNA o RNA per il rilevamento e monitoraggio di agenti patogeni (genoma virale nel DNA
umano)
Next Generation Sequencing: eseguire il controllo della qualità e quantificazione assoluta di librerie
NGS senza l'uso di una curva standard
La quantificazione ultrasensibile di Eventi di Genome Editing
Analisi su singola cellula
Monitoraggio ambientale: testare un'ampia varietà di campioni ambientali come suolo e acqua
Rilevamento di allergeni negli alimenti: analisi degli alimenti
Effettuare la valutazione di routine per la presenza di organismi geneticamente modificati (OGM)
Tecniche “omiche”
Per tecnologie “omiche” si intendono quelle applicazioni biotecnologiche che hanno un potere
d’indagine molecolare esteso all’intero potenziale codificante della cellula
•
•
•
la genomica, che studia tutti
i geni di un organismo (NGS;
SNP-array)
la trascrittomica, si occupa
dell'espressione dei geni
negli mRNA di un intero
organismo
o
di
un
particolare organo, tessuto o
cellula, in momento dello
sviluppo dell'organismo o in
particolari
condizioni
ambientali
(microarrays:
gene expression array; miRprofiling)
•
•
la proteomica, si occupa dell'insieme di tutte
le proteine di un organismo, tessuto o cellula,
con l'obbiettivo di determinarne la sequenza
aminoacidica, la funzione, la struttura
tridimensionale e le sue interazioni.
la metabolomica, è una branca della
biochimica che si occupa del metabolismo,
individuando ad esempio la quantità di
diversi metaboliti con raffinate tecniche
biochimiche quali la gascromatografia,
nonché specifici saggi di attività enzimatica.
Sul microchip possono essere immobilizzati:
anticorpi, proteine purificate, lisati cellulari
la biologia sistemica, si pone quindi come
obiettivo lo studio delle interazioni tra le
molecole
di
un
intero
organismo,
considerandolo nella sua totalità, a differenza
della tradizionale biologia molecolare che
parte dallo studio di singole interazioni.
Questa scienza interseca idealmente i dati
di genomica, trascrittomica, proteomica e
metabolomica.
Tecniche “omiche” per lo studio di malattie genetiche:
microarray CGH (Comparative Genomic Hybridization)
Il principio su cui si basa la tecnica dell’array CGH è la comparazione quantitativa del DNA in esame
estratto dalle cellule fetali (diagnosi prenatale) o dal prelievo ematico del soggetto (diagnosi post-natale)
e del DNA genomico di riferimento proveniente da un soggetto sano (reference DNA).
L’ibridazione genomica comparativa su microarray CGH è una
tecnica sviluppata per identificare anomalie cromosomiche di
tipo numerico (aneuploidie) a carico dei 22 autosomi
(cromosomi dal nr. 1 al nr. 22) e dei cromosomi sessuali (X e Y),
o anche variazioni del numero di copie (CNV) del contenuto di
piccole porzioni cromosomiche, come duplicazioni/amplificazioni
(presenza di copie in eccesso di segmenti di DNA), o delezioni
(perdite di porzioni di genoma). Queste anomalie del DNA
possono essere la causa di diverse patologie quali, ad esempio,
sindromi malformative, ritardo mentale, autismo, epilessia e
tumori.
Microarray CGH (Comparative Genomic Hybridization)
DNA
sano
Gene A
DNA
malato
Il DNA da analizzare e il DNA
reference sono marcati con
differenti molecole fluorescenti,
vengono mescolati in parti uguali
e fatti incubare (ibridazione) su
un microarray, costituito da un
supporto di vetro la cui superficie
è coperta di frammenti di DNA,
costituiti da oligonucleotidi o
sonde di maggiori dimensioni.
Ognuno di queste sonde
rappresenta
una
specifica
regione del genoma umano, fino
a ricomprendere l’intero assetto
cromosomico umano.
Gene B
3’
5’
5’
3’
Tanto più è elevato il numero di sonde
maggiore
è
l'efficacia
dell'array
nell'identificazione delle variazioni del
numero di copie. corrispondenti a piccole
porzione di ciascun cromosoma. Il potere
risolutivo della piattaforma utilizzata può
variare in funzione della densità e della
tipologia delle sonde utilizzate; attualmente
per scopi diagnostici vengono impiegati
array tra 1 Mb e 100 kb.
Gene A
Gene B
Al termine della suddetta
incubazione, sia il DNA in
esame che quello di controllo
si legheranno alle sonde
presenti sull’array.
Microarray GCH (Comparative Genomic Hybridization)
Il risultato sarà l’emissione di due distinti segnali
fluorescenti relativi ai due differenti DNA marcati, le cui
intensità saranno misurante a seguito di lettura degli
arrays mediante un apposito strumento (scanner).
Sull’immagine ottenuta verrà poi effettuata l’analisi
comparativa tra le intensità di fluorescenza emesse dai due
DNA, la relativa elaborazione dei dati mediante un apposito
software evidenzierà eventuali variazioni del numero di
copie del DNA in analisi rispetto al DNA reerence.
In caso di assetto cromosomico normale, il rapporto tra
le due emissioni è bilanciato (1:1). Qualora vi siano nel
DNA in esame (fetale) delle delezioni (assenza di un
cromosoma o parte di esso), il rapporto tra quest’ultimo ed il
DNA di controllo sarà di 1:2 (monosomia completa o
parziale). Nel caso di duplicazioni (presenza di un
cromosoma soprannumerario o parte di esso) il rapporto tra
il DNA embrionale e quello di controllo sarà di 2:1 (trisomia
completa o parziale).
Indicazioni all’uso della tecnica array-CGH
L’array-CGH viene prevalentemente utilizzata per la diagnosi di fenotipi complessi associati a ritardo mentale di grado
variabile.
Questo tecnica anche trova impiego anche come tecnica di routine nella diagnosi prenatale, ed è particolarmente
indicata per pazienti con feto i cui segni ecografici sono riconducibili ad una patologia cromosomica, il cui
cariotipo è però risultato normale. In questi casi, l’array-CGH rappresenta una procedura ideale di approfondimento
diagnostico di secondo livello, eseguita per integrare l’analisi citogenetica prenatale, al fine di definire più
accuratamente eventuali anomalie cromosomiche precedentemente identificate o per rivelare microriarrangiamenti non
evidenziabili con l'indagine del cariotipo fetale. L'integrazione dell'analisi citogenetica convenzionale con l'array-CGH
incrementa notevolmente le possibilità di determinare le cause della patologia riscontrata nel feto ed eventualmente
permette di definire più accuratamente il rischio di ricorrenza.
Le principali indicazioni per l’impiego dell’array-CGH quale tecnica di approfondimento diagnostico in diagnosi prenatale
sono le seguenti: difetti dello sviluppo fetale evidenziati tramite ecografia; anomalie cromosomiche (riarrangiamenti
sbilanciati, riarrangiamenti apparentemente bilanciati de novo e cromosomi marcatori) individuate attraverso l’analisi
citogenetica prenatale.
Particolare importanza riveste questa applicazione pre-natale nell’esame del liquido amniotico o dei villi coriali in quanto
con una sola determinazione è possibile rilevare circa 100 patologie non diagnosticabili diversamente, che
provocano ritardo mentale e patologie correlate nel bambino..
I limiti di tale tecnica in ambito prenatale sono rappresentati dall’impossibilità di identificare riarrangiamenti
cromosomici bilanciati e mosaicismi con una linea cellulare scarsamente rappresentata (inferiore al 10%).
E’ importante sottolineare la necessità che questa tecnica sia utilizzata da laboratori dotati di provata competenza di
genetica molecolare, nonché di esperienza nella interpretazione dei risultati prodotti dalla array-CGH.
I vantaggi della tecnica array-CGH
1
2
3
4
FISH
La citogenetica tradizionale, pur utilissima nell’individuare un gran numero di anomalie cromosomiche, numeriche e strutturali, è necessariamente limitata nelle sue
possibilità diagnostiche dal potere di risoluzione del microscopio.
Il maggiore progresso degli ultimi anni, nel campo della citogenetica, e’ rappresentato dallo sviluppo della tecnica FISH (Fluorescence In Situ Hybridization), una
metodologia citochimica che consente di individuare specifiche sequenze di DNA a livello cromosomico. Il principio su cui si basa è l’appaiamento tra una sonda marcata
(frammento di DNA specifico per la regione d’interesse contenente nucleotidi modificati) ed il DNA cromosomico del soggetto in studio, fissato su un vetrino. Tale tecnica
permette di identificare marcatori cromosomici, di visualizzare traslocazioni bilanciate e non bilanciate, duplicazioni e riarrangiamenti cromosomici.
La FISH, nonostante sia di grande applicazione nella diagnosi clinica odierna, è necessariamente limitata dal fatto di essere una tecnica di "indagine mirata": la sua
applicazione consente solo di poter individuare mutazioni specifiche a livello di precisi loci cromosomici.
Con l’impiego della tecnica array-CGH si è grado di valutare la presenza di eventuali anomalie cromosomiche a livello dell’intero genoma in un unico esperimento,
senza sapere in anticipo cosa cercare. Rispetto ad altre metodiche d’indagine, come l'esame tradizionale del cariotipo, l'analisi del genoma basata su array ha una
risoluzione molto più elevata (100 volte). Ciò consente di evidenziare anomalie del DNA che normalmente non potrebbero essere rilevate, incrementando notevolmente
le possibilità di raggiungere una diagnosi certa. Inoltre, l'array-CGH permette di definire esattamente la regione genomica alterata e quindi anche i geni in essa
contenuti, migliorando la comprensione delle relazioni esistenti tra variazioni del numero di copie e patologia.
Trascrittomica e utilizzo di microarray
Oggi è possibile analizzare l’espressione di
tutti i geni di una cellula, tessuto, etc..
contemporaneamente grazie ai microarray
o biochip.
Si tratta di piccole lastre di vetro o plastica
delle dimensioni di un chip di computer,
su cui sono immobilizzate migliaia di sonde
molecolari ognuna corrispondente ad una
singola sequenza genica, all’interno di
micropozzetti da 50 µm.
Oggi sono disponibili set di microarray che
coprono tutta la porzione codificante del
genoma umano (90 milioni di sonde).
Trascrittomica e utilizzo di microarray
Microarray o biochip:
Il chip ha una superficie di vetro o silicone
piccole sonde di DNA o RNA ancorate alla superficie (10-20 copie,
fino a centinaia di migliaia)
ciascun gene occupa una determinata posizione sul Chip (spot)
il campione in analisi viene amplificato per PCR
il campione da analizzare viene marcato con una molecola
fluorescente
il campione viene caricato sul chip
tutti i geni espressi nel campione in analisi ibridizzano con le
rispettive sonde specifiche sul chip contemporaneamente
la fluorescenza in ciascuno spot del chip, corrispondente a ciascun
gene viene rilevata come segnale ed analizzata da un sistema di
elaborazione dati
Detection
Due
biochip,
uno per l’analisi
del
genoma
umano e uno
per quella del
genoma
del
topo.
Tipi di microarray
1.
cDNA arrays
2. Oligonucleotidi arrays
Trascrittomica e utilizzo di microarray
Un’applicazione dei microarray è l’analisi
dell’espressione differenziale di geni in due
popolazioni cellulari.
1.
l’mRNA è estratto da cellule di controllo (ad
es. cellule di un individuo sano) e dal
campione
sperimentale
(es.
cellule
tumorali).
2.
l’mRNA viene convertito in cDNA e marcato
con sonde fluorescenti di diverso colore
per i controlli (ad es. verde) e i campioni (ad
es. rosso).
Trascrittomica e utilizzo di microarray
3.
i cDNA vengono ibridati al microarray.
L’illuminazione al laser rivelerà segnali rossi
o verdi in corrispondenza dei geni espressi
solo in ciascuna delle due popolazioni, e
gialle (rosso+verde) per quelli espressi in
entrambe.
Trascrittomica e utilizzo di microarray
https://www.youtube.com/watch?v=9U-9mlOzoZ8
Trascrittomica: analisi dei geni espressi in cellule trattate/controllo;
sane/patologiche
Trascrittomica: analisi dei geni espressi in diversi tessuti
Trascrittomica: effetti dell’inquinamento ambientale sull’espressione genica
Trascrittomica: analisi dei geni espressi in cellule neoplastiche di diversi pazienti
RNA
RNA
RNA
RNA
RNA
Geni comunemente espressi
Trascrittomica: analisi dei dati di espressione genica
Heatmap, o comparazione quantitativa:
Il lettore di fluorescenza compara il livello relativo ai due
segnali (rosso-verde)
geni espressi egualmente nei due campioni hanno livelli
uguali delle due fluorescenze e il loro un rapporto sarà
pari a 1
rapporti superiori o inferiori riflettono un’espressione
differenziale nei due campioni
L’operatore definisce un soglia di «differenza», un cut-off,
secondo il quale valori superiori a tale valore definiscono
un’espressione
differenziale
nei
due
campioni
(esempio=2….ma arbitrario)
I geni sono riportati più volte sul chip, quindi più spot per
ogni gene
Oppure ciascun campione almeno in triplicato, per dare
valenza statistica, e determinare se le differenze siano
quindi significative
Utilizzo di analisi statistiche
Trascrittomica: analisi dei dati di espressione genica
Il test t è un test statistico di
tipo parametrico con lo
scopo di verificare se il
valore medio di una
distribuzione si discosta
significativamente da un
certo valore di riferimento.
p = P(|X − E| ≥ |x − E|)
Utilizzo di analisi statistiche (non solo il p value<0,05 o
p<0,001) più complicate come metodo SAM (che considera
anche la frequenza di eventi falsi positivi e falsi negativi)
p value ci dice se i dati
osservati sono statisticamente
significativi. Se ho un p<0.05, la
discrepanza
viene
detta
statisticamente significativa.
Se
0.01≤p<0.05,
molto
significativa.
Se 0.001≤p<0.01 e p<0.001,
estremamente significativa
Trascrittomica: problematiche
Si assume che ciascun passaggio (estrazione RNA, reazione di retrotrascrizione, PCR,
marcatura, ibridazione) abbia la stessa efficienza per ogni gene e per ciascun
campione
La ripetizione del campione elimina variazioni casuali, ma non eventuali errori
sistematici: ad es. gene non amplificato bene per PCR a causa di una competizione
dei primers su altri bersagli
La stessa concentrazione definita per ciascun mRNA bersaglio non indica il livello di
trascrizione di quel gene, ma sarà influenzata dalla velocità di sintesi e di degradazione
del trascritto stesso, dalla sua stabilità (esempio mRNA poco trascritti, ma molto
stabili possono essere presenti a livelli più alti rispetto a trascritti meno stabili)
Dati da validare con altre tecniche, come RT-PCR quantitativa su singoli geni
specifici
Verificare la sua traduzione in proteina: proteomica e mediante studi di Biologia
sistemica
Biologia sistemica
Trascrittomica: limiti
Vengono analizzati i trascritti, ovvero gli mRNAs aventi sequenza nota, dei
quali risulta possibile quindi costruire le sequenze probe
Non possono essere identificate variazioni nella struttura secondaria
nell’mRNA
Non possono essere identificate isoforme di uno stesso mRNA
Non è un metodo di valutazione assoluta, bensì comparativa
Problematiche superate con l’RNA-sequencing
RNA-Sequencing
5´-pre-adenylated
RNA-Sequencing, che è stato introdotto come un nuovo modo per l'analisi di espressione genica, si riferisce al
sequenziamento della miscela di trascritti (RNA) che sono stati inizialmente convertito in cDNA mediante
trascrizione inversa. RNA-Sequencing ha portato a molte applicazioni e alla produzione di nuovi dati: per esempio, trovare
nuovi siti di splicing alternativo, identificando nuove giunzioni esone/introne; studiare la regolazione genica mediante RNA
antisenso; studiare l’espressione intragenica e di ''piccoli RNA'' (microRNA e altri RNA non codificanti).
RNA-Sequencing using ePCR
http://www.geospiza.com/finchtalk/uploaded_imag
es/plates-and-slides-718301.png
Genome transcriptome assembly and quantification
nuovi RNA
Trascritti con
variazioni
di splicing
RNA-Sequencing
*RPKM (reads per kilobase per million mapped reads)
Goals of Transcriptoma sequencing vs. gene expression microarray
Microarrays
RNA sequencing
Metodica affidabile, utilizzata da decenni
Fornisce una visione più completa del trascrittoma
Elevato numero di campioni analizzabili
Indipendente dalla conoscenza della sequenza
Facilmente automatizzabile
sensibilità modulabili
Analisi dei dati lineare
Può rilevare variazioni strutturali, isoforme, fusioni
geniche e variazioni di splicing
Basso costo
Tecnologia veramente digitalizzata, dati assoluti di
espressione genica




![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)