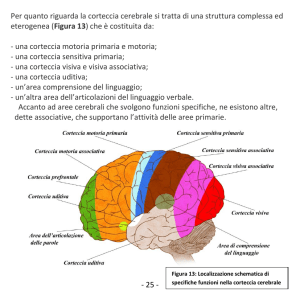
Il ruolo dell’espressione genica nella corteccia cerebrale
nuovi confini della neuroanatomia molecolare
Tra gli enigmi che ancora avvolgono i meccanismi dell’organismo umano, il funzionamento della corteccia cerebrale occupa indubbiamente un posizione di rilievo, già partendo da
considerazioni unicamente di carattere morfologico: la corteccia è una struttura esalaminare1 tutto sommato omogenea; tuttavia può essere divisa in regioni anatomicamente e
funzionalmente distinguibili che hanno impegnato molti neuroanatomici del secolo scorso, le cui classificazioni sono ancora oggi largamente utilizzate (Brodmann) e che hanno
trovato conferme nei più recenti studi di neuroimaging. Nonostante ciò non è ancora del tutto chiaro in che modo le differenze strutturali siano associate alle differenze funzionali.
Certamente uno dei fattori principali è costituito dalle connessioni che mettono in comunicazione una particolare area della corteccia con altre strutture del sistema nervoso
centrale, ma la questione è capire in che grado queste connessioni sono veramente determinanti per le differenze funzionali delle varie aree, ovvero quali altre differenze
fondamentali esistono tra aree funzionalmente distinte oltre ai loro rapporti?
Questi interrogativi hanno spinto diversi studiosi ad esaminare qualitativamente e quantitativamente le
biomolecole presenti nelle diverse aree corticali. Una certa eterogeneità chimica tra regioni differenti era già stata
registrata; bisogna però considerare che un semplice saggio chimico non sarebbe di nessuna utilità nella ricerca
della risposta ai quesiti che ci si è posti dal momento che i risultati potrebbero riflettere quelle che sono le
caratteristiche biochimiche non dell’area in sé, ma ancora una volta delle sue connessioni. Per questo motivo
successivamente si è preferito indagare sulle proprietà intrinseche delle aree attraverso lo studio degli mRNA
prodotti (dal momento che questi ultimi sono solitamente localizzati nel soma dei neuroni) e quindi dei diversi
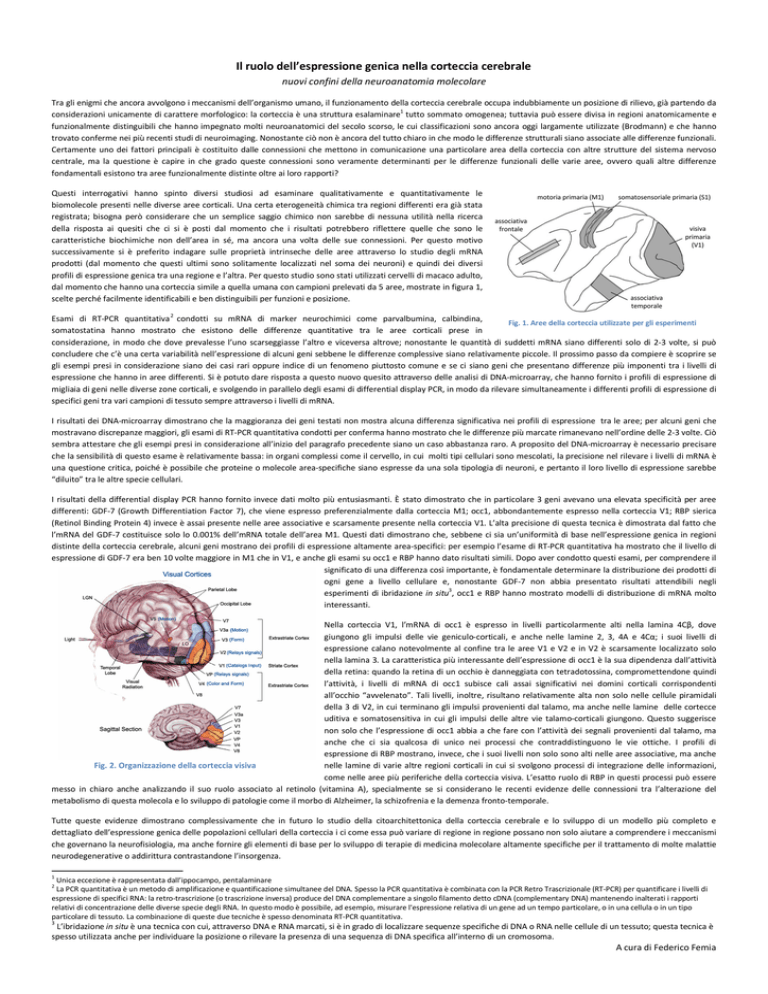
profili di espressione genica tra una regione e l’altra. Per questo studio sono stati utilizzati cervelli di macaco adulto,
dal momento che hanno una corteccia simile a quella umana con campioni prelevati da 5 aree, mostrate in figura 1,
scelte perché facilmente identificabili e ben distinguibili per funzioni e posizione.
Esami di RT-PCR quantitativa 2 condotti su mRNA di marker neurochimici come parvalbumina, calbindina,
Fig. 1. Aree della corteccia utilizzate per gli esperimenti
somatostatina hanno mostrato che esistono delle differenze quantitative tra le aree corticali prese in
considerazione, in modo che dove prevalesse l’uno scarseggiasse l’altro e viceversa altrove; nonostante le quantità di suddetti mRNA siano differenti solo di 2-3 volte, si può
concludere che c’è una certa variabilità nell’espressione di alcuni geni sebbene le differenze complessive siano relativamente piccole. Il prossimo passo da compiere è scoprire se
gli esempi presi in considerazione siano dei casi rari oppure indice di un fenomeno piuttosto comune e se ci siano geni che presentano differenze più imponenti tra i livelli di
espressione che hanno in aree differenti. Si è potuto dare risposta a questo nuovo quesito attraverso delle analisi di DNA-microarray, che hanno fornito i profili di espressione di
migliaia di geni nelle diverse zone corticali, e svolgendo in parallelo degli esami di differential display PCR, in modo da rilevare simultaneamente i differenti profili di espressione di
specifici geni tra vari campioni di tessuto sempre attraverso i livelli di mRNA.
I risultati dei DNA-microarray dimostrano che la maggioranza dei geni testati non mostra alcuna differenza significativa nei profili di espressione tra le aree; per alcuni geni che
mostravano discrepanze maggiori, gli esami di RT-PCR quantitativa condotti per conferma hanno mostrato che le differenze più marcate rimanevano nell’ordine delle 2-3 volte. Ciò
sembra attestare che gli esempi presi in considerazione all’inizio del paragrafo precedente siano un caso abbastanza raro. A proposito del DNA-microarray è necessario precisare
che la sensibilità di questo esame è relativamente bassa: in organi complessi come il cervello, in cui molti tipi cellulari sono mescolati, la precisione nel rilevare i livelli di mRNA è
una questione critica, poiché è possibile che proteine o molecole area-specifiche siano espresse da una sola tipologia di neuroni, e pertanto il loro livello di espressione sarebbe
“diluito” tra le altre specie cellulari.
I risultati della differential display PCR hanno fornito invece dati molto più entusiasmanti. È stato dimostrato che in particolare 3 geni avevano una elevata specificità per aree
differenti: GDF-7 (Growth Differentiation Factor 7), che viene espresso preferenzialmente dalla corteccia M1; occ1, abbondantemente espresso nella corteccia V1; RBP sierica
(Retinol Binding Protein 4) invece è assai presente nelle aree associative e scarsamente presente nella corteccia V1. L’alta precisione di questa tecnica è dimostrata dal fatto che
l’mRNA del GDF-7 costituisce solo lo 0.001% dell’mRNA totale dell’area M1. Questi dati dimostrano che, sebbene ci sia un’uniformità di base nell’espressione genica in regioni
distinte della corteccia cerebrale, alcuni geni mostrano dei profili di espressione altamente area-specifici: per esempio l’esame di RT-PCR quantitativa ha mostrato che il livello di
espressione di GDF-7 era ben 10 volte maggiore in M1 che in V1, e anche gli esami su occ1 e RBP hanno dato risultati simili. Dopo aver condotto questi esami, per comprendere il
significato di una differenza così importante, è fondamentale determinare la distribuzione dei prodotti di
ogni gene a livello cellulare e, nonostante GDF-7 non abbia presentato risultati attendibili negli
esperimenti di ibridazione in situ3, occ1 e RBP hanno mostrato modelli di distribuzione di mRNA molto
interessanti.
Nella corteccia V1, l’mRNA di occ1 è espresso in livelli particolarmente alti nella lamina 4C , dove
giungono gli impulsi delle vie geniculo-corticali, e anche nelle lamine 2, 3, 4A e 4C ; i suoi livelli di
espressione calano notevolmente al confine tra le aree V1 e V2 e in V2 è scarsamente localizzato solo
nella lamina 3. La caratteristica più interessante dell’espressione di occ1 è la sua dipendenza dall’attività
della retina: quando la retina di un occhio è danneggiata con tetradotossina, compromettendone quindi
l’attività, i livelli di mRNA di occ1 subisce cali assai significativi nei domini corticali corrispondenti
all’occhio “avvelenato”. Tali livelli, inoltre, risultano relativamente alta non solo nelle cellule piramidali
della 3 di V2, in cui terminano gli impulsi provenienti dal talamo, ma anche nelle lamine delle cortecce
uditiva e somatosensitiva in cui gli impulsi delle altre vie talamo-corticali giungono. Questo suggerisce
non solo che l’espressione di occ1 abbia a che fare con l’attività dei segnali provenienti dal talamo, ma
anche che ci sia qualcosa di unico nei processi che contraddistinguono le vie ottiche. I profili di
espressione di RBP mostrano, invece, che i suoi livelli non solo sono alti nelle aree associative, ma anche
Fig. 2. Organizzazione della corteccia visiva
nelle lamine di varie altre regioni corticali in cui si svolgono processi di integrazione delle informazioni,
come nelle aree più periferiche della corteccia visiva. L’esatto ruolo di RBP in questi processi può essere
messo in chiaro anche analizzando il suo ruolo associato al retinolo (vitamina A), specialmente se si considerano le recenti evidenze delle connessioni tra l’alterazione del
metabolismo di questa molecola e lo sviluppo di patologie come il morbo di Alzheimer, la schizofrenia e la demenza fronto-temporale.
Tutte queste evidenze dimostrano complessivamente che in futuro lo studio della citoarchitettonica della corteccia cerebrale e lo sviluppo di un modello più completo e
dettagliato dell’espressione genica delle popolazioni cellulari della corteccia i ci come essa può variare di regione in regione possano non solo aiutare a comprendere i meccanismi
che governano la neurofisiologia, ma anche fornire gli elementi di base per lo sviluppo di terapie di medicina molecolare altamente specifiche per il trattamento di molte malattie
neurodegenerative o addirittura contrastandone l’insorgenza.
1
Unica eccezione è rappresentata dall’ippocampo, pentalaminare
La PCR quantitativa è un metodo di amplificazione e quantificazione simultanee del DNA. Spesso la PCR quantitativa è combinata con la PCR Retro Trascrizionale (RT-PCR) per quantificare i livelli di
espressione di specifici RNA: la retro-trascrizione (o trascrizione inversa) produce del DNA complementare a singolo filamento detto cDNA (complementary DNA) mantenendo inalterati i rapporti
relativi di concentrazione delle diverse specie degli RNA. In questo modo è possibile, ad esempio, misurare l'espressione relativa di un gene ad un tempo particolare, o in una cellula o in un tipo
particolare di tessuto. La combinazione di queste due tecniche è spesso denominata RT-PCR quantitativa.
2
3
L’ibridazione in situ è una tecnica con cui, attraverso DNA e RNA marcati, si è in grado di localizzare sequenze specifiche di DNA o RNA nelle cellule di un tessuto; questa tecnica è
spesso utilizzata anche per individuare la posizione o rilevare la presenza di una sequenza di DNA specifica all’interno di un cromosoma.
A cura di Federico Femia
Liberamente rielaborato da:
Gene expression profiling of primate neocortex: molecular neuroanatomy of cortical areas - A. Watakabe, Y. Komatsu, H. Nawa, T. Yamamori - National
Institute for Basic Biology, Okazaki, and Department of Molecular Neurobiology, Brain Research Institute, Niigata University, Niigata, Japan
Altre references:
Campbell, M.J., Lewis, D.A., Benoit, R. & Morrison, J.H. (1987) - Regional heterogeneity in the distribution of somatostatin-28 and somatostatin-28(1–12)
immunoreactive profiles in monkey neocortex. J Neurosci 7, 1133–1144.
Tochitani S., Liang F., Watakabe A., Hashikawa T. & Yamamori T. (2001) - The occ1 gene is preferentially expressed in the primary visual cortex in an activitydependent manner: a pattern of gene expression related to the cytoarchitectonic area in adult macaque neocortex. Eur J Neurosci 13, 297–307.
Komatsu, Y., Watakabe, A., Hashikawa, T., Tochitani, S. & Yamamori, T. (2005) - Retinol-binding protein gene is highly expressed in higher-order
association areas of the primate neocortex. Cereb Cortex 15, 96–108.
Per ulteriori informazioni su RT-PCR quantitativa, Differential Display PCR, DNA-microarray e processi di ibridazione in situ:
http://it.wikipedia.org/wiki/Real_time_PCR
http://en.wikipedia.org/wiki/Differential_display
http://it.wikipedia.org/wiki/DNA_microarray
http://en.wikipedia.org/wiki/In_situ_hybridization
A cura di Federico Femia